Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , Mile Šikić 2
1 Faculdade de Engenharia Elétrica e Computação, Universidade de Zagreb, Croácia
2 Instituto Genome de Cingapura (GIS), Agência de Ciência, Tecnologia e Pesquisa (A*Star), Cingapura
3 Instituto de Bioinformática (BII), Agência de Ciência, Tecnologia e Pesquisa (A*Star), Cingapura
Esta é a implementação oficial do artigo "Rinalmo: modelos de idiomas de RNA de uso geral podem generalizar bem as tarefas de previsão de estrutura".
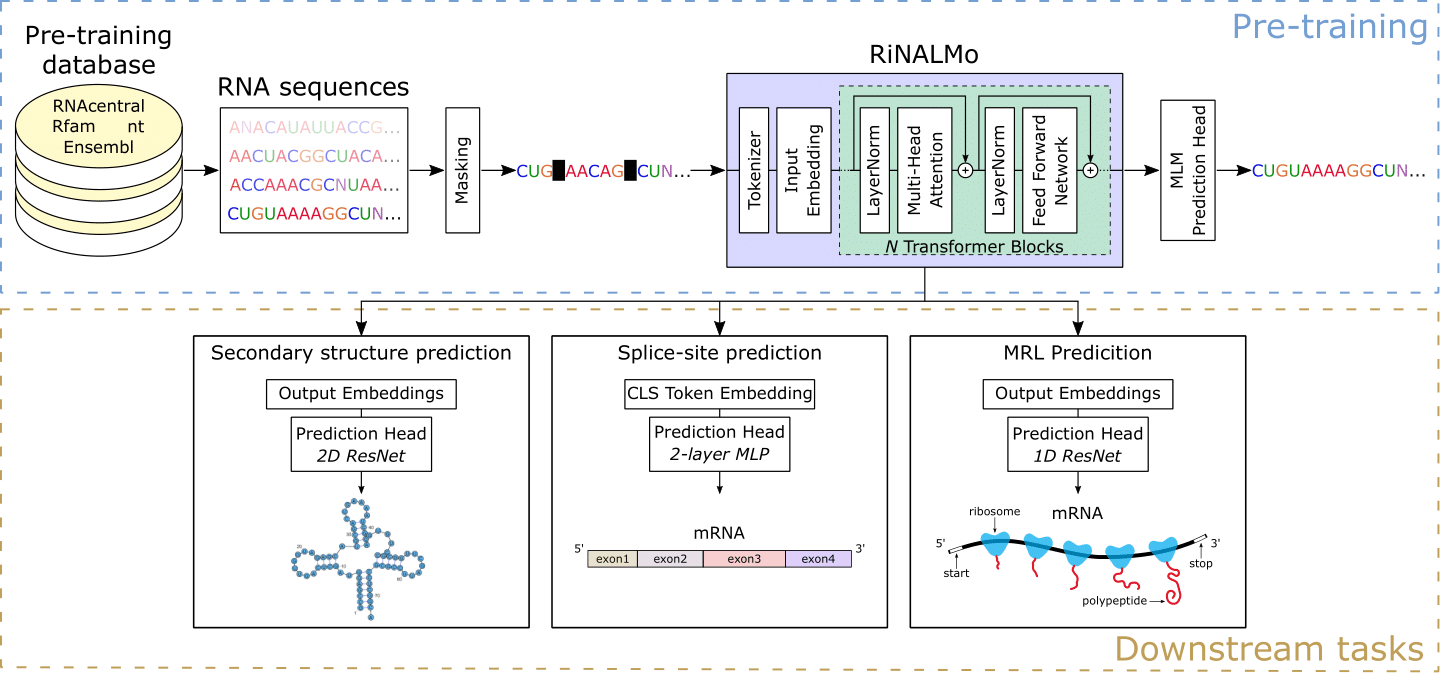
O ácido ribonucleico (RNA) desempenha uma variedade de papéis cruciais nos processos biológicos fundamentais. Recentemente, o RNA se tornou um alvo interessante de drogas, enfatizando a necessidade de melhorar nossa compreensão de suas estruturas e funções. Ao longo dos anos, as tecnologias de seqüenciamento produziram uma quantidade enorme de dados de RNA não marcados, o que oculta o conhecimento e o potencial importantes. Motivada pelos sucessos dos modelos de linguagem de proteínas, introduzimos o Modelo de Linguagem de Ácido Ribonucleico (RINALMO) para ajudar a revelar o código oculto de RNA. O Rinalmo é o maior modelo de idioma de RNA até o momento, com 650 milhões de parâmetros pré-treinados em 36 milhões de sequências de RNA não codificantes de vários bancos de dados disponíveis. O Rinalmo é capaz de extrair conhecimento oculto e capturar as informações da estrutura subjacente incorporadas implicitamente nas sequências de RNA. O Rinalmo alcança resultados de última geração em várias tarefas a jusante. Notavelmente, mostramos que suas capacidades de generalização podem superar a incapacidade de outros métodos profundos de aprendizado de previsão de estrutura secundária para generalizar em famílias de RNA invisíveis.
Use os seguintes comandos para a instalação (Pré -requisitos: Python>=3.8 e CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2Após a instalação, você pode usar facilmente o Rinalmo para obter representações de nucleotídeos:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml . # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..Você também pode baixar pesos pré-treinados para modelos Rinalmo menores (Rinalmo-148m (Mega) e Rinalmo-33,5m (micro)) em https://drive.google.com/drive/Folders/1VGK3GY8C010WQFPMYX5VOY4WY8E8EBQS.
Fornecemos pesos de rinalmo pré-treinados e pesos de ajuste fino para três tarefas a jusante: previsão média de carga do ribossomo, previsão de estrutura secundária e previsão do local de emenda. Para avaliação e ajuste fino, use train_<downstream_task>.py scripts.
Para avaliar os modelos Rinalmo e cabeças de previsão de ajustes finos fornecidos, execute os scripts usando os seguintes argumentos de entrada:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir Para avaliar o modelo Rinalmo e a cabeça de previsão de ajuste fino no conjunto de dados de teste de rRNA do Archiveii 5S para previsão da estrutura secundária, use os pesos rinalmo_giga_ss_archiveII-5s_ft.pt . Aqui, fornecemos um exemplo de comando de execução.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
Para ajustar o rinalmo, use --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt Argumento de entrada. Use --help para aprender sobre outros argumentos disponíveis. Para a tarefa de previsão do local de emenda, o código de pré-processamento de dados e dados está disponível em https://git.unistra.fr/nsscalzitti/spliceator.git.
Copyright 2024 Šikić Lab - AI em genômica
Licenciado sob a licença Apache, versão 2.0 (a "licença"); Você não pode usar esse arquivo, exceto em conformidade com a licença. Você pode obter uma cópia da licença em
http://www.apache.org/license/license-2.0
A menos que exigido pela lei aplicável ou acordada por escrito, o software distribuído sob a licença é distribuído "como está", sem garantias ou condições de qualquer tipo, expressa ou implícita. Consulte a licença para o idioma específico que rege as permissões e limitações sob a licença.
Os parâmetros Rinalmo são disponibilizados nos termos da licença Creative Commons Attribution 4.0 International (CC por 4.0). Você pode encontrar detalhes em: https://creativecommons.org/license/by/4.0/legalcode.
Se você achar nosso trabalho útil em sua pesquisa, cite:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}Se você tiver alguma dúvida, não hesite em enviar um email aos autores ou abrir um problema.
This work was supported in part by the National Research Foundation (NRF) Competitive Research Programme (CRP) under Project Identifying Functional RNA Tertiary Structures in Dengue Virus (NRF-CRP27-2021RS-0001) and in part by the A*STAR under Grant GAP2: A*STAR RNA-Foundation Model (A*STAR RNA-FM) (I23D1AG079).
O trabalho computacional para o artigo foi parcialmente realizado em recursos do Centro Nacional de Supercomputação, Cingapura https://www.nscc.sg.