Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , mil Šikić 2
1 Fakultas Teknik Listrik dan Komputasi, Universitas Zagreb, Kroasia
2 Genome Institute of Singapore (GIS), Agency for Science, Technology and Research (A*Star), Singapura
3 Bioinformatics Institute (BII), Agency for Science, Technology and Research (A*Star), Singapura
Ini adalah implementasi resmi dari makalah "RinalMo: Model Bahasa RNA tujuan umum dapat menggeneralisasi dengan baik pada tugas prediksi struktur".
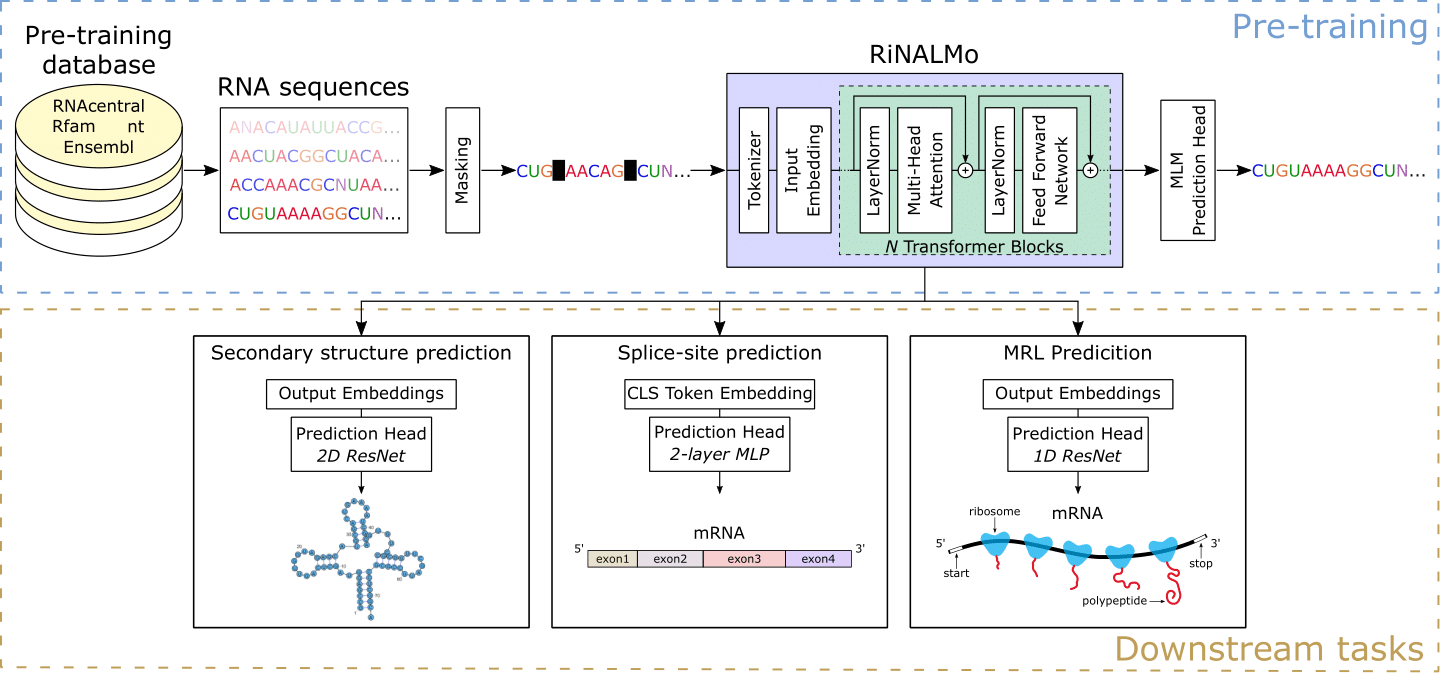
Asam ribonukleat (RNA) memainkan berbagai peran penting dalam proses biologis mendasar. Baru -baru ini, RNA telah menjadi target obat yang menarik, menekankan perlunya meningkatkan pemahaman kita tentang struktur dan fungsinya. Selama bertahun -tahun, teknologi sekuensing telah menghasilkan sejumlah besar data RNA yang tidak berlabel, yang menyembunyikan pengetahuan dan potensi penting. Termotivasi oleh keberhasilan model bahasa protein, kami memperkenalkan model bahasa asam ribonukleat (rinalMO) untuk membantu mengungkap kode tersembunyi RNA. RinalMO adalah model bahasa RNA terbesar hingga saat ini dengan 650 juta parameter yang telah dilatih sebelumnya pada 36 juta sekuens RNA non-coding dari beberapa database yang tersedia. Rinalmo mampu mengekstraksi pengetahuan tersembunyi dan menangkap informasi struktur yang mendasarinya secara implisit tertanam dalam urutan RNA. Rinalmo mencapai hasil canggih pada beberapa tugas hilir. Khususnya, kami menunjukkan bahwa kemampuan generalisasi dapat mengatasi ketidakmampuan metode pembelajaran mendalam lainnya untuk prediksi struktur sekunder untuk menggeneralisasi keluarga RNA yang tidak terlihat.
Gunakan perintah berikut untuk instalasi (prasyarat: Python>=3.8 dan CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2Setelah pemasangan Anda dapat dengan mudah menggunakan rinalmo untuk mendapatkan representasi nukleotida:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml . # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..Anda juga dapat mengunduh bobot pra-terlatih untuk model rinalmo yang lebih kecil (rinalMO-148M (mega) dan rinalmo-33,5m (micro)) di https://drive.google.com/drive/folders/1vgk3gy8c01o0wqfpmyx5voy4wy8eBqs5.
Kami memberikan bobot rinalmo pra-terlatih dan bobot yang disesuaikan untuk tiga tugas hilir: prediksi pemuatan ribosom rata-rata, prediksi struktur sekunder dan prediksi lokasi sambatan. Untuk evaluasi dan penyempurnaan penggunaan train_<downstream_task>.py skrip.
Untuk mengevaluasi model rinalmo yang disediakan dan kepala prediksi, silakan jalankan skrip menggunakan argumen input berikut:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir Untuk mengevaluasi model rinalmo yang disesuaikan dan prediksi kepala pada dataset uji rRNA Archiveii 5s untuk prediksi struktur sekunder, gunakan bobot rinalmo_giga_ss_archiveII-5s_ft.pt . Di sini, kami memberikan Contoh Perintah Jalankan.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
Untuk menyempurnakan rinalMO, gunakan --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt argumen input. Gunakan --help untuk mempelajari argumen lain yang tersedia. Untuk tugas prediksi situs splice, dataset dan kode preprocessing data tersedia di https://git.unistra.fr/nscalzitti/spliceator.git.
Hak Cipta 2024 Šikić Lab - AI dalam Genomik
Berlisensi di bawah lisensi Apache, versi 2.0 ("lisensi"); Anda tidak boleh menggunakan file ini kecuali sesuai dengan lisensi. Anda dapat memperoleh salinan lisensi di
http://www.apache.org/licenses/license-2.0
Kecuali diharuskan oleh hukum yang berlaku atau disepakati secara tertulis, perangkat lunak yang didistribusikan di bawah lisensi didistribusikan berdasarkan "sebagaimana adanya", tanpa jaminan atau ketentuan dalam bentuk apa pun, baik tersurat maupun tersirat. Lihat lisensi untuk bahasa spesifik yang mengatur izin dan batasan di bawah lisensi.
Parameter RinalMO tersedia di bawah ketentuan Lisensi Creative Commons Attribution 4.0 International (CC dengan 4.0). Anda dapat menemukan detail di: https://creativecommons.org/licenses/by/4.0/legalcode.
Jika Anda menemukan pekerjaan kami berguna dalam penelitian Anda, silakan kutip:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}Jika Anda memiliki pertanyaan, jangan ragu untuk mengirim email kepada penulis atau membuka masalah.
Pekerjaan ini didukung sebagian oleh Program Penelitian Kompetitif National Research Foundation (NRF) (CRP) di bawah proyek mengidentifikasi struktur tersier RNA fungsional dalam virus demam berdarah (NRF-CRP27-2021RS-0001) dan sebagian oleh A*Star di bawah hibah GAP2: A*Star RNA-Foundation Model (A*Star RNA-FM) (A*RNA-FM ).
Pekerjaan komputasi untuk makalah ini sebagian dilakukan pada sumber daya Pusat Superkomputer Nasional, Singapura https://www.nscc.sg.