Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , Mile Šikić 2
1 Faculté de génie électrique et informatique, Université de Zagreb, Croatie
2 Genome Institute of Singapour (SIG), Agence pour la science, la technologie et la recherche (A * Star), Singapour
3 Bioinformatics Institute (BII), Agency for Science, Technology and Research (A * Star), Singapour
Il s'agit de la mise en œuvre officielle de l'article "RinalMO: les modèles de langage d'ARN à usage général peuvent bien généraliser sur les tâches de prédiction de structure".
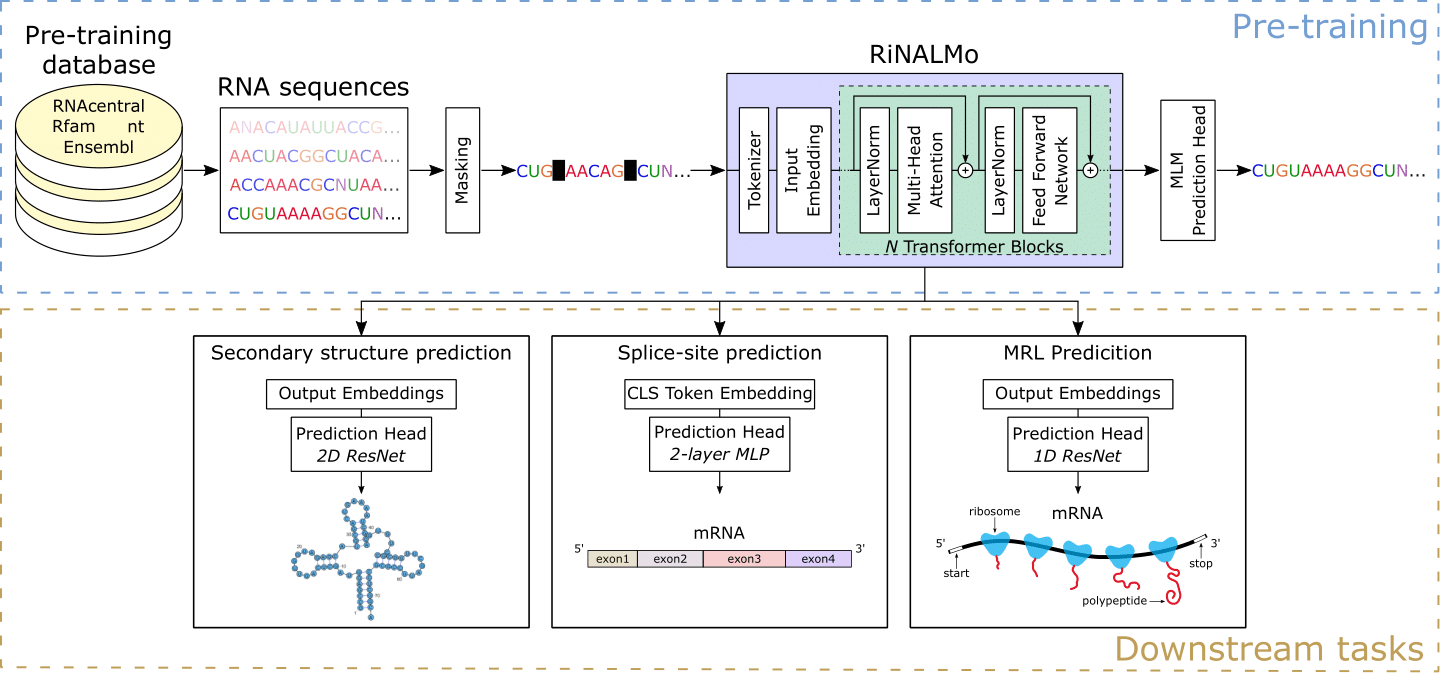
L'acide ribonucléique (ARN) joue une variété de rôles cruciaux dans les processus biologiques fondamentaux. Récemment, l'ARN est devenu une cible de médicament intéressante, soulignant la nécessité d'améliorer notre compréhension de ses structures et de ses fonctions. Au fil des ans, les technologies de séquençage ont produit une énorme quantité de données d'ARN non marquées, qui cachent des connaissances et un potentiel important. Motivés par les succès des modèles de langage protéique, nous introduisons le modèle de langage d'acide ribonucléique (RinalMO) pour aider à dévoiler le code caché d'ARN. RinalMO est le plus grand modèle de langage d'ARN à ce jour avec 650 millions de paramètres pré-formés sur 36 millions de séquences d'ARN non codantes à partir de plusieurs bases de données disponibles. Rinalmo est capable d'extraire les connaissances cachées et de capturer les informations sous-jacentes de la structure implicitement intégrées dans les séquences d'ARN. Rinalmo obtient des résultats de pointe sur plusieurs tâches en aval. Nous montrons notamment que ses capacités de généralisation peuvent surmonter l'incapacité d'autres méthodes d'apprentissage en profondeur pour la prédiction de la structure secondaire afin de généraliser sur les familles d'ARN invisibles.
Utilisez les commandes suivantes pour l'installation (Prérequis: Python>=3.8 et CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2Après l'installation, vous pouvez facilement utiliser RinalMO pour obtenir des représentations nucléotidiques:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml . # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..Vous pouvez également télécharger des poids pré-formés pour les modèles Rinalmo plus petits (RinalMo-148m (Mega) et RinalMo-33,5m (Micro)) à https://drive.google.com/drive/folders/1vgk3gy8c01o0wqfpmyx5voy4wy8ebqs5.
Nous fournissons des poids Rinalmo pré-formés et des poids affinés pour trois tâches en aval: prédiction moyenne de chargement des ribosomes, prédiction de la structure secondaire et prédiction du site d'épissure. Pour l'évaluation et le réglage fin, utilisez des scripts train_<downstream_task>.py .
Afin d'évaluer les modèles Rinalmo et les têtes de prédiction réglés à réglage fin fourni, veuillez exécuter les scripts en utilisant les arguments d'entrée suivants:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir Pour évaluer le modèle RinalMO et la tête de prédiction affinés sur l'ensemble de données de test RRNA Archiveii 5S pour la prédiction de la structure secondaire, utilisez les poids rinalmo_giga_ss_archiveII-5s_ft.pt . Ici, nous fournissons un exemple de commande d'exécution.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
Afin d'adapter RinalMo, utilisez --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt argument d'entrée. Utiliser --help pour en savoir plus sur les autres arguments disponibles. Pour la tâche de prédiction de sites Splice, l'ensemble de données et le code de prétraitement des données sont disponibles sur https://git.unistra.fr/nscalzitti/spliceator.git.
Copyright 2024 Šikić Lab - AI en génomique
Licencié sous la licence Apache, version 2.0 (la "licence"); Vous ne pouvez pas utiliser ce fichier sauf conforme à la licence. Vous pouvez obtenir une copie de la licence à
http://www.apache.org/licenses/license-2.0
Sauf exiger la loi applicable ou convenu par écrit, les logiciels distribués en vertu de la licence sont distribués sur une base «tel quel», sans garantie ou conditions d'aucune sorte, expresse ou implicite. Voir la licence pour la langue spécifique régissant les autorisations et les limitations sous la licence.
Les paramètres Rinalmo sont mis à disposition en vertu des termes de la licence Creative Commons Attribution 4.0 International (CC by 4.0). Vous pouvez trouver des détails sur: https://creativecommons.org/licenses/by/4.0/legalcode.
Si vous trouvez notre travail utile dans vos recherches, veuillez citer:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}Si vous avez des questions, n'hésitez pas à envoyer un e-mail aux auteurs ou à ouvrir un problème.
Ce travail a été soutenu en partie par le Programme de recherche concurrentiel (CRP) de la National Research Foundation (NRF) dans le cadre du projet d'identification des structures tertiaires de l'ARN fonctionnelle dans le virus de la dengue (NRF-CRP27-2021RS-0001) et en partie par le modèle A * Star sous Grant 2: A * Star ARN-Foundation) .
Le travail de calcul pour le journal a été partiellement effectué sur les ressources du National Supercomputing Center, Singapour https://www.nscc.sg.