Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , Mile Šikić 2
1 Fakultät für Elektrotechnik und Computing, Universität Zagreb, Kroatien
2 Genome Institute of Singapore (GIS), Agentur für Wissenschaft, Technologie und Forschung (ein*Star), Singapur
3 Bioinformatics Institute (BII), Agentur für Wissenschaft, Technologie und Forschung (A*Star), Singapur
Dies ist die offizielle Implementierung des Papiers "Rinalmo: Allzweck-RNA-Sprachmodelle können gut auf Strukturvorhersageaufgaben verallgemeinert".
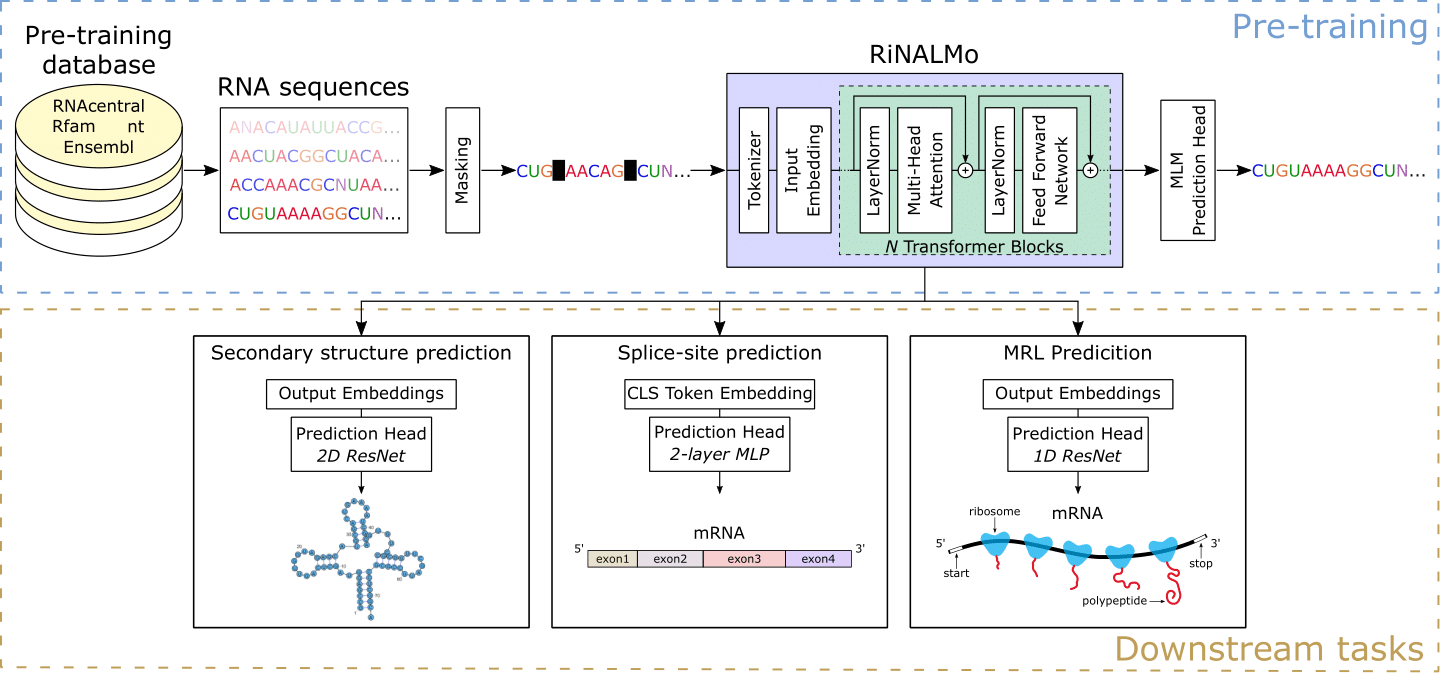
Ribonukleinsäure (RNA) spielt eine Vielzahl von entscheidenden Rollen bei grundlegenden biologischen Prozessen. In jüngster Zeit ist RNA zu einem interessanten Arzneimittelziel geworden und betont die Notwendigkeit, unser Verständnis seiner Strukturen und Funktionen zu verbessern. Im Laufe der Jahre haben Sequenzierungstechnologien eine enorme Menge an nicht markierten RNA -Daten erzeugt, die wichtiges Wissen und Potenzial verborgen. Motiviert durch die Erfolge von Proteinsprachmodellen führen wir Ribonukleinsäure -Sprachmodell (RINALMO) ein, um den versteckten Code der RNA zu enthüllen. Rinalmo ist das bisher größte RNA-Sprachmodell mit 650 Millionen Parametern, die auf 36 Millionen nicht-kodierenden RNA-Sequenzen aus mehreren verfügbaren Datenbanken ausgebildet sind. Rinalmo ist in der Lage, verborgenes Wissen zu extrahieren und die zugrunde liegenden Strukturinformationen implizit in die RNA -Sequenzen eingebettet zu erfassen. Rinalmo erzielt hochmoderne Ergebnisse bei mehreren nachgeschalteten Aufgaben. Insbesondere zeigen wir, dass seine Verallgemeinerungsfähigkeiten die Unfähigkeit anderer Deep -Lern -Methoden zur Vorhersage der sekundären Struktur zur Verallgemeinerung unsichtbarer RNA -Familien überwinden können.
Verwenden Sie die folgenden Befehle für die Installation (Voraussetzungen: Python>=3.8 und CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2Nach der Installation können Sie Rinalmo leicht verwenden, um Nucleotid -Darstellungen zu erhalten:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml enthalten sein. # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..You can also download pre-trained weights for smaller RiNALMo models (RiNALMo-148M (mega) and RiNALMo-33,5M (micro)) at https://drive.google.com/drive/folders/1vGk3gY8c01o0wQfpmyX5vOY4Wy8EbQS5.
Wir liefern vorgebildete Rinalmo-Gewichte und fein abgestimmte Gewichte für drei nachgeschaltete Aufgaben: die mittlere Ribosomenladungsvorhersage, die Vorhersage der Sekundärstruktur und die Vorhersage von Splice-Site. Verwenden Sie sowohl für die Bewertung als auch für die Feinabstimmung train_<downstream_task>.py -Skripte.
Um die bereitgestellten fein abgestimmten Rinalmo-Modelle und Vorhersageköpfe zu bewerten, führen Sie die Skripte bitte mit den folgenden Eingabemarions aus:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir Um das fein abgestimmte RINALMO-Modell und den Vorhersagekopf auf ArchiveII 5S-rRNA-Testdatensatz für die Vorhersage der Sekundärstruktur zu bewerten, verwenden Sie die rinalmo_giga_ss_archiveII-5s_ft.pt 5S_FT.PT-Gewichte. Hier geben wir einen Beispiel für ein Beispiel für den Lauf.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
Um rinalmo zu fein abzustimmen, verwenden Sie --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt Eingabeargument. Verwenden Sie --help , um andere verfügbare Argumente zu erfahren. Für die Task-Site-Vorhersageaufgabe sind der Datensatz- und Datenvorverarbeitungscode unter https://git.unistra.fr/nscalzitti/spliceator.git verfügbar.
Copyright 2024 Šikić Lab - AI in der Genomik
Lizenziert unter der Apache -Lizenz, Version 2.0 (der "Lizenz"); Sie dürfen diese Datei nur in Übereinstimmung mit der Lizenz verwenden. Sie können eine Kopie der Lizenz bei erhalten
http://www.apache.org/licenses/license-2.0
Sofern nicht nach geltendem Recht oder schriftlich zu vereinbart wird, wird die im Rahmen der Lizenz verteilte Software auf "As ist" Basis ohne Gewährleistung oder Bedingungen jeglicher Art, entweder ausdrücklich oder impliziert, verteilt. Siehe die Lizenz für die spezifischen Sprachberechtigungen und Einschränkungen im Rahmen der Lizenz.
Die Rinalmo -Parameter werden gemäß den Bestimmungen der Creative Commons Attribution 4.0 International (CC by 4.0) zur Verfügung gestellt. Sie finden Details unter: https://creatvecommons.org/licenses/by/4.0/legalcode.
Wenn Sie unsere Arbeit in Ihrer Forschung nützlich finden, zitieren Sie bitte:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}Wenn Sie Fragen haben, können Sie die Autoren per E -Mail senden oder ein Problem öffnen.
Diese Arbeit wurde teilweise vom Wettbewerbsprogramm der National Research Foundation (NRF) (CRP) im Rahmen von Projekten zur Identifizierung funktionaler RNA-Tertiärstrukturen in Dengue-Virus (NRF-CRP27-2021RS-0001) und teilweise durch das A*star unter Grant Gap2: A*Stern RNA-Fundierungsmodell (a*star rna-fm) (i23d1ag079) unterstützt.
Die Computerarbeit für das Papier wurde teilweise auf Ressourcen des National Supercomputing Center, Singapur https://www.nscc.sg, durchgeführt.