Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , Mile Šikić 2
1 크로아티아 자그레브 대학교 전기 공학 및 컴퓨팅 학부
2 싱가포르 GIS (Genome Institute of Singapore), 과학, 기술 및 연구 기관 (A*Star), 싱가포르
3 Bioinformatics Institute (BII), 과학, 기술 및 연구 기관 (A*Star), 싱가포르
이것은 논문 "Rinalmo : General-Purpose RNA 언어 모델이 구조 예측 작업에서 일반화 할 수있는 논문의 공식 구현입니다.
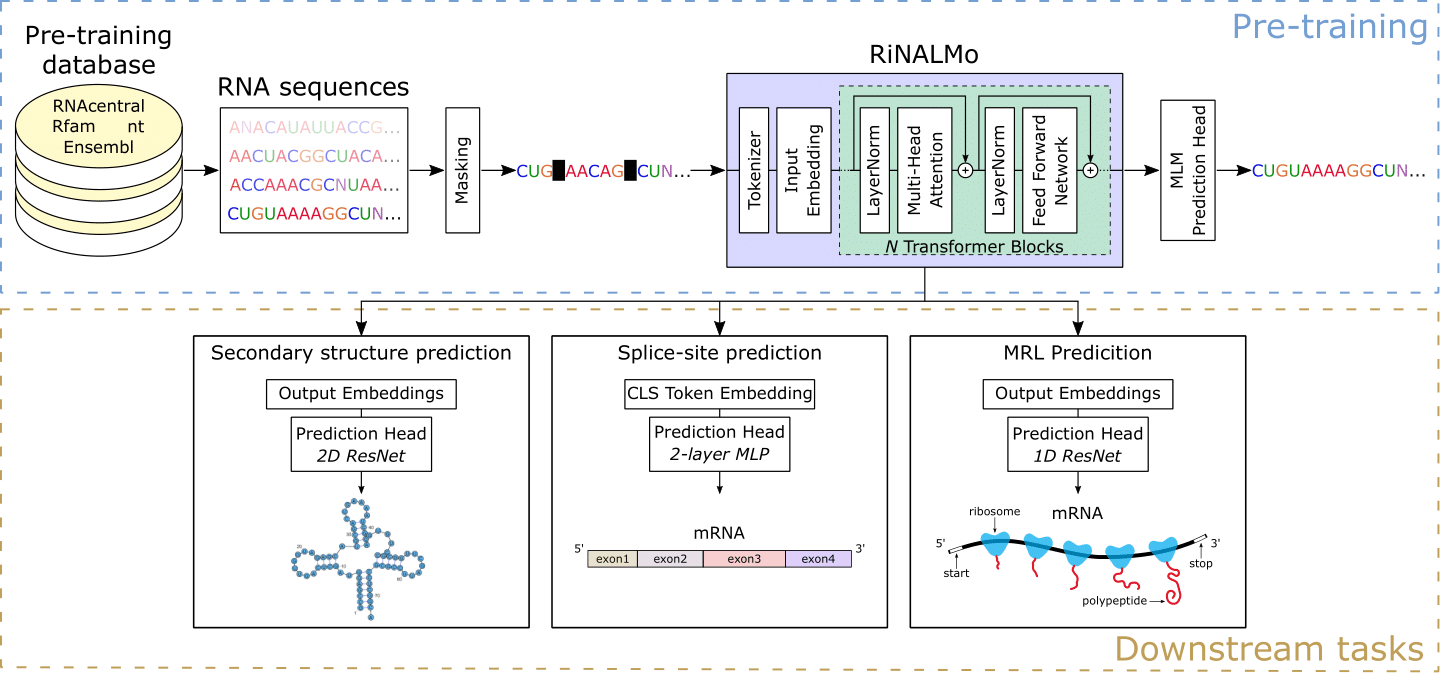
리보 핵산 (RNA)은 기본 생물학적 과정에서 다양한 중요한 역할을합니다. 최근 RNA는 흥미로운 약물 표적이되어 구조와 기능에 대한 이해를 향상시켜야 할 필요성을 강조했습니다. 수년에 걸쳐, 시퀀싱 기술은 막대한 양의 표지되지 않은 RNA 데이터를 생산하여 중요한 지식과 잠재력을 숨기고 있습니다. 단백질 언어 모델의 성공에 동기를 부여한 우리는 RNA의 숨겨진 코드를 공개하기 위해 리보 핵산 언어 모델 (Rinalmo)을 소개합니다. Rinalmo는 현재 이용 가능한 여러 데이터베이스에서 3,600 만 비 코딩 RNA 서열에 미리 훈련 된 6 억 6 천만 개의 매개 변수를 가진 최대의 RNA 언어 모델입니다. Rinalmo는 숨겨진 지식을 추출하고 RNA 서열 내에 암시 적으로 내장 된 기본 구조 정보를 포착 할 수 있습니다. Rinalmo는 여러 다운 스트림 작업에서 최첨단 결과를 달성합니다. 특히, 우리는 일반화 능력이 보이지 않는 RNA 가족을 일반화하기 위해 2 차 구조 예측을위한 다른 딥 러닝 방법의 무능력을 극복 할 수 있음을 보여줍니다.
설치에 다음 명령을 사용하십시오 (전제 조건 : Python>=3.8 및 CUDA>=11.8 ) :
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2설치 후 Rinalmo를 사용하여 뉴클레오티드 표현을 쉽게 얻을 수 있습니다.
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml 에 포함되어야합니다. # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..https://drive.google.com/drive/dolders/1vgk3gy8c01o0wqfppyx5voy4wy8ebqs5에서 소규모 Rinalmo 모델 (Rinalmo-148m (Mega) 및 Rinalmo-33,5m (Micro))에 미리 훈련 된 가중치를 다운로드 할 수도 있습니다.
우리는 세 가지 다운 스트림 작업에 대해 미리 훈련 된 RINALMO 가중치 및 미세 조정 가중치를 제공합니다. 평균 리보솜 하중 예측, 2 차 구조 예측 및 스플 라이스 사이트 예측. 평가 및 미세 조정 사용 train_<downstream_task>.py 스크립트.
제공된 미세 조정 된 rinalmo 모델 및 예측 헤드를 평가하려면 다음 입력 인수를 사용하여 스크립트를 실행하십시오.
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir 보조 구조 예측을위한 Archiveii 5S RRNA 테스트 데이터 세트에서 미세 조정 된 RINALMO 모델 및 예측 헤드를 평가하려면 rinalmo_giga_ss_archiveII-5s_ft.pt 가중치를 사용하십시오. 여기서는 예제 런 명령을 제공합니다.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
rinalmo를 미세 조정하려면 --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt 입력 인수를 사용하십시오. 사용 가능한 다른 주장에 대해 배우기 위해 --help 사용하십시오. 스플 라이스 사이트 예측 작업의 경우 데이터 세트 및 데이터 전처리 코드는 https://git.unistra.fr/nscalzitti/spliceator.git에서 사용할 수 있습니다.
저작권 2024 Šikić Lab -AI Genomics
Apache 라이센스, 버전 2.0 ( "라이센스")에 따라 라이센스가 부여되었습니다. 라이센스를 준수하는 것 외에는이 파일을 사용할 수 없습니다. 라이센스 사본을 얻을 수 있습니다
http://www.apache.org/licenses/license-2.0
해당 법률에 의해 요구되거나 서면에 동의하지 않는 한, 라이센스에 따라 배포 된 소프트웨어는 명시 적 또는 묵시적 보증 또는 조건없이 "그대로"기준으로 배포됩니다. 라이센스에 따른 특정 언어 통치 권한 및 제한 사항에 대한 라이센스를 참조하십시오.
Rinalmo 매개 변수는 Creative Commons Attribution 4.0 International (CC By 4.0) 라이센스의 용어에 따라 제공됩니다. https://creativecommons.org/licenses/by/4.0/legalcode에서 세부 정보를 찾을 수 있습니다.
귀하의 연구에 우리의 작업이 유용하다는 것을 알게되면 다음을 인용하십시오.
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}궁금한 점이 있으시면 저자에게 이메일을 보내거나 문제를 열어주십시오.
이 연구는 뎅기 바이러스 (NRF-CRP27-2021RS-0001)에서 기능적 RNA 3 차 구조를 식별하는 프로젝트 하에서 NRF (National Research Foundation) 경쟁 연구 프로그램 (NRF) 경쟁 연구 프로그램 (CRP)에 의해 부분적으로 지원되었다.
이 논문의 계산 작업은 싱가포르의 National Supercomputing Center의 자원 https://www.nscc.sg의 자원에 대해 부분적으로 수행되었습니다.