Rafael Josip Penić 1 , Tin Vlašić 2 , Roland G. Huber 3 , Yue Wan 2 , milla Šikić 2
1 Facultad de Ingeniería y Computación Eléctrica, Universidad de Zagreb, Croacia
2 Genome Institute of Singapur (SIG), Agencia de Ciencia, Tecnología e Investigación (A*Star), Singapur
3 Bioinformatics Institute (BII), Agencia de Ciencia, Tecnología e Investigación (A*Star), Singapur
Esta es la implementación oficial del documento "Rinalmo: los modelos de lenguaje de ARN de uso general pueden generalizarse bien en las tareas de predicción de la estructura".
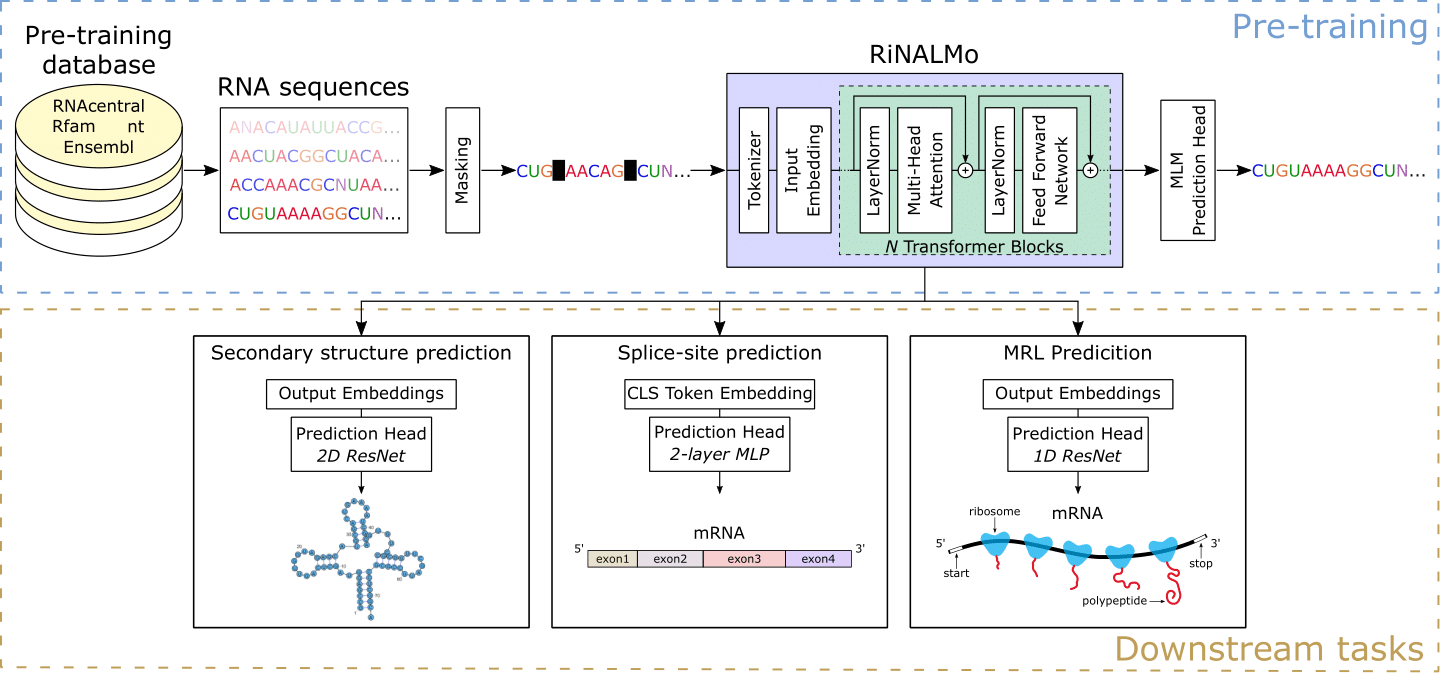
El ácido ribonucleico (ARN) juega una variedad de roles cruciales en procesos biológicos fundamentales. Recientemente, el ARN se ha convertido en un objetivo fármaco interesante, enfatizando la necesidad de mejorar nuestra comprensión de sus estructuras y funciones. A lo largo de los años, las tecnologías de secuenciación han producido una enorme cantidad de datos de ARN no etiquetados, que oculta un conocimiento y potencial importantes. Motivado por los éxitos de los modelos de lenguaje de proteínas, presentamos el modelo de lenguaje de ácido ribonucleico (RinalMO) para ayudar a revelar el código oculto de ARN. Rinalmo es el modelo de lenguaje de ARN más grande hasta la fecha con 650 millones de parámetros previamente capacitados en 36 millones de secuencias de ARN no codificantes de varias bases de datos disponibles. Rinalmo puede extraer conocimiento oculto y capturar la información de estructura subyacente implícitamente integrada dentro de las secuencias de ARN. Rinalmo logra resultados de última generación en varias tareas aguas abajo. En particular, mostramos que sus capacidades de generalización pueden superar la incapacidad de otros métodos de aprendizaje profundo para la predicción de la estructura secundaria para generalizar en familias de ARN invisibles.
Use los siguientes comandos para la instalación (Requisitos previos: Python>=3.8 y CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2Después de la instalación, puede usar fácilmente Rinalmo para obtener representaciones de nucleótidos:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml . # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..También puede descargar pesos previamente capacitados para modelos Rinalmo más pequeños (Rinalmo-148m (MEGA) y Rinalmo-33,5m (Micro)) en https://drive.google.com/drive/folders/1VGK3GY8C01O0WQFPMYX5VOY4WY8EBQS5.
Proporcionamos pesos RinalMO previamente capacitados y pesos ajustados para tres tareas aguas abajo: predicción media de carga de ribosomas, predicción de la estructura secundaria y predicción del sitio de empalme. Para evaluación y ajuste, use train_<downstream_task>.py scripts.
Para evaluar los modelos Rinalmo y los cabezales de predicción proporcionados, ejecutar los scripts utilizando los siguientes argumentos de entrada:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir Para evaluar el modelo RinalMO ajustado y la cabeza de predicción en el conjunto de datos de prueba de ARNr de ArchiveII 5S para la predicción de la estructura secundaria, use los pesos rinalmo_giga_ss_archiveII-5s_ft.pt . Aquí, proporcionamos un comando Ejemplo de ejecución.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
Para ajustar rinalmo, use- --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt argumento de entrada. Use --help para aprender sobre otros argumentos disponibles. Para la tarea de predicción del sitio de empalme, el conjunto de datos de datos y el código de preprocesamiento de datos están disponibles en https://git.unistra.fr/nscalzitti/spliceator.git.
Copyright 2024 Šikić Lab - Ai en genómica
Licenciado bajo la licencia Apache, versión 2.0 (la "licencia"); No puede usar este archivo, excepto de conformidad con la licencia. Puede obtener una copia de la licencia en
http://www.apache.org/licenses/license-2.0
A menos que la ley aplicable sea requerida o acordado por escrito, el software distribuido bajo la licencia se distribuye de manera "como es", sin garantías ni condiciones de ningún tipo, ya sea expresas o implícitas. Consulte la licencia para los permisos y limitaciones de rigor de idioma específico bajo la licencia.
Los parámetros RinalMO están disponibles bajo los términos de la licencia Creative Commons Attribution 4.0 International (CC por 4.0). Puede encontrar detalles en: https://createivecommons.org/licenses/by/4.0/legalcode.
Si encuentra útil nuestro trabajo en su investigación, cite:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}Si tiene alguna pregunta, no dude en enviar un correo electrónico a los autores o abrir un problema.
Este trabajo fue apoyado en parte por el Programa de Investigación Competitiva de la Fundación Nacional de Investigación (NRF) (CRP) bajo el proyecto que identifica las estructuras terciarias de ARN funcional en el virus del dengue (NRF-CRP27-2021RS-0001) y en parte por un*Star Gap2: A*modelo de terminación de ARN*(A*Star RNA-FM) (I23D1AG079).
El trabajo computacional para el documento se realizó parcialmente en recursos del Centro Nacional de Supercomputación, Singapur https://www.nscc.sg.