Рафаэль Джосип Пенич 1 , Тин Влашич 2 , Роланд Г. Хубер 3 , Юэ Ван 2 , Миля Шикич 2
1 факультет электротехники и вычислений, Университет Загреба, Хорватия
2 Институт генома Сингапура (ГИС), Агентство по науке, технологиям и исследованиям (A*Star), Сингапур
3 Институт биоинформатики (BII), Агентство по науке, технологиям и исследованиям (A*Star), Сингапур
Это официальная реализация статьи «Rinalmo: модели языка РНК общего назначения могут хорошо обобщать задачи прогнозирования структуры».
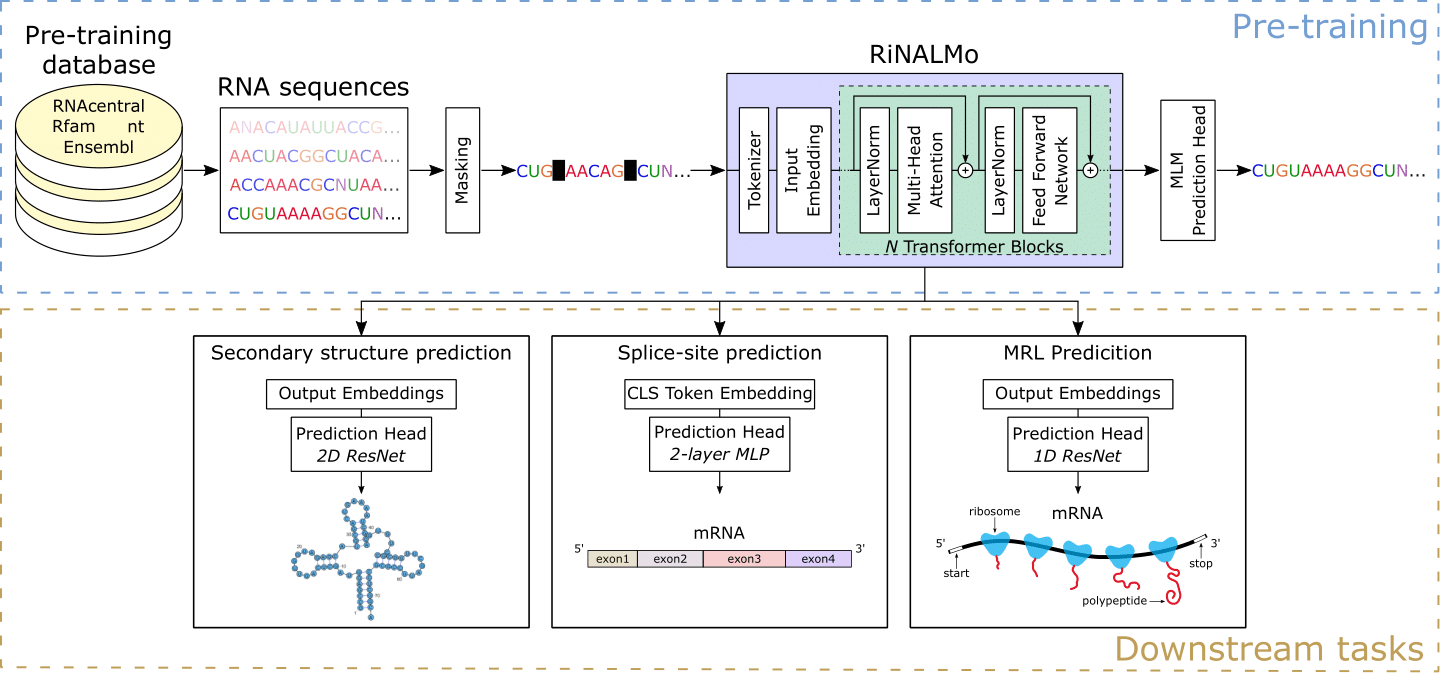
Рибонуклеиновая кислота (РНК) играет множество важнейших ролей в фундаментальных биологических процессах. Недавно РНК стала интересной целью лекарств, подчеркивая необходимость улучшения нашего понимания его структур и функций. На протяжении многих лет технологии секвенирования создали огромное количество немеченых данных РНК, которые скрывают важные знания и потенциал. Мотивированный успехами моделей белкового языка, мы вводим модель языка рибонуклеиновой кислоты (Rinalmo), чтобы помочь представить скрытый код РНК. Rinalmo является крупнейшей моделью языка РНК на сегодняшний день с 650 миллионами параметров, предварительно обученных на 36 миллионов некодирующих последовательностей РНК из нескольких доступных баз данных. Rinalmo способен извлекать скрытые знания и захватывать базовую информацию о структуре, неявно встроенная в последовательности РНК. Rinalmo достигает самых современных результатов по нескольким нижестоящим задачам. Примечательно, что мы показываем, что его возможности обобщения могут преодолеть неспособность других методов глубокого обучения для прогнозирования вторичной структуры, чтобы обобщать невидимые семейства РНК.
Используйте следующие команды для установки (предварительные условия: Python>=3.8 и CUDA>=11.8 ):
git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMo
pip install .
pip install flash-attn==2.3.2После установки вы можете легко использовать Rinalmo для получения нуклеотидных представлений:
import torch
from rinalmo . pretrained import get_pretrained_model
DEVICE = "cuda:0"
model , alphabet = get_pretrained_model ( model_name = "giga-v1" )
model = model . to ( device = DEVICE )
model . eval ()
seqs = [ "ACUUUGGCCA" , "CCCGGU" ]
tokens = torch . tensor ( alphabet . batch_tokenize ( seqs ), dtype = torch . int64 , device = DEVICE )
with torch . no_grad (), torch . cuda . amp . autocast ():
outputs = model ( tokens )
print ( outputs [ "representation" ])git clone https://github.com/lbcb-sci/RiNALMo
cd RiNALMoenvironment.yml . # create conda environment for RiNALMo
conda env create -f environment.yml
# activate RiNALMo environment
conda activate rinalmomkdir weights
cd weights
wget https://zenodo.org/records/10725749/files/rinalmo_giga_pretrained.pt # Download fine-tuned weights for secondary structure prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-16s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-23s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-5s_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-srp_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-grp1_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-telomerase_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tmRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-tRNA_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_archiveII-RNaseP_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_ss_bprna_ft.pt
# Download fine-tuned weights for splice-site prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_acceptor_ft.pt
wget https://zenodo.org/records/10725749/files/rinalmo_giga_splice_donor_ft.pt
# Download fine-tuned weights for mean ribosome loading prediction.
wget https://zenodo.org/records/10725749/files/rinalmo_giga_mrl_ft.pt
cd ..Вы также можете скачать предварительно обученные веса для более мелких моделей Rinalmo (Rinalmo-148M (MEGA) и Rinalmo-33,5M (Micro)) по адресу https://drive.google.com/drive/folders/1vgk3gy8c01o0wqfpyx5voy4wy8ebqs5.
Мы обеспечиваем предварительно обученные веса Rinalmo и тонкие веса для трех нижестоящих задач: среднее прогноз нагрузки рибосомы, прогноз вторичной структуры и прогноз сайта сплайсинга. Как для оценки, так и для тонкой настройки используйте сценарии train_<downstream_task>.py .
Чтобы оценить предоставленные тонко настроенные модели Rinalmo и головки прогнозирования, запустите сценарии, используя следующие входные аргументы:
# skip fine-tuning and run the evaluation on the test set
--test_only
# path to the '.pt' file containing fine-tuned model weights
--init_params
# dataset on which you would like to evaluate the fine-tuned model
--dataset
# download and prepare data (if needed)
--prepare_data
# Directory that will contain or already contains training, validation and test data
data_dir
# directory for all the output files
--output_dir Чтобы оценить тонкую настройку модели Rinalmo и головки прогнозирования на наборе тестирования rRNA Archiveii 5S для прогнозирования вторичной структуры, используйте вес веса rinalmo_giga_ss_archiveII-5s_ft.pt . Здесь мы предоставляем пример команды запуска.
python train_sec_struct_prediction.py ./ss_data --test_only --init_params ./weights/rinalmo_giga_ss_archiveII-5s_ft.pt --dataset archiveII_5s --prepare_data --output_dir ./outputs/archiveII/5s/ --accelerator gpu --devices 1
Чтобы точно настраивать rinalmo, используйте --pretrained_rinalmo_weights ./weights/rinalmo_giga_pretrained.pt входной аргумент. Используйте --help , чтобы узнать о других доступных аргументах. Для задачи прогнозирования сплайсинга набор данных и кода предварительной обработки данных доступны по адресу https://git.unistra.fr/nscalzitti/spliceator.git.
Copyright 2024 Labić Lab - AI в геномике
Лицензировано по лицензии Apache, версия 2.0 («Лицензия»); Вы не можете использовать этот файл, кроме как в соответствии с лицензией. Вы можете получить копию лицензии на
http://www.apache.org/licenses/license-2.0
Если это не требуется применимого законодательства или не согласовано в письменной форме, программное обеспечение, распространяемое по лицензии, распределяется по основам «как есть», без каких -либо гарантий или условий, явных или подразумеваемых. См. Лицензию для конкретного языка, регулирующих разрешения и ограничения по лицензии.
Параметры Rinalmo предоставляются в соответствии с условиями лицензии Creative Commons Attribution 4.0 International (CC By 4.0). Вы можете найти подробности по адресу: https://creativecommons.org/licenses/by/4.0/legalcode.
Если вы найдете нашу работу полезной в своем исследовании, пожалуйста, укажите:
@article { penic2024_rinalmo ,
title = { RiNALMo: General-Purpose RNA Language Models Can Generalize Well on Structure Prediction Tasks } ,
author = { Penić, Rafael Josip and Vlašić, Tin and Huber, Roland G. and Wan, Yue and Šikić, Mile } ,
journal = { arXiv preprint arXiv:2403.00043 } ,
year = { 2024 }
}Если у вас есть какие -либо вопросы, пожалуйста, не стесняйтесь написать авторам или открыть проблему.
Эта работа была частично поддержана программой конкурентной исследовательской программы Национального исследовательского фонда (NRF) в рамках проекта идентифицирующей функциональные РНК-третичные структуры в вирусе денге (NRF-CRP27-2021RS-0001) и частично с помощью A*Star в рамках Grant Gap2: A*Star-RNA-модель (A*Star RNA-FM) (I23D1AG079).
Вычислительная работа для статьи была частично выполнена по ресурсам Национального центра суперкомпьютеров, Сингапур https://www.nscc.sg.