[新聞| 2021年8月22日] Sapbert被整合到Nvidia的深度學習工具包Nemo中,作為其實體鏈接模塊(謝謝Nvidia!)。您可以在Google Colab中使用它。
該倉庫擁有(1)我們NAACL 2021論文中提出的Sapbert模型的代碼,數據和預處理的權重:生物醫學實體表示的自我調整預審計; (2)在我們的ACL 2021論文中提出的基準( XL-BEL )的跨語性Sapbert和一個跨語性的生物醫學實體:學習域特殊的表示跨語性生物醫學實體鏈接的表示。
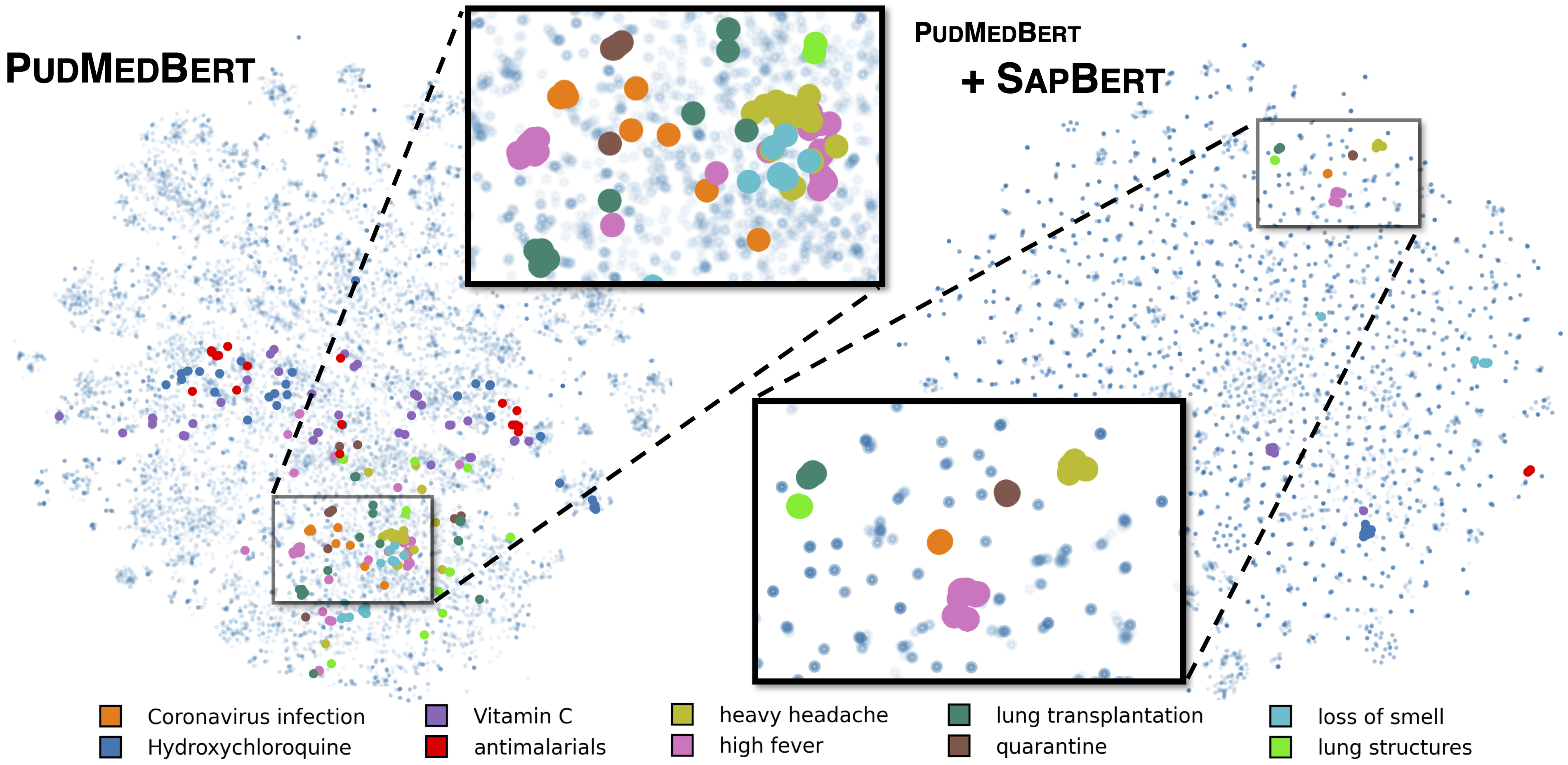
如[Liu等人,NAACL 2021]中所述的標準Sapbert。使用microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext作為基本模型,接受了UMLS 2020AA(僅英語)培訓。對於[Sapbert],請使用[CLS] (池之前)作為輸入的表示;對於[Sapbert-Mean-Token],請在所有令牌中使用平均泵。
如[Liu等人,ACL 2021]中所述的跨語性Sapbert。使用xlm-roberta-base / xlm-roberta-large作為基本模型,接受了UMLS 2020AB(所有語言)培訓。使用[CLS] (池之前)作為輸入的表示。
該代碼用Python 3.8,Torch 1.7.0和HuggingFace Transferes 4.4.2進行了測試。請查看requirements.txt以獲取更多詳細信息。
以下腳本將字符串(實體名稱)列表轉換為嵌入。
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )有關更廣泛的推理示例,請參見推理/inperion_on_snomed.ipynb。
從training_data/generate_pretraining_data.ipynb中提取umls的培訓數據(由於許可問題,我們無法直接發布培訓文件)。
跑步:
>> cd train/
>> ./pretrain.sh 0,1 其中0,1指定GPU設備。
要在您的自定義數據集中進行填充,請以
concept_id || entity_name_1 || entity_name_2
...
其中entity_name_1和entity_name_2是從給定標記的數據集採樣的同義詞對(屬於同一概念概念concept_id )。如果一個概念與數據集中的多個實體名稱相關聯,則可以穿越所有成對組合。
對於使用通用域並行數據(MUSE,Wiki標題或兩者)的跨語性SAP調整,可以在training_data/general_domain_parallel_data/中找到數據。示例腳本: train/xling_train.sh 。
要進行評估(無論是弦詞還是跨語言),請查看evaluation/README.md了解詳細信息。 evaluation/xl_bel/包含[Liu等,ACL 2021]中提出的XL-BEL基準。
薩普伯特:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}跨語言Sapbert和XL-Bel:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}該代碼的一部分是從Biosyn修改的。我們感謝作者將Biosyn開源。
Sapbert已獲得麻省理工學院的許可。有關詳細信息,請參見許可證文件。