[Nachrichten | 22. August 2021] Sapbert ist in das Deep Learning Toolkit Nemo von NVIDIA als das Entitätsverbindenmodul integriert (danke Nvidia!). Sie können damit in diesem Google Colab spielen.
Dieses Repo enthält Code, Daten und vorbereitete Gewichte für (1) das in unserem NAACL 2021-Papier vorgestellte Sapbert -Modell: Selbstausrichtung vorab der Darstellungen der biomedizinischen Entität ; .
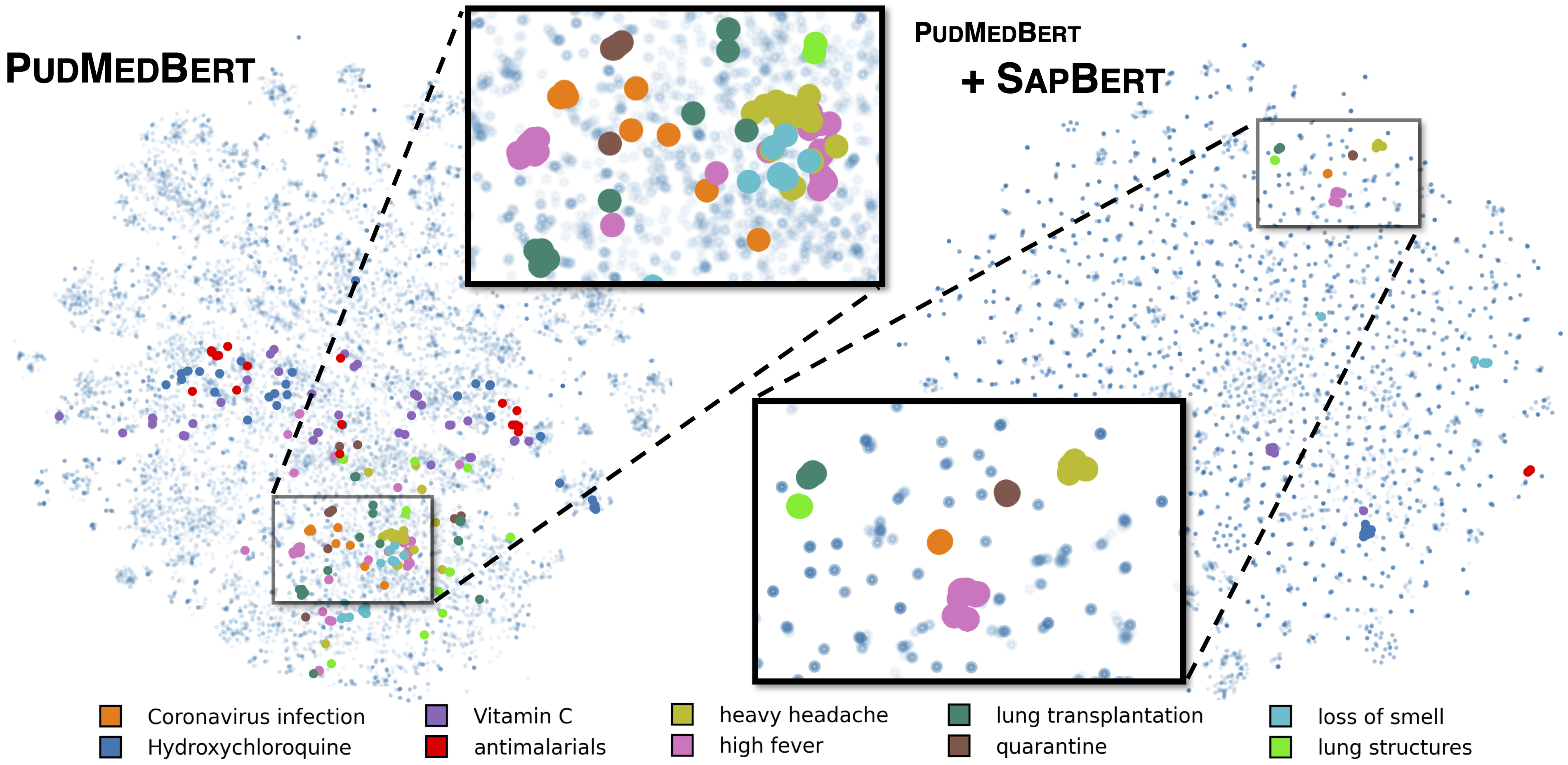
Standard Sapbert, wie in [Liu et al., Naacl 2021] beschrieben. Ausgebildet mit UMLS 2020AA (nur Englisch) unter Verwendung von microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext als Basismodell. Verwenden Sie für [Sapbert] [CLS] (vor dem Pooler) als Darstellung der Eingabe; Verwenden Sie für [Sapbert-Mean-Token] das mittlere Pooling über alle Token.
Kreuzsprachiger Sapbert wie in [Liu et al., ACL 2021] beschrieben. Ausgebildet mit UMLS 2020AB (alle Sprachen) unter Verwendung von xlm-roberta-base / xlm-roberta-large als Basismodell. Verwenden Sie [CLS] (vor dem Pooler) als Darstellung der Eingabe.
Der Code wird mit Python 3.8, Torch 1.7.0 und Huggingface -Transformatoren 4.4.2 getestet. Bitte sehen Sie requirements.txt für weitere Details.
Das folgende Skript wandelt eine Liste von Zeichenfolgen (Entitätsnamen) in Einbettungen um.
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )Bitte beachten Sie Inference/Inference_on_Snomed.ipynb, um ein umfangreicheres Inferenzbeispiel zu erhalten.
Extrahieren Sie Trainingsdaten aus UMLs, die in training_data/generate_pretraining_data.ipynb als inSrtructed extrahieren (wir können die Trainingsdatei aufgrund von Lizenzierungsproblemen nicht direkt veröffentlichen).
Laufen:
>> cd train/
>> ./pretrain.sh 0,1 wobei 0,1 die GPU -Geräte angeben.
Generieren Sie Daten im Format von FELLINGNING in Ihrem benutzerdefinierten Datensatz, um Daten zu erstellen
concept_id || entity_name_1 || entity_name_2
...
wobei entity_name_1 und entity_name_2 Synonympaare (zu demselben concept_id gehörten) aus einem bestimmten Datensatz abgetastet sind. Wenn ein Konzept mit mehreren Entitätsnamen im Datensatz zugeordnet ist, können Sie alle paarweisen Kombinationen durchqueren.
Für die Kreuzung mit allgemeinen Domänen-Paralleldaten (Muse, Wiki-Titel oder beides) finden Sie die Daten in training_data/general_domain_parallel_data/ . Ein Beispielskript: train/xling_train.sh .
Für die Bewertung (sowohl monlingual als auch bricht) finden Sie evaluation/README.md für Einzelheiten. evaluation/xl_bel/ enthält den in [Liu et al., ACL 2021] vorgeschlagenen XL-Bel-Benchmark.
Sapbert:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}Cross-Lingual Sapbert und XL-Bel:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}Teile des Codes werden von Biosyn geändert. Wir schätzen die Autoren für die Herstellung von Biosyn Open-Sourcing.
Sapbert ist MIT lizenziert. Weitere Informationen finden Sie in der Lizenzdatei.