[ข่าว | 22 ส.ค. 2021] Sapbert ถูกรวมเข้ากับชุดเครื่องมือการเรียนรู้เชิงลึกของ Nvidia Nemo เป็นโมดูลเชื่อมโยงเอนทิตี (ขอบคุณ Nvidia!) คุณสามารถเล่นกับมันใน Google Colab นี้
repo นี้ถือรหัสข้อมูลและน้ำหนักที่ได้รับการปรับแต่งสำหรับ (1) โมเดล Sapbert ที่นำเสนอในกระดาษ NAACL 2021 ของเรา: การจัดตำแหน่งตนเองก่อนการจัดเรียงตัวเองสำหรับการเป็นตัวแทนของกิจการชีวการแพทย์ ; (2) Sapbert แบบข้ามภาษา และเอนทิตีชีวการแพทย์ข้ามภาษา ( XL-bel ) ที่นำเสนอในบทความ ACL 2021 ของเรา: การเรียนรู้การเป็นตัวแทนของโดเมน-พิเศษสำหรับการเชื่อมโยงเอนทิตีชีวการแพทย์ข้ามภาษา
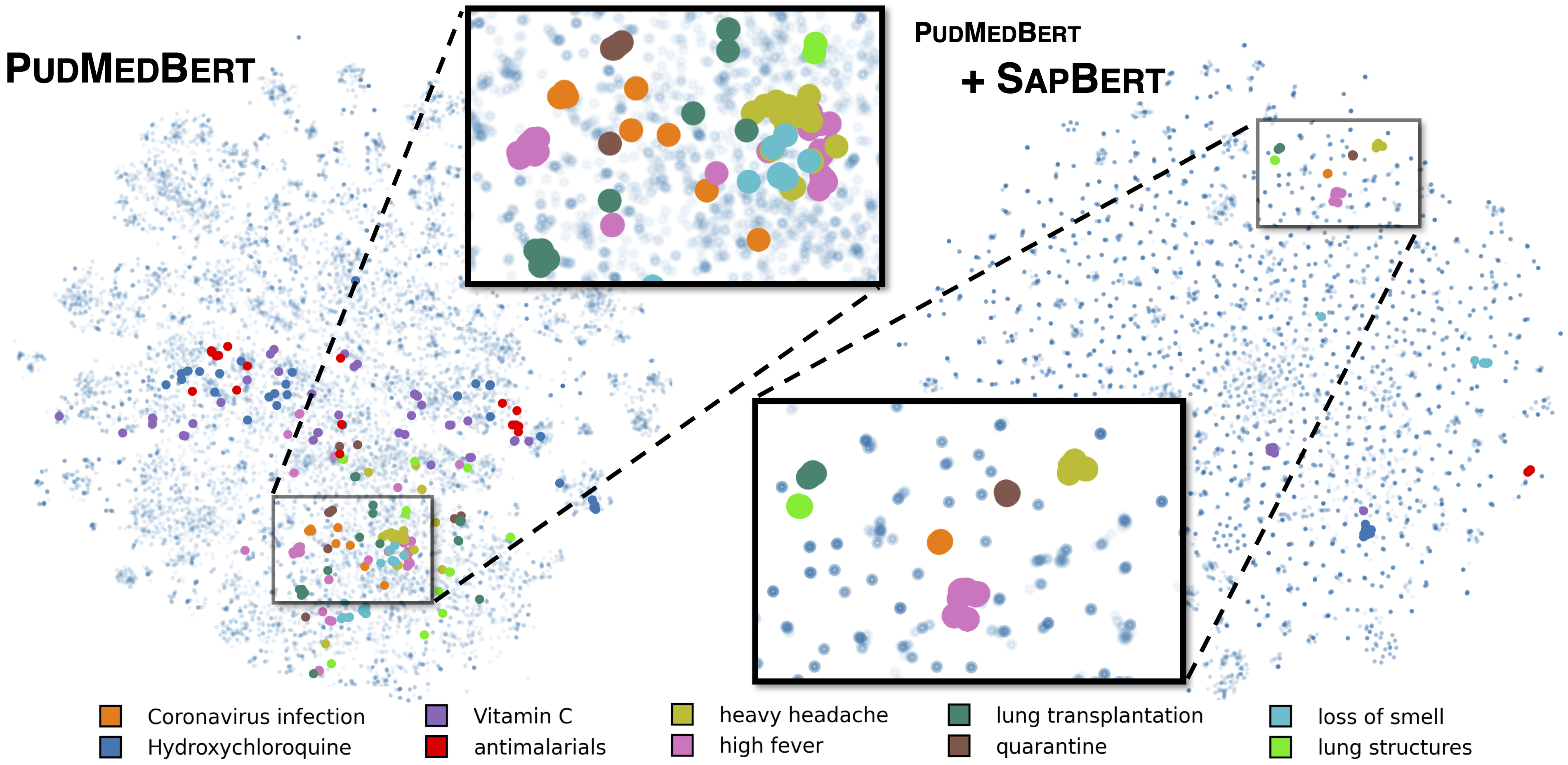
Sapbert Standard ตามที่อธิบายไว้ใน [Liu et al., NAACL 2021] ได้รับการฝึกฝนกับ UMLS 2020AA (ภาษาอังกฤษเท่านั้น) โดยใช้ microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext เป็นรุ่นพื้นฐาน สำหรับ [Sapbert] ใช้ [CLS] (ก่อน pooler) เป็นตัวแทนของอินพุต; สำหรับ [Sapbert-Mean-Token] ให้ใช้ค่าเฉลี่ยการระบายความหมายข้ามโทเค็นทั้งหมด
Sapbert แบบข้ามภาษาตามที่อธิบายไว้ใน [Liu et al., ACL 2021] ได้รับการฝึกฝนด้วย UMLS 2020AB (ทุกภาษา) โดยใช้ xlm-roberta-base / xlm-roberta-large เป็นรุ่นพื้นฐาน ใช้ [CLS] (ก่อน pooler) เป็นตัวแทนของอินพุต
รหัสถูกทดสอบด้วย Python 3.8, Torch 1.7.0 และ HuggingFace Transformers 4.4.2 โปรดดู requirements.txt สำหรับรายละเอียดเพิ่มเติม
สคริปต์ต่อไปนี้จะแปลงรายการสตริง (ชื่อเอนทิตี) เป็น embeddings
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )โปรดดู Inference/Inference_on_snomed.ipynb สำหรับตัวอย่างการอนุมานที่กว้างขวางยิ่งขึ้น
แยกข้อมูลการฝึกอบรมจาก UMLs ที่ไม่ได้ใช้งานใน training_data/generate_pretraining_data.ipynb (เราไม่สามารถปล่อยไฟล์การฝึกอบรมโดยตรงเนื่องจากปัญหาการออกใบอนุญาต)
วิ่ง:
>> cd train/
>> ./pretrain.sh 0,1 โดยที่ 0,1 ระบุอุปกรณ์ GPU
สำหรับ finetuning ในชุดข้อมูลที่กำหนดเองของคุณสร้างข้อมูลในรูปแบบของ
concept_id || entity_name_1 || entity_name_2
...
โดยที่ entity_name_1 และ entity_name_2 เป็นคู่คำพ้องความหมาย (เป็นของ concept_id แนวคิดเดียวกัน) ตัวอย่างจากชุดข้อมูลที่มีป้ายกำกับที่กำหนด หากแนวคิดหนึ่งเชื่อมโยงกับชื่อเอนทิตีหลายชื่อในชุดข้อมูลคุณสามารถสำรวจชุดค่าผสมทั้งหมดได้
สำหรับการปรับแต่ง sap-lingual กับข้อมูลขนานโดเมนทั่วไป (Muse, ชื่อวิกิหรือทั้งสองอย่าง) ข้อมูลสามารถพบได้ใน training_data/general_domain_parallel_data/ ตัวอย่างสคริปต์: train/xling_train.sh
สำหรับการประเมินผล (ทั้ง monlingual และ cross-lingual) โปรดดู evaluation/README.md สำหรับรายละเอียด evaluation/xl_bel/ มีเกณฑ์มาตรฐาน XL-bel ที่เสนอใน [Liu et al., ACL 2021]
Sapbert:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}Sapbert และ XL-bel:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}บางส่วนของรหัสได้รับการแก้ไขจาก Biosyn เราขอขอบคุณผู้เขียนที่เปิดแหล่งข้อมูล Biosyn
Sapbert ได้รับใบอนุญาต MIT ดูไฟล์ใบอนุญาตสำหรับรายละเอียด