[Новости | 22 августа 2021 г.] Сапберт интегрирован в инструментарий NVIDIA Nemo Nemo в качестве модуля связывания сущностей (спасибо, nvidia!). Вы можете играть с этим в этом Google Colab.
Этот репо содержит код, данные и предварительно подготовленные веса для (1) модели Сапберта , представленной в нашей бумаге NAACL 2021: предварительное подготовку самооплаты для представлений биомедицинских сущностей ; (2) Крестовой Сапберт и межсочевое биомедицинское сущность, связывающая ( XL-BEL ), предложенные в нашей статье ACL 2021 Paper: специфические для обучения представления для связывания биомедицинских сущностей .
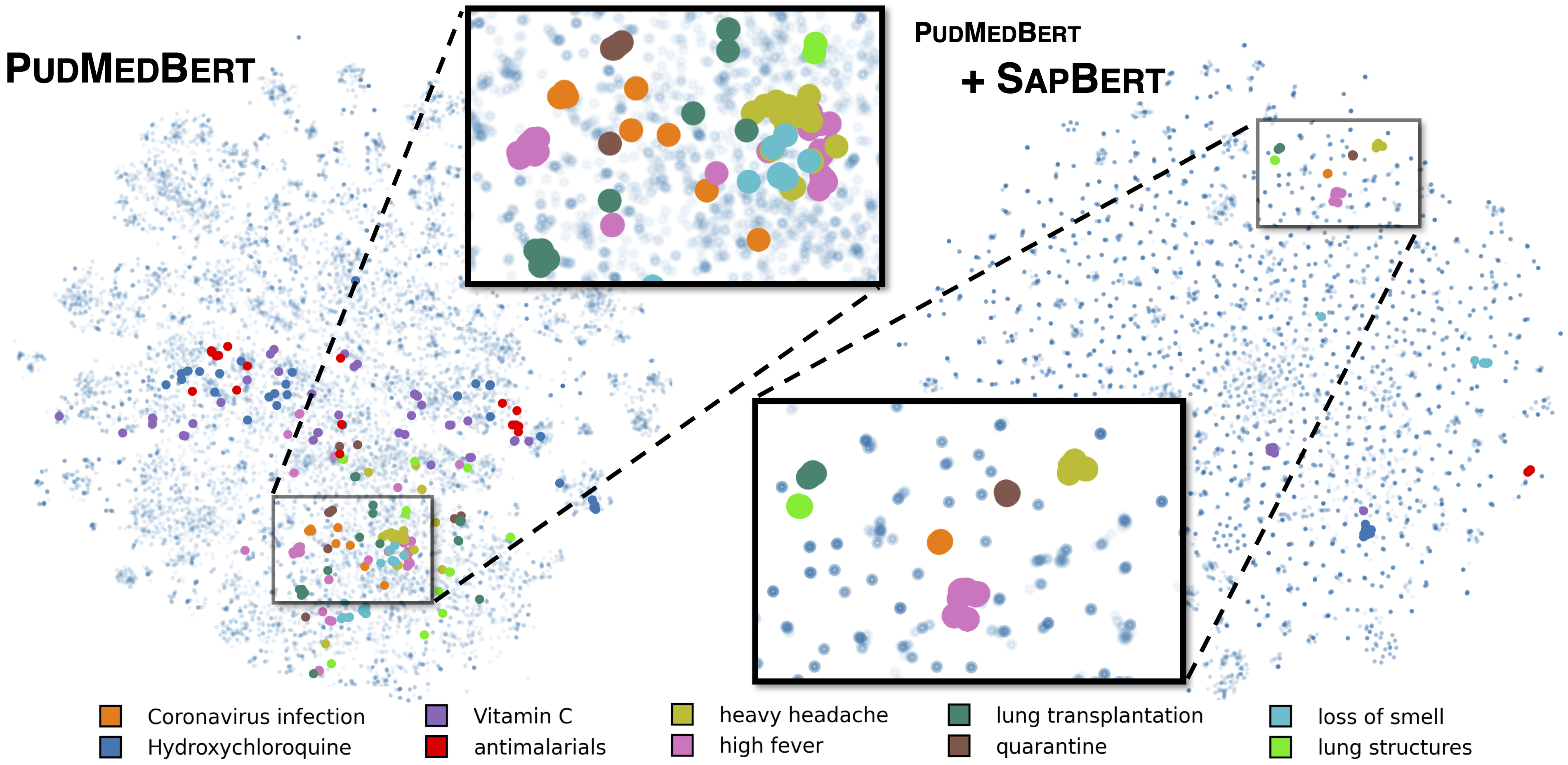
Стандартный Сапберт, как описано в [Liu et al., NAACL 2021]. Обученный UMLS 2020AA (только английский), используя microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext в качестве базовой модели. Для [Sapbert] используйте [CLS] (до балу) в качестве представления ввода; Для [Sapbert-Mean-Token] используйте среднее количество токенов.
Крестный сапберт, как описано в [Liu et al., ACL 2021]. Обученный UMLS 2020AB (все языки), используя xlm-roberta-base / xlm-roberta-large в качестве базовой модели. Используйте [CLS] (до балу) в качестве представления ввода.
Код тестируется с помощью Python 3.8, Torch 1.7.0 и трансформаторов Huggingface 4.4.2. Пожалуйста, просмотрите requirements.txt для получения более подробной информации.
Следующий скрипт преобразует список строк (имена сущностей) в встраивания.
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )Пожалуйста, смотрите snucement/spence_on_snomed.ipynb для более обширного примера вывода.
Извлеките учебные данные из UMLS как взимаемые в training_data/generate_pretraining_data.ipynb (мы не можем напрямую выпустить файл обучения из -за проблем лицензирования).
Бегать:
>> cd train/
>> ./pretrain.sh 0,1 где 0,1 указывает устройства GPU.
Для создания в вашем настраиваемом наборе данных генерируйте данные в формате
concept_id || entity_name_1 || entity_name_2
...
где entity_name_1 и entity_name_2 являются парами синонимов (принадлежащие к той же concept_id ), отобранные из данного помеченного набора данных. Если одна концепция связана с несколькими именами объектов в наборе данных, вы можете пройти все парные комбинации.
Для перекрестной настройки SAP с параллельными данными общего домена (Muse, Wiki названия или оба) данные можно найти в training_data/general_domain_parallel_data/ . Пример сценария: train/xling_train.sh .
Для оценки (как Monlingual, так и CrossLyling), пожалуйста, просмотрите evaluation/README.md для получения подробной информации. evaluation/xl_bel/ содержит эталон XL-Bel, предложенный в [Liu et al., ACL 2021].
Сапберт:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}Кросс-лингвиальный сапберт и XL-бель:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}Части кода изменяются из Biosyn. Мы ценим авторов за создание биосин с открытым исходным кодом.
Сапберт лицензирован MIT. Смотрите файл лицензии для получения подробной информации.