[뉴스 | 2021 년 8 월 22 일] Sapbert는 NVIDIA의 딥 러닝 툴킷 NEMO에 엔터티 링크 모듈로 통합되었습니다 (NVIDIA 감사합니다!). 이 Google Colab에서 플레이 할 수 있습니다.
이 repo는 (1) NAACL 2021 논문에 제시된 SAPBERT 모델에 대한 코드, 데이터 및 사전 상인 가중치를 보유하고 있습니다. (2) ACL 2021 논문에서 제안 된 벤치 마크 ( XL-BEL ) 의 교차 언어 Sapbert 및 교차-언어 생물 의학 실체 : 교차-언어 생물 의학적 단체 연결에 대한 학습 영역 별 표현 .
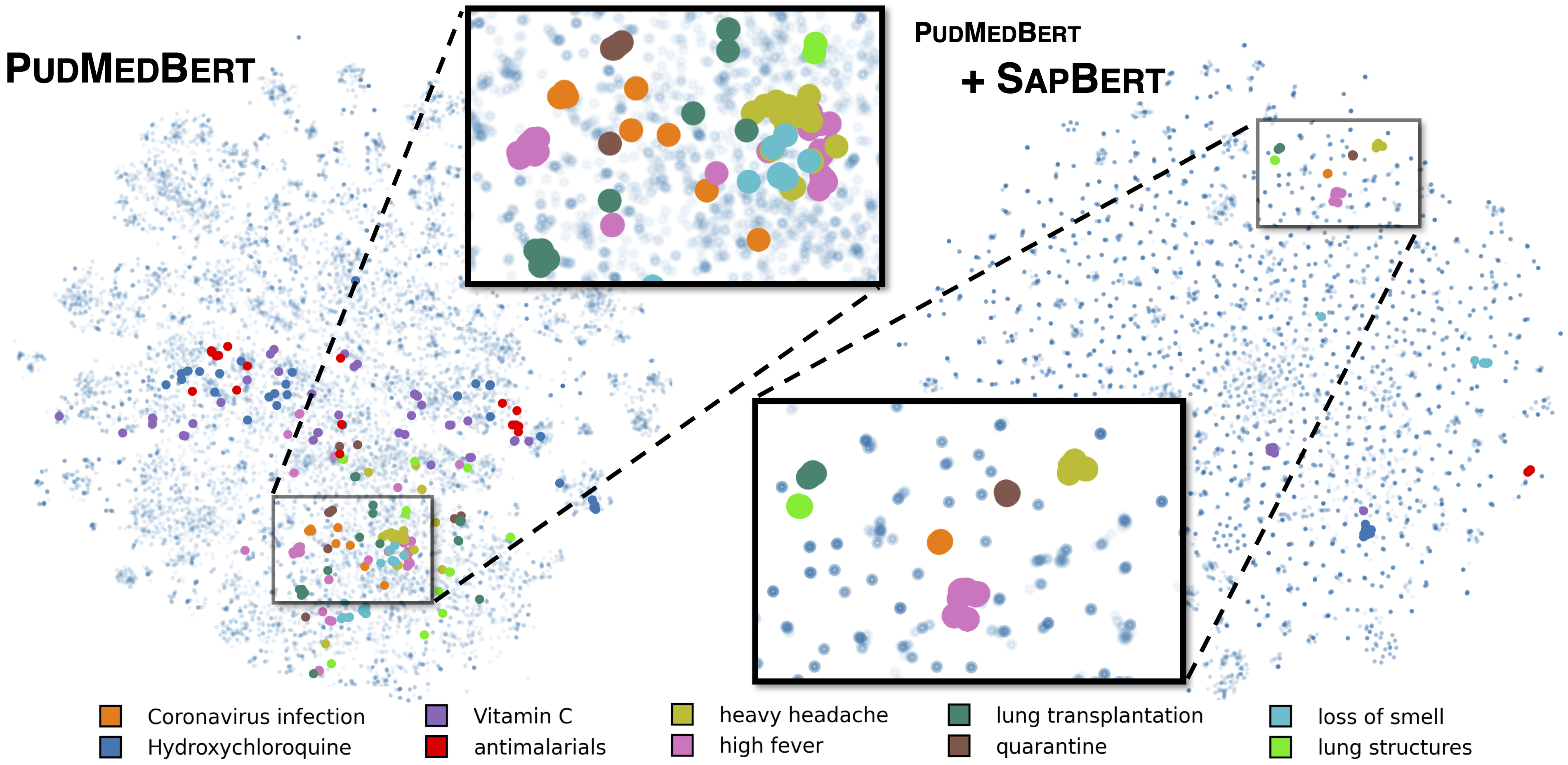
[Liu et al., NAACL 2021]에 설명 된 표준 Sapbert. microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext 기본 모델로 사용하여 UMLS 2020AA (English Only)로 교육. [Sapbert]의 경우 [CLS] (Pooler 이전)를 입력의 표현으로 사용하십시오. [Sapbert-Mean-Token]의 경우 모든 토큰에 대한 평균 설비를 사용하십시오.
[Liu et al., ACL 2021]에 설명 된 바와 같이 언어 간 Sapbert. xlm-roberta-base / xlm-roberta-large 기본 모델로 사용하여 UMLS 2020AB (모든 언어)로 교육을 받았습니다. [CLS] (Pooler 이전)를 입력의 표현으로 사용하십시오.
코드는 Python 3.8, Torch 1.7.0 및 Huggingface Transformers 4.4.2로 테스트됩니다. 자세한 내용은 requirements.txt 참조하십시오.
다음 스크립트는 문자열 목록 (엔티티 이름)을 임베딩으로 변환합니다.
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )보다 광범위한 추론 예는 추론/inference_on_snomed.ipynb를 참조하십시오.
training_data/generate_pretraining_data.ipynb 에서 insrtrated로 UML의 교육 데이터를 추출합니다 (라이센스 문제로 인해 교육 파일을 직접 해제 할 수 없습니다).
달리다:
>> cd train/
>> ./pretrain.sh 0,1 여기서 0,1 GPU 장치를 지정합니다.
사용자 정의 된 데이터 세트에 대한 결합을 위해서는 형식으로 데이터를 생성하십시오.
concept_id || entity_name_1 || entity_name_2
...
여기서 entity_name_1 및 entity_name_2 주어진 라벨이 붙은 데이터 세트에서 샘플링 된 동의어 쌍 (동일한 개념 concept_id 에 속함)입니다. 하나의 개념이 데이터 세트의 여러 엔티티 이름과 관련이있는 경우 모든 쌍별 조합을 가로 질러 갈 수 있습니다.
일반적인 도메인 병렬 데이터 (Muse, Wiki Titles 또는 둘 다)와 함께하는 교차 SAP 튜닝의 경우 데이터는 training_data/general_domain_parallel_data/ 에서 찾을 수 있습니다. 예제 스크립트 : train/xling_train.sh .
평가 (Monlingual 및 Cross-LINUAL)는 자세한 내용은 evaluation/README.md 참조하십시오. evaluation/xl_bel/ 에는 [Liu et al., ACL 2021]에 제안 된 XL- 벨 벤치 마크가 포함되어 있습니다.
Sapbert :
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}교차 언어 Sapbert 및 XL-Bel :
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}코드의 일부는 Biosyn에서 수정됩니다. Biosyn을 열린 소스를 만드는 저자에게 감사드립니다.
Sapbert는 MIT 라이센스가 부여되었습니다. 자세한 내용은 라이센스 파일을 참조하십시오.