[News | 22 août 2021] Sapbert est intégré à la boîte à outils Deep Learning de Nvidia Nemo comme module de liaison entité (merci nvidia!). Vous pouvez jouer avec dans ce Google Colab.
Ce repo contient du code, des données et des poids pré-entraînés pour (1) le modèle de Sapbert présenté dans notre article NAACL 2021: prélèvement d'auto-alignement pour les représentations d'entités biomédicales ; (2) Le sapbert inter-greatrice et une entité biomédicale inter-greatrice liant la référence ( XL-Bel ) proposée dans notre article ACL 2021: Représentations spécialisées du domaine d'apprentissage pour la liaison de l'entité biomédicale cross-linguale .
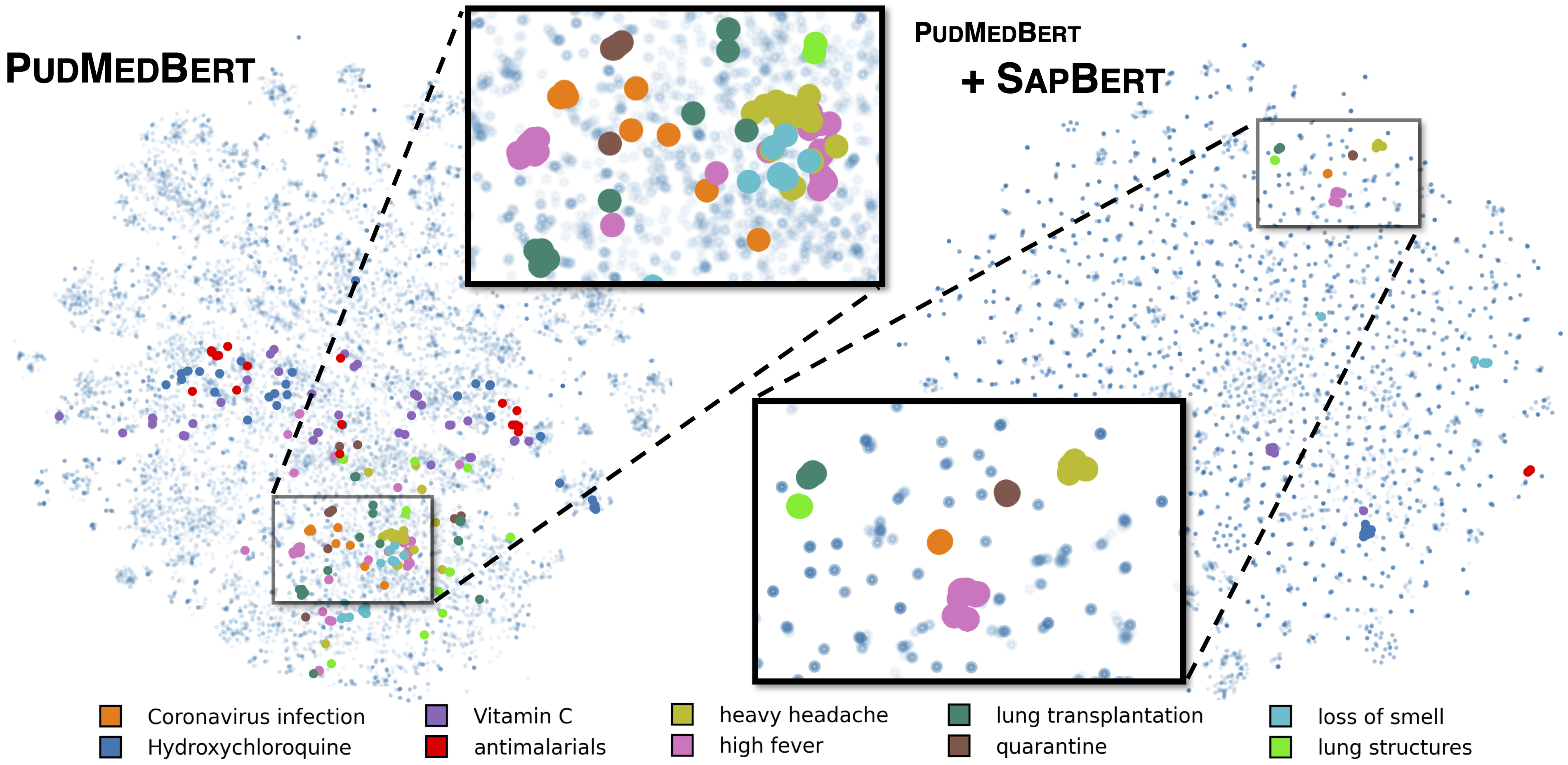
Sapbert standard comme décrit dans [Liu et al., NAACL 2021]. Formé avec UMLS 2020AA (anglais uniquement), en utilisant microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext comme modèle de base. Pour [Sapbert], utilisez [CLS] (avant Pooler) comme représentation de l'entrée; Pour [Sapbert-Mean-Token], utilisez des réchauffeurs moyens sur tous les jetons.
Sapbert inter-lingue comme décrit dans [Liu et al., ACL 2021]. Formé avec UMLS 2020AB (toutes les langues), en utilisant xlm-roberta-base / xlm-roberta-large comme modèle de base. Utilisez [CLS] (avant le Pooler) comme représentation de l'entrée.
Le code est testé avec Python 3.8, Torch 1.7.0 et HuggingFace Transformers 4.4.2. Veuillez afficher requirements.txt pour plus de détails.
Le script suivant convertit une liste de chaînes (noms d'entités) en intégres.
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )Veuillez consulter Inference / INFERGE_ON_SNOMED.IPYNB pour un exemple d'inférence plus étendu.
Extraire les données de formation des UMLS telles que inscrites dans training_data/generate_pretraining_data.ipynb (nous ne pouvons pas publier directement le fichier de formation en raison de problèmes de licence).
Courir:
>> cd train/
>> ./pretrain.sh 0,1 où 0,1 spécifie les appareils GPU.
Pour la fin de la fin de votre ensemble de données personnalisé, générez des données sous le format de
concept_id || entity_name_1 || entity_name_2
...
où entity_name_1 et entity_name_2 sont des paires de synonymes (appartenant au même concept concept_id ) échantillonnés à partir d'un ensemble de données étiqueté donné. Si un concept est associé à plusieurs noms d'entités dans l'ensemble de données, vous pouvez traverser toutes les combinaisons par paires.
Pour le tun sap interdictionnel avec des données parallèles de domaine général (muse, titres wiki, ou les deux), les données peuvent être trouvées dans training_data/general_domain_parallel_data/ . Un exemple de script: train/xling_train.sh .
Pour l'évaluation (à la fois monlingues et cross-lingual), veuillez consulter evaluation/README.md pour plus de détails. evaluation/xl_bel/ Contient la référence XL-Bel proposée dans [Liu et al., ACL 2021].
Sapbert:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}Cross-lingue Sapbert et XL-Bel:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}Les parties du code sont modifiées à partir de Biosyn. Nous apprécions les auteurs d'avoir fait de Biosyn Open-source.
Sapbert est sous licence MIT. Voir le fichier de licence pour plus de détails.