[Noticias | 22 de agosto de 2021] Sapbert está integrado en el kit de herramientas de aprendizaje profundo de NVIDIA como su módulo de enlace de entidad (¡gracias nvidia!). Puedes jugar con él en este Colab de Google.
Este repositorio contiene el código, los datos y los pesos previos al detenido para (1) el modelo Sapbert presentado en nuestro documento NAACL 2021: previagración de autoalineación para representaciones de entidades biomédicas ; (2) El sapbert interlingüístico y una entidad biomédica interlingüística que vincula el punto de referencia ( XL-Bel ) propuesto en nuestro artículo ACL 2021: representaciones especializadas por dominio de aprendizaje para la vinculación de entidad biomédica interlingüística .
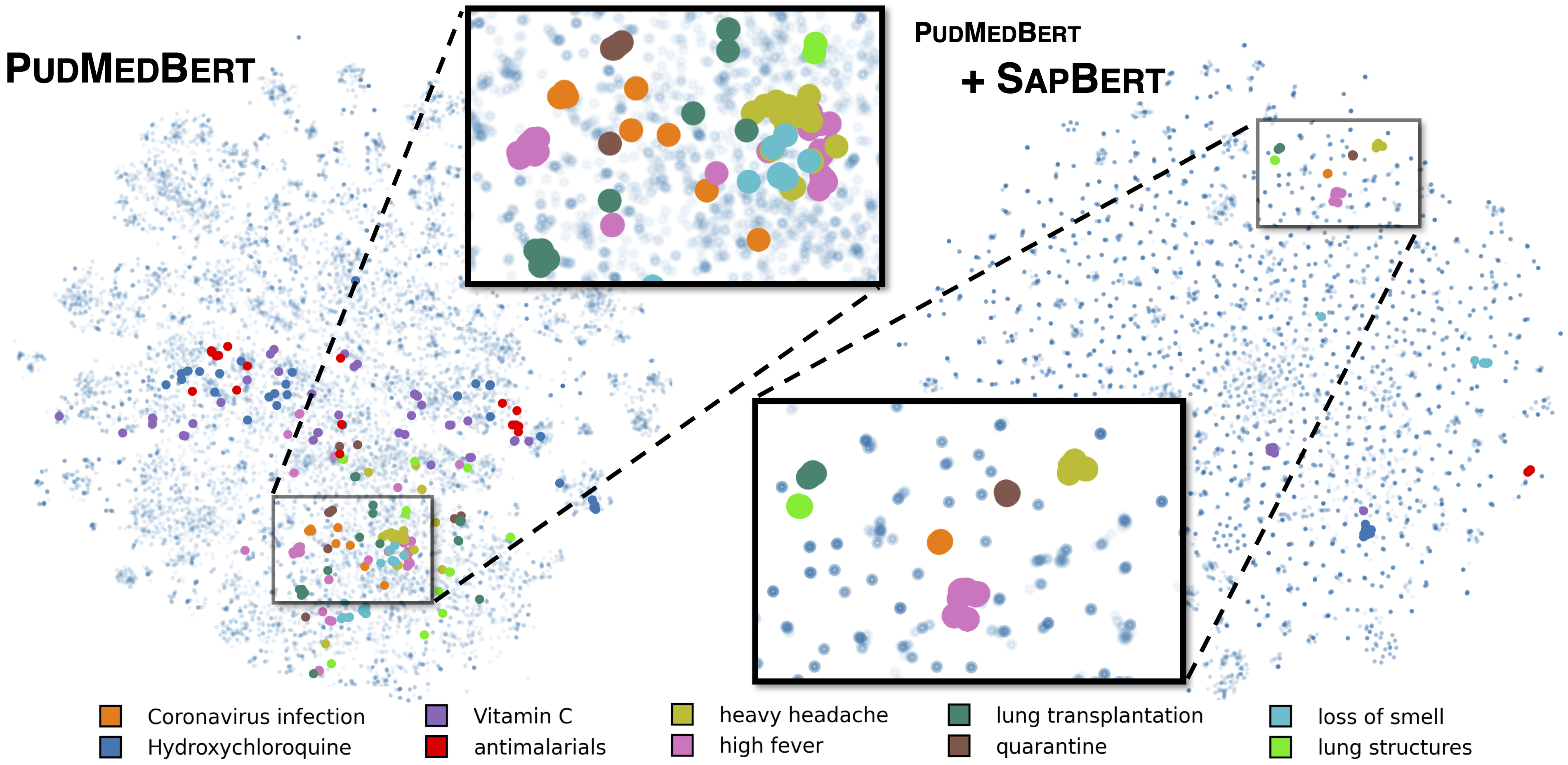
Sapbert estándar como se describe en [Liu et al., NAACL 2021]. Entrenado con UMLS 2020AA (solo en inglés), utilizando microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext como modelo base. Para [Sapbert], use [CLS] (antes del grupo) como la representación de la entrada; Para [Sapbert-Mean-Token], use la medición media en todas las fichas.
Sapbert interlingüe como se describe en [Liu et al., ACL 2021]. Entrenado con UMLS 2020AB (todos los idiomas), utilizando xlm-roberta-base / xlm-roberta-large como modelo base. Use [CLS] (antes del grupo) como la representación de la entrada.
El código se prueba con Python 3.8, Torch 1.7.0 y Huggingface Transformers 4.4.2. Ver requirements.txt para obtener más detalles.
El siguiente script convierte una lista de cadenas (nombres de entidad) en incrustaciones.
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )Consulte Inference/Inference_on_snomed.ipynb para un ejemplo de inferencia más extenso.
Extraiga datos de entrenamiento de UMLS como Insrtructed en training_data/generate_pretraining_data.ipynb (no podemos liberar directamente el archivo de capacitación debido a problemas de licencia).
Correr:
>> cd train/
>> ./pretrain.sh 0,1 donde 0,1 especifica los dispositivos GPU.
Para la sintonización de finos en su conjunto de datos personalizado, genere datos en el formato de
concept_id || entity_name_1 || entity_name_2
...
donde entity_name_1 y entity_name_2 son pares de sinónimos (pertenecientes al mismo concepto concept_id ) muestreados de un conjunto de datos etiquetado dado. Si un concepto está asociado con múltiples nombres de entidades en el conjunto de datos, podría atravesar todas las combinaciones por pares.
Para el ajuste de SAP multilingüe con datos paralelos de dominio general (Muse, Wiki títulos o ambos), los datos se pueden encontrar en training_data/general_domain_parallel_data/ . Un script de ejemplo: train/xling_train.sh .
Para la evaluación (tanto monlingual como interlingüe), vea evaluation/README.md para obtener más detalles. evaluation/xl_bel/ contiene el punto de referencia XL-Bel propuesto en [Liu et al., ACL 2021].
Sapbert:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}Sapbert y XL-Bel interlingüe:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}Las partes del código se modifican de Biosyn. Apreciamos a los autores por hacer biosyn de código abierto.
Sapbert tiene licencia MIT. Consulte el archivo de licencia para obtener más detalles.