[ニュース| 2021年8月22日] Sapbertは、モジュールをリンクするエンティティとしてNvidiaのDeep Learning Toolkit Nemoに統合されています(Nvidiaに感謝します!)。このGoogle Colabでプレイできます。
このレポは、 (1) NAACL 2021ペーパーに示されているSAPBERTモデルのコード、データ、および事前処理された重みを保持しています:生物医学的エンティティ表現の自己調整事前。 (2) ACL 2021論文で提案されているベンチマーク( XL-BEL )をリンクするクロスリングルの生物医学的エンティティ:言語横断生物医学的エンティティリンクの学習ドメイン特異的表現。
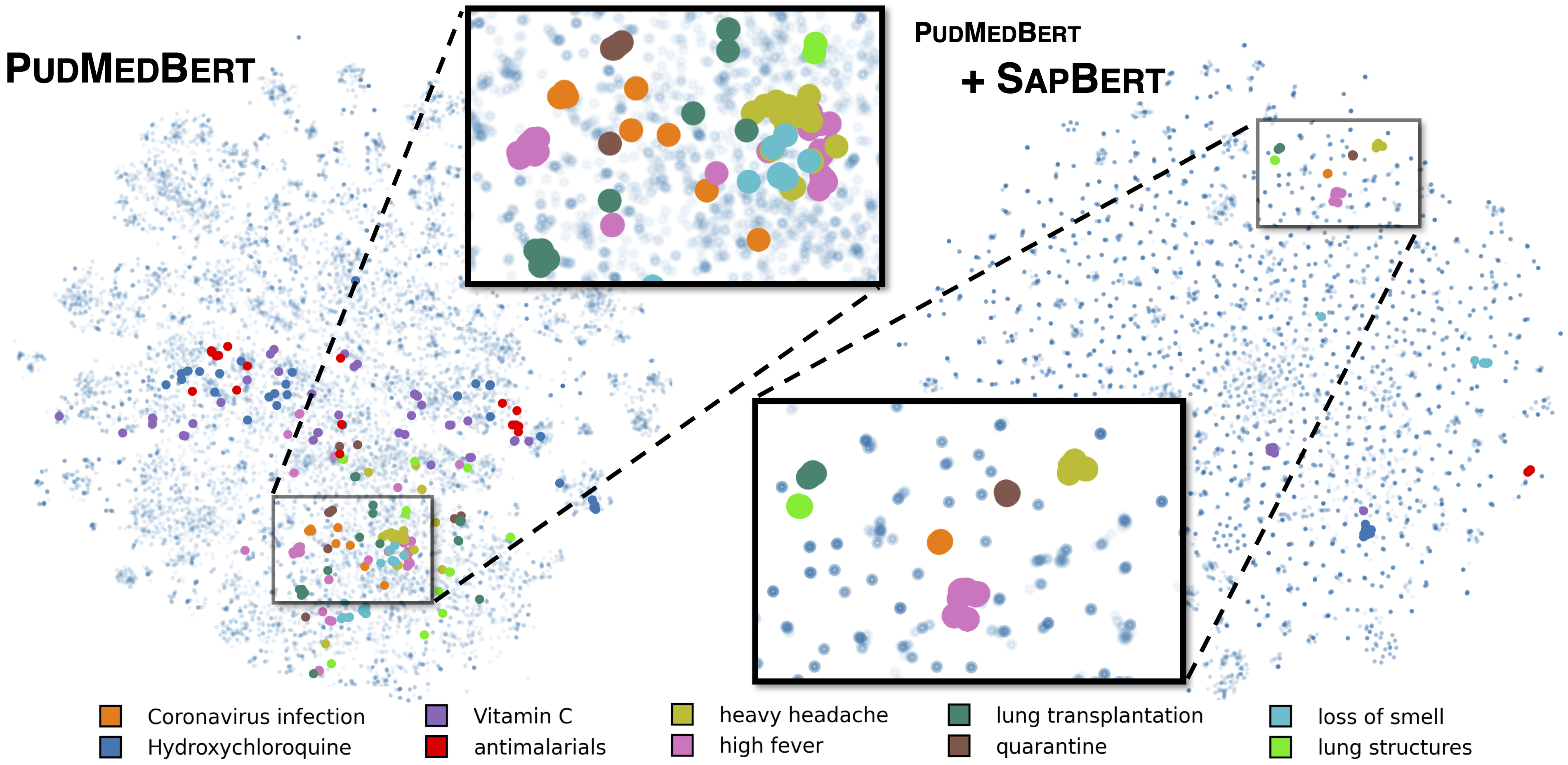
[Liu et al。、naacl 2021]に記載されている標準的なサプバート。 microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext基本モデルとして使用して、UMLS 2020AA(英語のみ)で訓練されています。 [sapbert]の場合、 [CLS] (プーラーの前)を入力の表現として使用します。 [Sapbert-Mean-Token]の場合、すべてのトークンで平均プーリングを使用します。
[Liu et al。、ACL 2021]に記載されているように、横断的サプバート。 xlm-roberta-base / xlm-roberta-large基本モデルとして使用して、UMLS 2020AB(すべての言語)で訓練されています。 [CLS] (プーラーの前)を入力の表現として使用します。
このコードは、Python 3.8、Torch 1.7.0、およびHuggingface Transformers 4.4.2でテストされています。詳細については、 requirements.txtをご覧ください。
次のスクリプトは、文字列(エンティティ名)のリストを埋め込みに変換します。
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )より広範な推論の例については、Inference/Inference_on_snomed.ipynbを参照してください。
training_data/generate_pretraining_data.ipynbで構造化されたUMLSからトレーニングデータを抽出します(ライセンスの問題のためにトレーニングファイルを直接リリースすることはできません)。
走る:
>> cd train/
>> ./pretrain.sh 0,1 ここで、 0,1 GPUデバイスを指定します。
カスタマイズされたデータセットでFinetuningを使用するには、の形式でデータを生成します
concept_id || entity_name_1 || entity_name_2
...
entity_name_1およびentity_name_2は、特定のラベル付きデータセットからサンプリングされた同義語のペア(同じ概念concept_idに属する)です。 1つの概念がデータセット内の複数のエンティティ名に関連付けられている場合、すべてのペアワイズの組み合わせを通過できます。
一般的なドメインの並列データ(Muse、Wikiタイトル、またはその両方)を使用した横断的なSAP調整の場合、データはtraining_data/general_domain_parallel_data/に記載されています。例:スクリプト: train/xling_train.sh 。
評価(一流および横断的な両方の両方)については、詳細についてはevaluation/README.mdをご覧ください。 evaluation/xl_bel/には、[Liu et al。、ACL 2021]で提案されているXL-BELベンチマークが含まれています。
SAPBERT:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}横断的なサプバートとXLベル:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}コードの一部はBiosynから変更されています。 Biosynをオープンソースにしてくれた著者に感謝します。
SapbertはMITライセンスを取得しています。詳細については、ライセンスファイルを参照してください。