[Notícias | 22 de agosto de 2021] Sapbert é integrado ao Deep Learning Toolkit Nemo da NVIDIA como seu módulo de ligação da entidade (obrigado Nvidia!). Você pode brincar com ele neste Google Colab.
Este repositório mantém código, dados e pesos pré-elaborados para (1) o modelo de Sapbert apresentado em nosso artigo NAACL 2021: auto-alinhamento pré-treinamento para representações de entidades biomédicas ; (2) O Sapbert cruzado e uma entidade biomédica transversal que liga a referência ( XL-Bel ) em nosso artigo da ACL 2021: Aprendendo representações especiais de domínio para a entidade biomédica transversal .
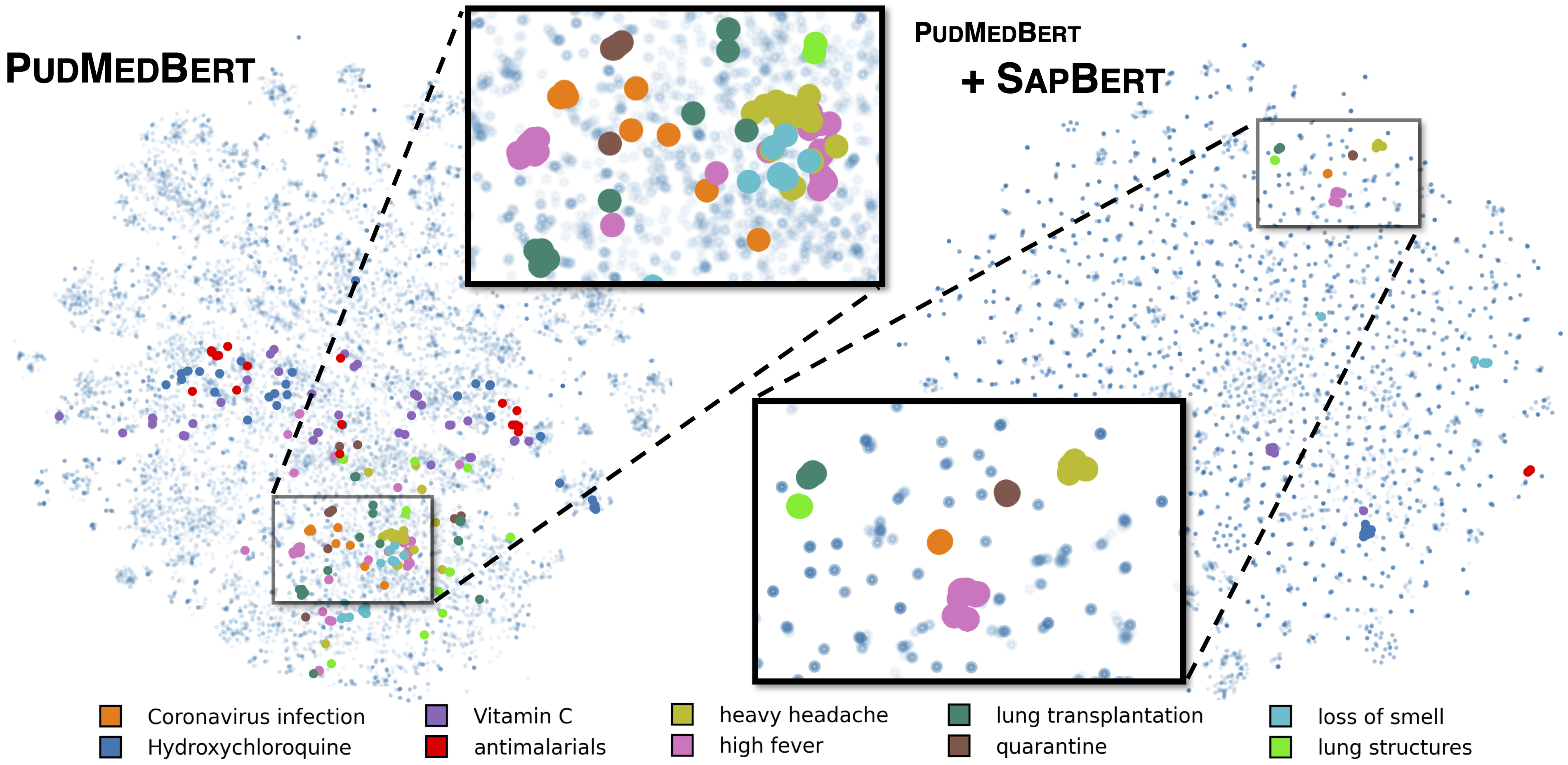
Sapbert padrão, conforme descrito em [Liu et al., NAACL 2021]. Treinado com o UMLS 2020AA (somente em inglês), usando microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext como modelo básico. Para [Sapbert], use [CLS] (antes do pooler) como representação da entrada; Para [Sapbert-Mean-Token], use pool de todos os tokens.
Sapbert cruzado como descrito em [Liu et al., ACL 2021]. Treinado com UMLS 2020AB (todos os idiomas), usando xlm-roberta-base / xlm-roberta-large como modelo base. Use [CLS] (antes do pooler) como a representação da entrada.
O código é testado com o Python 3.8, Torch 1.7.0 e Huggingface Transformers 4.4.2. Consulte requirements.txt para obter mais detalhes.
O script a seguir converte uma lista de strings (nomes de entidades) em incorporação.
import numpy as np
import torch
from tqdm . auto import tqdm
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" )
model = AutoModel . from_pretrained ( "cambridgeltl/SapBERT-from-PubMedBERT-fulltext" ). cuda ()
# replace with your own list of entity names
all_names = [ "covid-19" , "Coronavirus infection" , "high fever" , "Tumor of posterior wall of oropharynx" ]
bs = 128 # batch size during inference
all_embs = []
for i in tqdm ( np . arange ( 0 , len ( all_names ), bs )):
toks = tokenizer . batch_encode_plus ( all_names [ i : i + bs ],
padding = "max_length" ,
max_length = 25 ,
truncation = True ,
return_tensors = "pt" )
toks_cuda = {}
for k , v in toks . items ():
toks_cuda [ k ] = v . cuda ()
cls_rep = model ( ** toks_cuda )[ 0 ][:, 0 ,:] # use CLS representation as the embedding
all_embs . append ( cls_rep . cpu (). detach (). numpy ())
all_embs = np . concatenate ( all_embs , axis = 0 )Consulte a inferência/inference_on_snomed.ipynb para um exemplo de inferência mais extenso.
Extrair dados de treinamento do UMLS, conforme insrtrutos em training_data/generate_pretraining_data.ipynb (não podemos liberar diretamente o arquivo de treinamento devido a problemas de licenciamento).
Correr:
>> cd train/
>> ./pretrain.sh 0,1 onde 0,1 especifica os dispositivos GPU.
Para o Finetuning no seu conjunto de dados personalizado, gerar dados no formato de
concept_id || entity_name_1 || entity_name_2
...
onde entity_name_1 e entity_name_2 são pares de sinônimos (pertencentes ao mesmo conceito concept_id ) amostrados de um determinado conjunto de dados rotulado. Se um conceito estiver associado a vários nomes de entidades no conjunto de dados, você poderá atravessar todas as combinações pareadas.
Para ajuste de seiva transversal com dados paralelos de domínio geral (Muse, títulos wiki ou ambos), os dados podem ser encontrados em training_data/general_domain_parallel_data/ . Um exemplo de script: train/xling_train.sh .
Para avaliação (monlingual e transversal), consulte evaluation/README.md para obter detalhes. evaluation/xl_bel/ contém a referência XL-Bel proposta em [Liu et al., ACL 2021].
Sapbert:
@inproceedings { liu2021self ,
title = { Self-Alignment Pretraining for Biomedical Entity Representations } ,
author = { Liu, Fangyu and Shareghi, Ehsan and Meng, Zaiqiao and Basaldella, Marco and Collier, Nigel } ,
booktitle = { Proceedings of the 2021 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies } ,
pages = { 4228--4238 } ,
month = jun,
year = { 2021 }
}Sapbert e XL-Bel cruzados:
@inproceedings { liu2021learning ,
title = { Learning Domain-Specialised Representations for Cross-Lingual Biomedical Entity Linking } ,
author = { Liu, Fangyu and Vuli{'c}, Ivan and Korhonen, Anna and Collier, Nigel } ,
booktitle = { Proceedings of ACL-IJCNLP 2021 } ,
pages = { 565--574 } ,
month = aug,
year = { 2021 }
}Partes do código são modificadas a partir de Biosyn. Agradecemos os autores por fazer de origem aberta do Biosyn.
Sapbert é licenciado pelo MIT. Consulte o arquivo de licença para obter detalhes.