Diffdock口袋是在口袋級別運行的全部原子對接模型。它實現了側鏈扭轉角度的擴散,並針對計算生成的結構進行了優化。在此存儲庫中,您將找到用於訓練模型,運行推理,可視化的代碼以及我們用來實現論文中列出的數字的權重。該存儲庫最初是來自Diffdock的叉子,因此某些命令似乎似乎很熟悉 - 但是它已經在許多地方進行了擴展,改編和更改,以至於您無法期望這兩個程序的任何兼容性。請注意,不要混合這兩個程序!
如果您想要一種簡單的運行推理方法,則可以在此處使用HuggingFace接口。
如果您在此存儲庫中有任何問題,請隨意創建任何問題,或者PRS!請考慮行為準則。
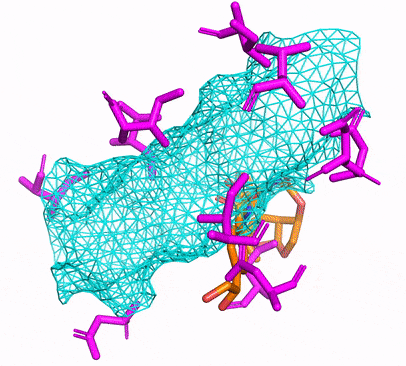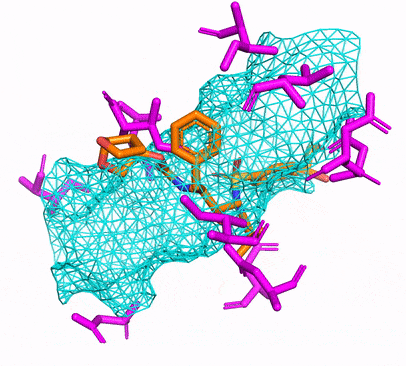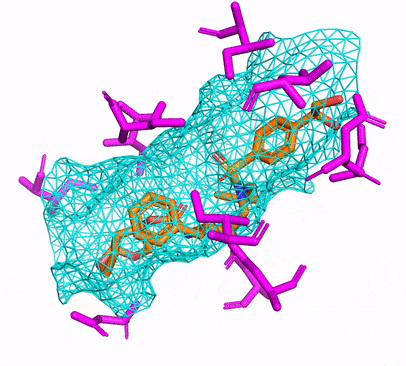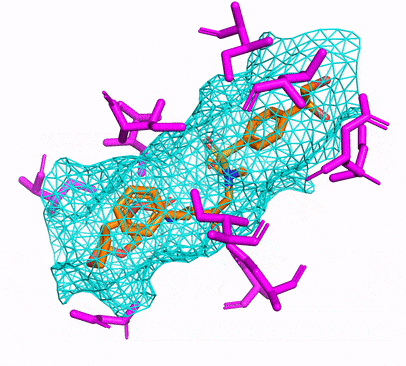
您可以通過簡單地運行來創建CPU或CUDA CONDA環境
conda env create -f environment[-cpu].yml
具有或不帶CPU標誌。建議使用CUDA版本,因為它要快得多。
在這個最小的示例中,我們使用複雜的3dpf並用柔性的側面cahins重新插入配體。我們繪製40個樣本,並用置信度模型對它們進行排名。所有預測將存儲在results/中。
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
在這裡,我們指定了--keep_local_structures標誌以不修改配體。這樣,可以自動確定口袋和要模型的側鏈。
如果此命令耗盡內存,則可以減少--batch_size或--samples_per_complex參數。降低--batch_size不會影響結果的質量,而是會增加運行時。
計算生成的結構的類似命令是:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
為了在Top-1預測的配體姿勢上執行能量最小化,您可以使用--relax標誌。
如果您知道自己的裝訂袋以及要彈性的側鏈,則可以指定它們。在這種情況下,您也可以刪除--keep_local_structures 。我們建議僅使用提供的模型,這些模型與柔性氨基酸至少在任何重配體原子的3.5a之內至少具有一個重原子。這樣可以防止分佈數據。
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
或者,如果您沒有配體,也可以使用笑容表示。
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
但是,在這種情況下,您必須指定口袋中心和靈活的側鏈。
如果您要處理多個複合物,請使用標誌來查看--protein_ligand_csv data/protein_ligand_example_csv.csv data/protein_ligand_example_csv /protein_ligand_example_csv.csv.csv 。這使您可以一次指定多個複合物的推斷,您可以在其中指定口袋中心和每個複合物的柔性側鏈。
選擇口袋中心時,最好確定靠近結合位點的氨基酸併計算其C-α坐標的平均值。在我們的培訓中,我們選擇了所有氨基酸,其中C-Alpha原子在配體的任何重原子的5a之內。
我們正在使用PDBBIND,您可以從許多來源下載。但是,我們還在此處提供了預處理的PDBBIND數據集,您可以通過unzip打開包裝。所有蛋白質都與PDBFixer固定並對齊,以最大程度地減少口袋中的RMSD。將其解放到data/PDBBIND_atomCorrected 。
訓練大分模型:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
模型權重保存在workdir目錄中,可用於推斷。
為了訓練置信度模型,您可以使用先前訓練的分數模型來生成樣品。但是,我們選擇訓練另一個具有較高最大配體翻譯的(較小)得分模型,以便置信模型看到更多的樣本,並且可以更快地生成訓練數據。
要訓練這個具有較高最大翻譯的較小分數模型,請運行以下內容:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
用於生成樣品來訓練置信模型的分數模型不必與推斷期間與該置信度模型一起使用的分數模型相同。
通過運行以下操作來訓練置信度模型:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
首先是--cache_creation_id 1然後--cache_creation_id 2等。最多4
現在,一切都經過訓練,您可以使用新模型進行推斷:)。
如果您在研究中使用diffdock口袋,請引用以下論文:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
感謝@jsilter使此代碼更易於使用和創建HuggingFace接口!