Diffdock-Pocket เป็นแบบจำลองการเชื่อมต่ออะตอมทั้งหมดที่ทำงานในระดับพกพา มันใช้การแพร่กระจายผ่านมุมบิดโซ่ด้านข้างและได้รับการปรับให้เหมาะสมสำหรับโครงสร้างที่สร้างขึ้นโดยการคำนวณ ในที่เก็บนี้คุณจะพบรหัสเพื่อฝึกอบรมแบบจำลองการอนุมานการสร้างภาพและน้ำหนักที่เราใช้เพื่อให้ได้ตัวเลขที่นำเสนอในกระดาษ ที่เก็บนี้เดิมเป็นส้อมจาก Diffdock ดังนั้นคำสั่งบางอย่างอาจดูคุ้นเคย - แต่มันได้รับการขยายดัดแปลงและเปลี่ยนแปลงในหลาย ๆ สถานที่ที่คุณไม่สามารถคาดหวังความเข้ากันได้ของทั้งสองโปรแกรม ระวังและอย่าผสมโปรแกรมทั้งสองนี้!
หากคุณต้องการวิธีที่ง่ายในการอนุมานคุณสามารถใช้อินเทอร์เฟซ HuggingFace ได้ที่นี่
อย่าลังเลที่จะสร้างปัญหาหรือ PRS หากคุณมีปัญหาใด ๆ กับที่เก็บนี้! โปรดพิจารณาจรรยาบรรณ
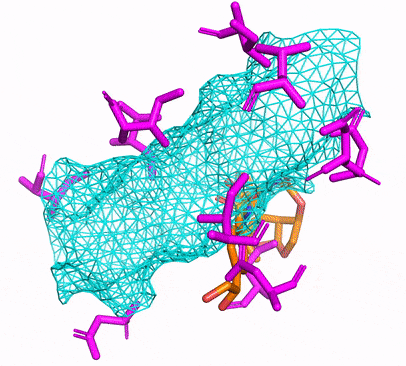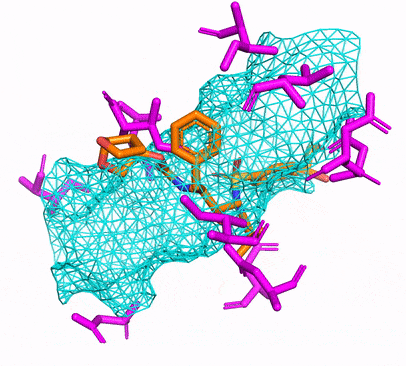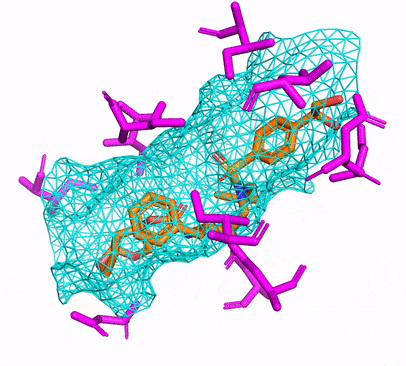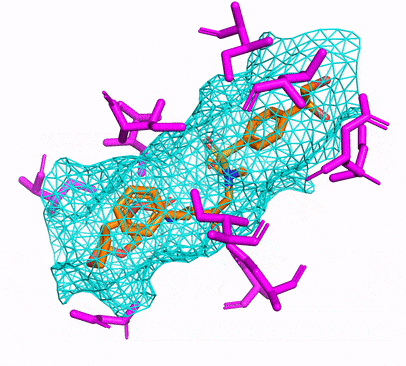
คุณสามารถสร้างสภาพแวดล้อม CPU หรือ CUDA Conda ได้โดยเพียงแค่วิ่ง
conda env create -f environment[-cpu].yml
มีหรือไม่มีธง CPU แนะนำให้ใช้เวอร์ชัน CUDA เนื่องจากเร็วกว่ามาก
ในตัวอย่างที่น้อยที่สุดนี้เราใช้ 3dpf ที่ซับซ้อนและนำลิแกนด์มาใช้ใหม่ด้วย Cahins ด้านที่ยืดหยุ่น เราวาดตัวอย่าง 40 ตัวอย่างและจัดอันดับด้วยรูปแบบความมั่นใจ การคาดการณ์ทั้งหมดจะถูกเก็บไว้ใน results/
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
ที่นี่เราได้ระบุธง --keep_local_structures เพื่อไม่แก้ไขแกนด์ ด้วยสิ่งนี้กระเป๋าและโซ่ด้านใดที่จะสร้างแบบจำลองความยืดหยุ่นโดยอัตโนมัติ
หากคำสั่งนี้หมดจากหน่วยความจำคุณสามารถลดพารามิเตอร์ --batch_size หรือพารามิเตอร์ --samples_per_complex การลดลง --batch_size จะไม่ส่งผลกระทบต่อคุณภาพของผลลัพธ์ แต่จะเพิ่มรันไทม์
คำสั่งที่คล้ายกันสำหรับโครงสร้างที่สร้างโดยการคำนวณคือ:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
ในการลดการลดพลังงานในโพสต์ลิแกนด์ที่คาดการณ์ไว้ 1 อันดับแรกคุณสามารถใช้ธง --relax ได้
หากคุณรู้ว่ากระเป๋าที่มีผลผูกพันของคุณและโซ่ด้านใดที่จะสร้างแบบจำลองที่ยืดหยุ่นคุณสามารถระบุได้ ในกรณีนี้คุณสามารถวาง --keep_local_structures เราแนะนำให้ใช้โมเดลที่ให้มาของเราเฉพาะกับกรดอะมิโนที่ยืดหยุ่นซึ่งมีอะตอมหนักอย่างน้อยหนึ่งอะตอมภายใน 3.5a ของอะตอมลิแกนด์หนัก สิ่งนี้จะช่วยป้องกันข้อมูลนอกการกระจาย
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
หรือถ้าคุณไม่มีแกนด์อยู่ในมือคุณสามารถใช้การแสดงรอยยิ้มได้
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
อย่างไรก็ตามในกรณีนี้คุณต้องระบุศูนย์พ็อกเก็ตและโซ่ด้านข้างที่ยืดหยุ่น
หากคุณกำลังจัดการกับคอมเพล็กซ์หลายอย่างให้ดูที่ data/protein_ligand_example_csv ด้วยการตั้งค่าสถานะ --protein_ligand_csv data/protein_ligand_example_csv.csv สิ่งนี้ช่วยให้คุณสามารถระบุการอนุมานได้หลายคอมเพล็กซ์พร้อมกันซึ่งคุณสามารถระบุศูนย์กลางของกระเป๋าและโซ่ด้านข้างที่ยืดหยุ่นสำหรับแต่ละคอมเพล็กซ์
เมื่อเลือกตรงกลางกระเป๋าจะเป็นการดีที่สุดที่จะกำหนดกรดอะมิโนที่อยู่ใกล้กับไซต์ที่มีผลผูกพันและคำนวณค่าเฉลี่ยของพิกัด C-alpha ของพวกเขา ในการฝึกอบรมของเราเราได้เลือกกรดอะมิโนทั้งหมดที่อะตอม C-alpha ของพวกเขาอยู่ภายใน 5A ของอะตอมหนักของแกนด์
เราใช้ PDBBIND ซึ่งคุณสามารถดาวน์โหลดได้จากหลาย ๆ แหล่ง อย่างไรก็ตามเรายังจัดทำชุดข้อมูล PDBBIND ที่ผ่านการประมวลผลไว้ล่วงหน้าที่นี่ซึ่งคุณสามารถแกะสลักด้วย unzip ได้ โปรตีนทั้งหมดได้รับการแก้ไขด้วย PDBFixer และจัดตำแหน่งเพื่อลด RMSD ในกระเป๋า แกะออกไปยัง data/PDBBIND_atomCorrected
ฝึกอบรมรุ่นคะแนนขนาดใหญ่:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
น้ำหนักแบบจำลองจะถูกบันทึกไว้ในไดเรกทอรี workdir และสามารถใช้สำหรับการอนุมาน
ในการฝึกอบรมโมเดลความมั่นใจคุณสามารถใช้โมเดลคะแนนที่ผ่านการฝึกอบรมมาก่อนเพื่อสร้างตัวอย่าง อย่างไรก็ตามเราเลือกที่จะฝึกอบรมแบบจำลองคะแนน (เล็กกว่า) อีกครั้งด้วยการแปลแกนด์สูงสุดที่สูงขึ้นเพื่อให้แบบจำลองความมั่นใจเห็นตัวอย่างที่หลากหลายมากขึ้นและข้อมูลการฝึกอบรมสามารถผลิตได้เร็วขึ้น
ในการฝึกอบรมรุ่นคะแนนที่เล็กกว่านี้ด้วยการแปลสูงสุดให้สูงขึ้นให้เรียกใช้สิ่งต่อไปนี้:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
รูปแบบคะแนนที่ใช้ในการสร้างตัวอย่างเพื่อฝึกอบรมโมเดลความมั่นใจไม่จำเป็นต้องเหมือนกับโมเดลคะแนนที่ใช้กับโมเดลความมั่นใจในระหว่างการอนุมาน
ฝึกอบรมโมเดลความมั่นใจโดยใช้สิ่งต่อไปนี้:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
อันดับแรกด้วย --cache_creation_id 1 จากนั้น --cache_creation_id 2 เป็นต้นสูงสุด 4
ตอนนี้ทุกอย่างได้รับการฝึกฝนและคุณสามารถอนุมานกับรุ่นใหม่ของคุณ :)
หากคุณใช้ Diffdock-Pocket ในการวิจัยของคุณโปรดอ้างอิงเอกสารต่อไปนี้:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
ขอบคุณ @JSilter ที่ทำให้รหัสนี้ใช้งานง่ายขึ้นและสำหรับการสร้างอินเทอร์เฟซ HuggingFace!