Diffdock Pocket es un modelo de acoplamiento de átomo que funciona en el nivel de bolsillo. Implementa la difusión sobre los ángulos de torsión de la cadena lateral y está optimizado para estructuras generadas computacionalmente. En este repositorio, encontrará el código para entrenar un modelo, ejecutar inferencia, visualizaciones y los pesos que utilizamos para lograr los números presentados en el documento. Este repositorio es originalmente una bifurcación de Diffdock, por lo que algunos de los comandos pueden parecer familiares, pero se ha extendido, adaptado y cambiado en tantos lugares que no puede esperar ninguna compatibilidad de los dos programas. ¡Tenga en cuenta y no mezcle estos dos programas!
Si desea una manera fácil de ejecutar una inferencia, puede usar la interfaz Huggingface aquí.
¡No dude en crear algún problema o PRS si tiene algún problema con este repositorio! Considere el código de conducta.
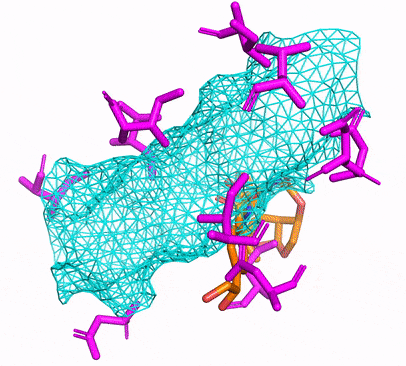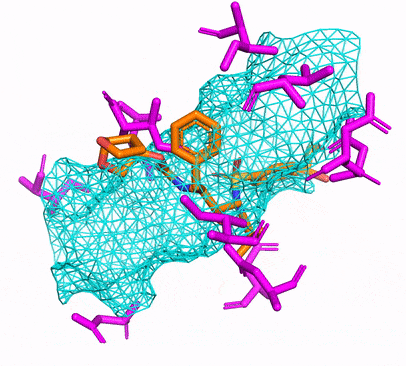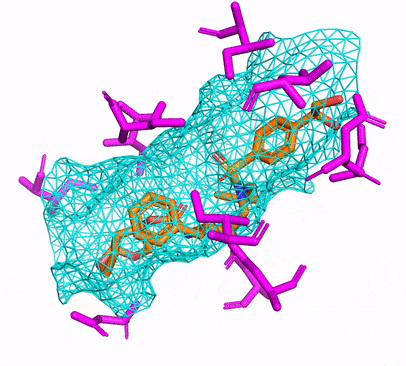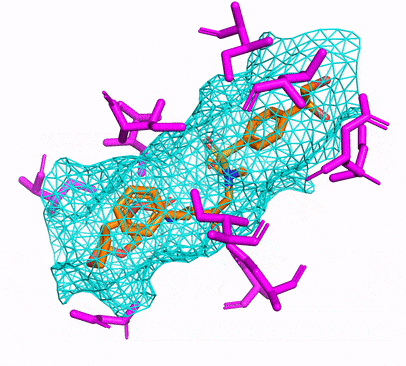
Puede crear el entorno CPU o CUDA Conda simplemente ejecutando
conda env create -f environment[-cpu].yml
con o sin la bandera de la CPU. Se recomienda la versión CUDA, ya que es mucho más rápida.
En este ejemplo mínimo, usamos el complejo 3dpf y volvemos a colocar el ligando con Cahins laterales flexibles. Dibujamos 40 muestras y las clasificamos con el modelo de confianza. Todas las predicciones se almacenarán en results/ .
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
Aquí, especificamos el indicador --keep_local_structures para no modificar el ligando. Con esto, el bolsillo y qué cadenas laterales se determinan automáticamente.
Si este comando se agota fuera de la memoria, puede disminuir el parámetro --batch_size o el --samples_per_complex . Bajando --batch_size no afectará la calidad de los resultados, sino que aumentará el tiempo de ejecución.
Un comando similar para las estructuras generadas computacionalmente sería:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
Para realizar la minimización de energía en la pose de ligando predicha top-1, puede usar el indicador --relax .
Si conoce su bolsillo vinculante y qué cadenas laterales modelar como flexibles, puede especificarlas. En este caso, también puede soltar --keep_local_structures . Aconsejamos usar nuestro modelo proporcionado solo con aminoácidos flexibles que tienen al menos un átomo pesado dentro de 3.5A de cualquier átomo de ligando pesado. Esto evita los datos fuera de distribución.
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
O si no tiene un ligando a mano, también puede usar la representación de sonrisas.
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
Sin embargo, en este caso, debe especificar su centro de bolsillo y las cadenas laterales flexibles.
Si está tratando con múltiples complejos, eche un vistazo a data/protein_ligand_example_csv con el indicador --protein_ligand_csv data/protein_ligand_example_csv.csv . This allows you to specify inference on multiple complexes at once, where you can specify the pocket center and flexible side chains for each complex.
Al elegir el centro de bolsillo, es mejor determinar los aminoácidos que están cerca del sitio de unión y calcular la media de sus coordenadas C-alfa. En nuestro entrenamiento, hemos elegido todos los aminoácidos donde su átomo c-alfa se encuentra dentro de 5A de cualquier átomo pesado del ligando.
Estamos utilizando PDBBind que puede descargar de muchas fuentes. Sin embargo, también proporcionamos un conjunto de datos PDBBind preprocesado aquí que puede desempaquetar con unzip . Todas las proteínas se han solucionado con PDBFixer y alineadas para minimizar el RMSD en el bolsillo. Desempaquélo a data/PDBBIND_atomCorrected .
Entrena el modelo de puntaje grande:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
Los pesos del modelo se guardan en el directorio workdir y se pueden usar para la inferencia.
Para entrenar el modelo de confianza, puede usar el modelo de puntaje previamente entrenado para generar las muestras. Sin embargo, optamos por entrenar otro modelo de puntaje (más pequeño) con una traducción máxima de ligando más alta, de modo que el modelo de confianza ve muestras más diversas y los datos de entrenamiento se pueden producir más rápidamente.
Para entrenar este modelo de puntaje más pequeño con una traducción máxima más alta, ejecute lo siguiente:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
El modelo de puntaje utilizado para generar las muestras para entrenar el modelo de confianza no tiene que ser el mismo que el modelo de puntaje que se usa con ese modelo de confianza durante la inferencia.
Entrena el modelo de confianza ejecutando lo siguiente:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
Primero con --cache_creation_id 1 luego --cache_creation_id 2 etc. hasta 4
Ahora todo está entrenado y puedes ejecutar una inferencia con tu nuevo modelo :).
Si usa Diffdock Pocket en su investigación, cite el siguiente documento:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
¡Gracias @jsilter por hacer que este código sea más fácil de usar y por crear la interfaz Huggingface!