Diffdock口袋是在口袋级别运行的全部原子对接模型。它实现了侧链扭转角度的扩散,并针对计算生成的结构进行了优化。在此存储库中,您将找到用于训练模型,运行推理,可视化的代码以及我们用来实现论文中列出的数字的权重。该存储库最初是来自Diffdock的叉子,因此某些命令似乎似乎很熟悉 - 但是它已经在许多地方进行了扩展,改编和更改,以至于您无法期望这两个程序的任何兼容性。请注意,不要混合这两个程序!
如果您想要一种简单的运行推理方法,则可以在此处使用HuggingFace接口。
如果您在此存储库中有任何问题,请随意创建任何问题,或者PRS!请考虑行为准则。
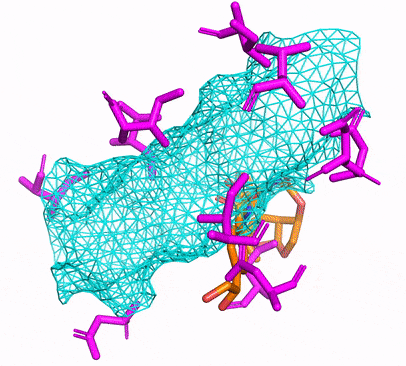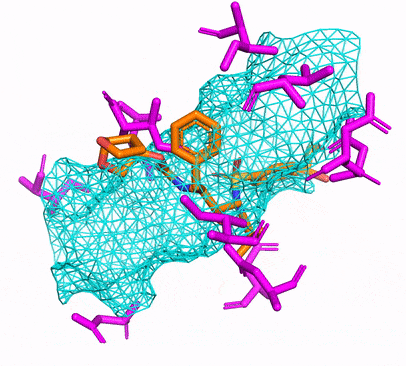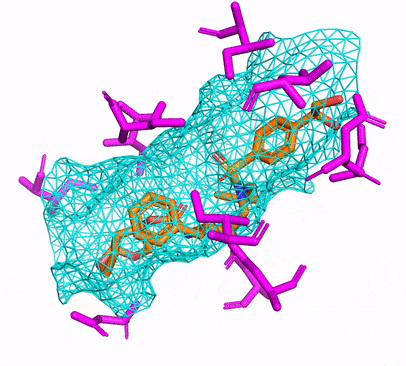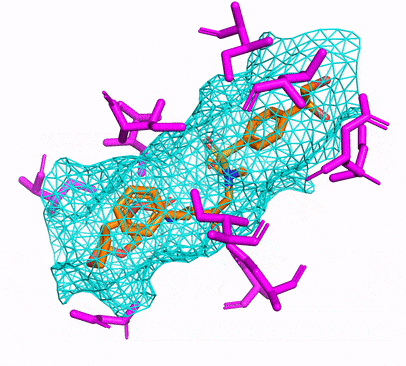
您可以通过简单地运行来创建CPU或CUDA CONDA环境
conda env create -f environment[-cpu].yml
具有或不带CPU标志。建议使用CUDA版本,因为它要快得多。
在这个最小的示例中,我们使用复杂的3dpf并用柔性的侧面cahins重新插入配体。我们绘制40个样本,并用置信度模型对它们进行排名。所有预测将存储在results/中。
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
在这里,我们指定了--keep_local_structures标志以不修改配体。这样,可以自动确定口袋和要模型的侧链。
如果此命令耗尽内存,则可以减少--batch_size或--samples_per_complex参数。降低--batch_size不会影响结果的质量,而是会增加运行时。
计算生成的结构的类似命令是:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
为了在Top-1预测的配体姿势上执行能量最小化,您可以使用--relax标志。
如果您知道自己的装订袋以及要弹性的侧链,则可以指定它们。在这种情况下,您也可以删除--keep_local_structures 。我们建议仅使用提供的模型,这些模型与柔性氨基酸至少在任何重配体原子的3.5a之内至少具有一个重原子。这样可以防止分布数据。
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
或者,如果您没有配体,也可以使用笑容表示。
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
但是,在这种情况下,您必须指定口袋中心和灵活的侧链。
如果您要处理多个复合物,请使用标志来查看data/protein_ligand_example_csv --protein_ligand_csv data/protein_ligand_example_csv.csv 。这使您可以一次指定多个复合物的推断,您可以在其中指定口袋中心和每个复合物的柔性侧链。
选择口袋中心时,最好确定靠近结合位点的氨基酸并计算其C-α坐标的平均值。在我们的培训中,我们选择了所有氨基酸,其中C-Alpha原子在配体的任何重原子的5a之内。
我们正在使用PDBBIND,您可以从许多来源下载。但是,我们还在此处提供了预处理的PDBBIND数据集,您可以通过unzip打开包装。所有蛋白质都与PDBFixer固定并对齐,以最大程度地减少口袋中的RMSD。将其解放到data/PDBBIND_atomCorrected 。
训练大分模型:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
模型权重保存在workdir目录中,可用于推断。
为了训练置信度模型,您可以使用先前训练的分数模型来生成样品。但是,我们选择训练另一个具有较高最大配体翻译的(较小)得分模型,以便置信模型看到更多的样本,并且可以更快地生成训练数据。
要训练这个具有较高最大翻译的较小分数模型,请运行以下内容:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
用于生成样品来训练置信模型的分数模型不必与推断期间与该置信度模型一起使用的分数模型相同。
通过运行以下操作来训练置信度模型:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
首先是--cache_creation_id 1然后--cache_creation_id 2等。最多4
现在,一切都经过训练,您可以使用新模型进行推断:)。
如果您在研究中使用diffdock口袋,请引用以下论文:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
感谢@jsilter使此代码更易于使用和创建HuggingFace接口!