DiffDock-Pocketは、ポケットレベルで動作する全原子ドッキングモデルです。サイドチェーンねじれの角度よりも拡散を実装し、計算的に生成された構造に最適化されています。このリポジトリには、モデルをトレーニングし、推論、視覚化を実行し、視覚化を実行し、論文で提示された数字を達成するために使用した重みを見つけることができます。このリポジトリはもともとDiffdockのフォークであるため、コマンドの一部は馴染みのあるように見えるかもしれませんが、2つのプログラムの互換性を期待できないほど多くの場所で拡張、適応、変更されています。注意して、これら2つのプログラムを混同しないでください!
推論を実行する簡単な方法が必要な場合は、Huggingfaceインターフェイスをこちらを使用できます。
このリポジトリに問題がある場合は、問題やPRを自由に作成してください!行動規範を検討してください。
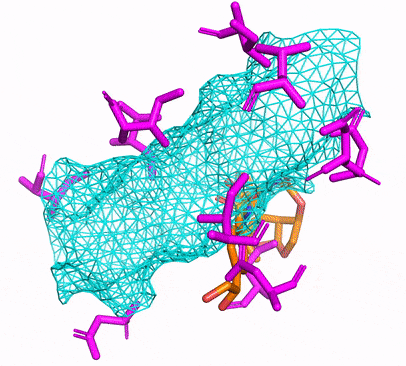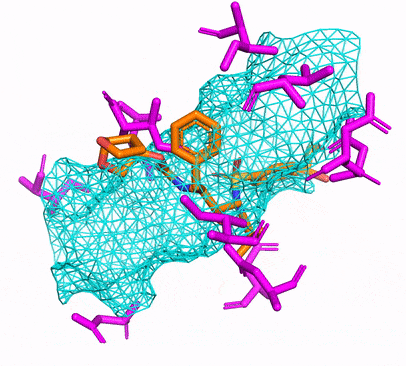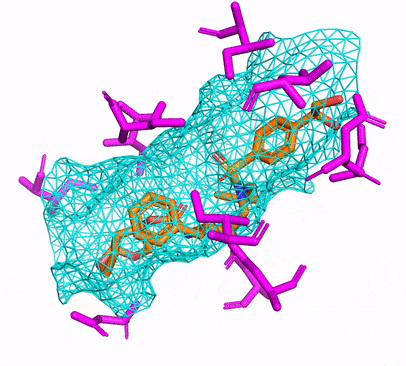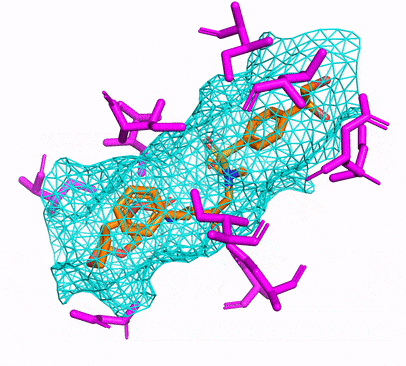
単に実行するだけで、CPUまたはCUDA Conda環境を作成できます
conda env create -f environment[-cpu].yml
CPUフラグを持つ、または存在しない。 CUDAバージョンは、はるかに高速であるため、推奨されます。
この最小限の例では、複雑な3dpfを使用して、柔軟なサイドケイヒンでリガンドを再ドックします。 40個のサンプルを描画し、自信モデルでランク付けします。すべての予測はresults/に保存されます。
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
ここでは、リガンドを変更しないように--keep_local_structuresフラグを指定しました。これにより、柔軟性をモデル化するポケットとどのサイドチェーンが自動的に決定されます。
このコマンドがメモリがなくなった場合、 --batch_sizeまたは--samples_per_complexパラメーターを減らすことができます。 --batch_sizeを下げることは、結果の品質に影響を与えませんが、ランタイムを増やします。
計算的に生成された構造の同様のコマンドは次のとおりです。
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
TOP-1予測リガンドポーズでエネルギー最小化を実行するには、 --relaxフラグを使用できます。
バインディングポケットと柔軟性のあるモデル化するサイドチェーンがわかっている場合は、それらを指定できます。この場合、 --keep_local_structuresをドロップすることもできます。私たちは、重いリガンド原子の3.5a以内に少なくとも1つの重原子を持つ柔軟なアミノ酸でのみ提供されたモデルを使用することをお勧めします。これにより、分散除外データが防止されます。
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
または、手元にリガンドがない場合は、笑顔の表現を使用することもできます。
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
ただし、この場合、ポケットセンターと柔軟なサイドチェーンを指定する必要があります。
複数の複合体を扱っている場合は、フラグを使用してdata/protein_ligand_example_csvをご覧ください--protein_ligand_csv data/protein_ligand_example_csv.csv 。これにより、複数の複合体の推論を一度に指定でき、各複合体のポケットセンターと柔軟なサイドチェーンを指定できます。
ポケットセンターを選択するときは、結合部位に近いアミノ酸を決定し、C-alpha座標の平均を計算するのが最善です。私たちのトレーニングでは、C-alpha原子がリガンドの重い原子の5a以内にあるすべてのアミノ酸を選択しました。
多くのソースからダウンロードできるpdbbindを使用しています。ただし、ここでは以前に処理されたPDBBINDデータセットも提供しており、 unzipで開梱できます。すべてのタンパク質はPDBFixerで固定されており、ポケット内のRMSDを最小限に抑えるために整列しています。 data/PDBBIND_atomCorrectedに解除します。
大きなスコアモデルをトレーニングします:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
モデルの重みはworkdirディレクトリに保存され、推論に使用できます。
信頼モデルをトレーニングするために、以前にトレーニングしたスコアモデルを使用してサンプルを生成できます。ただし、信頼モデルではより多様なサンプルを見て、トレーニングデータをより迅速に生成できるように、最大リガンド翻訳で別の(小さい)スコアモデルをトレーニングすることを選択しました。
より高い最大翻訳でこの小さなスコアモデルをトレーニングするには、以下を実行します。
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
信頼モデルをトレーニングするためにサンプルを生成するために使用されるスコアモデルは、推論中にその信頼モデルで使用されるスコアモデルと同じである必要はありません。
以下を実行して自信モデルをトレーニングします。
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
最初は--cache_creation_id 1 then --cache_creation_id 2など
これですべてが訓練されており、新しいモデルで推論を実行できます:)。
あなたの研究でdiffdockポケットを使用する場合は、次の論文を引用してください。
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
このコードを使いやすくしてくれて、Huggingfaceインターフェイスを作成してくれた@jsilterに感謝します!