Diffdock-Pocket هو نموذج إرساء بالكامل يعمل على مستوى الجيب. وهو ينفذ الانتشار على زوايا تواء السلسلة الجانبية ويتم تحسينه للهياكل التي تم إنشاؤها حسابية. في هذا المستودع ، ستجد الكود لتدريب نموذج ، وتشغيل الاستدلال ، والتصورات ، والأوزان التي استخدمناها لتحقيق الأرقام المقدمة في الورقة. هذا المستودع هو في الأصل شوكة من diffdock ، لذلك قد تبدو بعض الأوامر مألوفة - ولكن تم تمديدها وتكييفها وتغييرها في العديد من الأماكن التي لا يمكنك توقع أي موافقة للبرامجين. كن على دراية ، ولا تقم بخلط هذين البرنامجين!
إذا كنت تريد طريقة سهلة لتشغيل الاستدلال ، فيمكنك استخدام واجهة Huggingface هنا.
لا تتردد في إنشاء أي مشكلات ، أو PRS إذا كان لديك أي مشاكل مع هذا المستودع! يرجى النظر في مدونة السلوك.
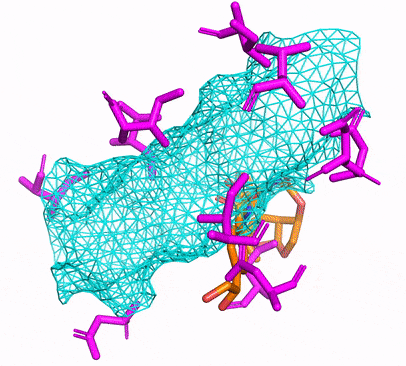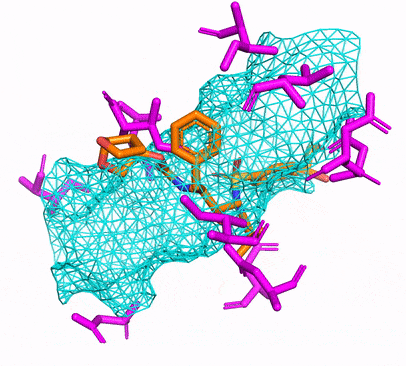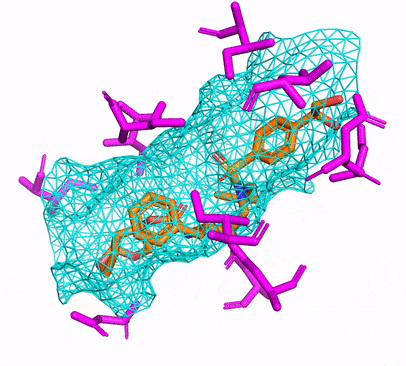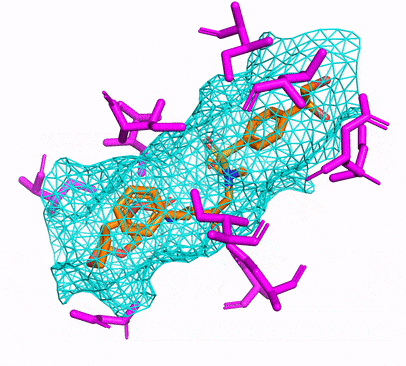
يمكنك إما إنشاء بيئة CPU أو CUDA Conda عن طريق التشغيل ببساطة
conda env create -f environment[-cpu].yml
مع ، أو بدون علم وحدة المعالجة المركزية. ينصح إصدار CUDA ، لأنه أسرع بكثير.
في هذا المثال الأدنى ، نستخدم 3dpf المعقدة ونعيد صرف يجند مع cahins الجانب المرن. نرسم 40 عينة ، ونرسلها مع نموذج الثقة. سيتم تخزين جميع التنبؤات في results/ .
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
هنا ، حددنا علامة --keep_local_structures لعدم تعديل يجند. مع هذا ، يتم تحديد الجيب وأي سلاسل جانبية للنموذج المرنة تلقائيًا.
إذا كان هذا الأمر ينفد من الذاكرة ، فيمكنك تقليل المعلمة --batch_size أو --samples_per_complex . لن يؤثر التخفيض --batch_size على جودة النتائج ، ولكنه سيزيد من وقت التشغيل.
أمر مشابه للهياكل التي تم إنشاؤها حسابية هي:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
لأداء تقليل الطاقة على أفضل ليجند المتوقع ، يمكنك استخدام علامة- --relax .
إذا كنت تعرف جيبك الملزم وأي السلاسل الجانبية التي يجب تصميمها على أنها مرنة ، فيمكنك تحديدها. في هذه الحالة ، يمكنك أيضًا إسقاط --keep_local_structures . ننصح باستخدام نموذجنا المقدم فقط مع الأحماض الأمينية المرنة التي لديها ذرة ثقيلة واحدة على الأقل ضمن 3.5A من أي ذرة يجند الثقيلة. هذا يمنع بيانات التوزيع.
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
أو إذا لم يكن لديك يجند في متناول اليد ، فيمكنك أيضًا استخدام تمثيل الابتسامات.
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
ومع ذلك ، في هذه الحالة ، يجب عليك تحديد مركز جيبك وسلاسل جانبية مرنة.
إذا كنت تتعامل مع مجمعات متعددة ، فقم بإلقاء نظرة على data/protein_ligand_example_csv مع Flag --protein_ligand_csv data/protein_ligand_example_csv.csv . يتيح لك ذلك تحديد الاستدلال على مجمعات متعددة في وقت واحد ، حيث يمكنك تحديد مركز الجيب والسلاسل الجانبية المرنة لكل مجمع.
عند اختيار مركز الجيب ، من الأفضل تحديد الأحماض الأمينية القريبة من موقع الربط وحساب متوسط إحداثيات C-alpha. في تدريبنا ، اخترنا جميع الأحماض الأمينية حيث تقع ذرة C-alpha ضمن 5A من أي ذرة ثقيلة من يجند.
نحن نستخدم PDBBIND الذي يمكنك تنزيله من العديد من المصادر. ومع ذلك ، فإننا نقدم أيضًا مجموعة بيانات PDBBIND التي تم تجهيزها مسبقًا هنا والتي يمكنك إلغاء unzip بها. تم إصلاح جميع البروتينات باستخدام PDBFixer ومحاذاة لتقليل RMSD في الجيب. قم بفكها على data/PDBBIND_atomCorrected .
تدريب نموذج الدرجات الكبيرة:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
يتم حفظ الأوزان النموذجية في دليل workdir ويمكن استخدامها للاستدلال.
لتدريب نموذج الثقة ، يمكنك استخدام نموذج الدرجات المدربة مسبقًا لإنشاء العينات. ومع ذلك ، اخترنا تدريب نموذج آخر (أصغر) من الدرجات مع ترجمة أقصى ليجند أعلى ، بحيث يرى نموذج الثقة عينات أكثر تنوعًا ويمكن إنتاج بيانات التدريب بسرعة أكبر.
لتدريب نموذج الدرجات الأصغر هذا بترجمة أقصى أعلى ، قم بتشغيل ما يلي:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
لا يجب أن يكون نموذج النتيجة المستخدم لإنشاء العينات لتدريب نموذج الثقة هو نفسه نموذج الدرجات المستخدم مع نموذج الثقة هذا أثناء الاستدلال.
تدريب نموذج الثقة من خلال تشغيل ما يلي:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
أولاً مع --cache_creation_id 1 ثم --cache_creation_id 2 إلخ.
الآن تم تدريب كل شيء ويمكنك تشغيل الاستدلال مع نموذجك الجديد :).
إذا كنت تستخدم Diffdock-Pocket في بحثك ، فيرجى الاستشهاد بالورقة التالية:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
شكرًا لك @JSilter لجعل هذا الرمز أسهل في الاستخدام وإنشاء واجهة HuggingFace!