Diffdock-карман-это модель стыковки все-атома, работающая на уровне кармана. Он реализует диффузию по углам кручения боковой цепи и оптимизируется для вычислительно сгенерированных структур. В этом репозитории вы найдете код для обучения модели, выполнения вывода, визуализаций и весов, которые мы использовали для достижения чисел, представленных в статье. Этот репозиторий изначально представляет собой вилку от Diffdock, поэтому некоторые команды могут показаться знакомыми, но она была расширена, адаптирована и изменена во многих местах, что вы не можете ожидать какой -либо совместимости двух программ. Имейте в виду, и не смешивайте эти две программы!
Если вы хотите простой способ сделать вывод, вы можете использовать здесь интерфейс HuggingFace.
Не стесняйтесь создавать какие -либо проблемы или PRS, если у вас есть какие -либо проблемы с этим репозиторием! Пожалуйста, рассмотрите кодекс поведения.
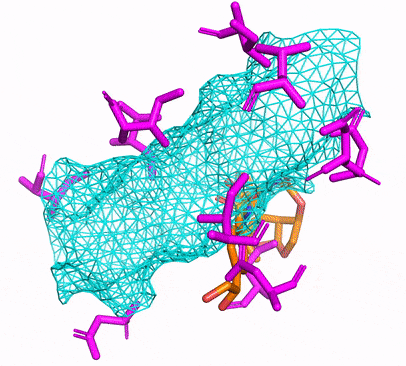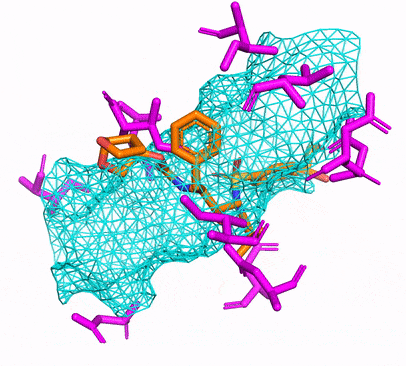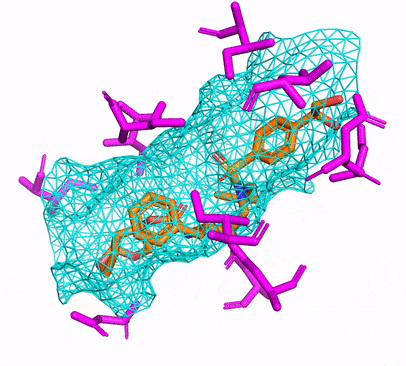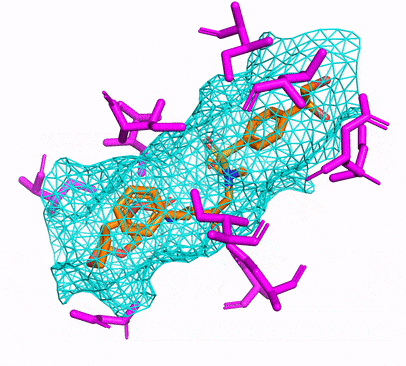
Вы можете создать среду CPU или Cuda Conda, просто запустив
conda env create -f environment[-cpu].yml
с или без флага процессора. Рекомендуется версия CUDA, так как она намного быстрее.
В этом минимальном примере мы используем сложный 3dpf и переоцените лиганд с гибкой стороной кахинс. Мы рисуем 40 образцов и оцениваем их с помощью модели доверия. Все прогнозы будут храниться в results/ .
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
Здесь мы определили флаг --keep_local_structures , чтобы не изменить лиганд. При этом карман и какие боковые цепи моделируют гибкость, автоматически определяется.
Если эта команда заканчивает память, вы можете уменьшить параметр --batch_size или параметр --samples_per_complex . Снижение --batch_size не повлияет на качество результатов, но увеличит время выполнения.
Аналогичная команда для вычислительно сгенерированных структур была бы:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
Чтобы выполнить минимизацию энергии на прогнозируемой позе TOP-1, вы можете использовать флаг --relax .
Если вы знаете свой привязывающий карман и какие боковые цепи моделировать как гибкие, вы можете указать их. В этом случае вы также можете отказаться от --keep_local_structures . Мы советуем использовать нашу предоставленную модель только с гибкими аминокислотами, которые имеют как минимум один тяжелый атом в пределах 3,5А любого атома тяжелого лиганда. Это предотвращает данные об ограничении.
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
Или, если у вас нет лиганда под рукой, вы также можете использовать представление Smiles.
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
Однако в этом случае вам нужно указать свой карманный центр и гибкие боковые цепи.
Если вы имеете дело с несколькими комплексами, посмотрите на data/protein_ligand_example_csv с флага --protein_ligand_csv data/protein_ligand_example_csv.csv . Это позволяет вам определить вывод на несколько комплексов одновременно, где вы можете указать центр кармана и гибкие боковые цепи для каждого комплекса.
При выборе карманного центра лучше всего определить аминокислоты, которые близки к сайту связывания, и вычислить среднее значение их координат C-альфа. В нашей тренировке мы выбрали все аминокислоты, где их атом C-альфа находится в пределах 5А от любого тяжелого атома лиганда.
Мы используем PDBBIND, который вы можете скачать из многих источников. Тем не менее, мы также предоставляем предварительно обработанный набор данных PDBBind, который вы можете распаковать с помощью unzip . Все белки были зафиксированы с помощью PDBFixer и выровнены, чтобы минимизировать RMSD в кармане. Распаковать его до data/PDBBIND_atomCorrected .
Обучить большую модель баллов:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
Веса модели сохраняются в каталоге workdir и могут использоваться для вывода.
Чтобы обучить модель доверия, вы можете использовать ранее обученную модель баллов для генерации образцов. Тем не менее, мы решили обучить другую (меньшую) модель баллов с более высоким максимальным переводом лиганда, чтобы модель доверия видит более разнообразные образцы, и данные обучения могут быть получены быстрее.
Чтобы обучить эту модель меньшего балла с более высоким максимальным переводом, запустите следующее:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
Модель баллов, используемая для генерации образцов для обучения модели доверия, не должна быть такой же, как модель баллов, которая используется с этой моделью доверия во время вывода.
Обучить модель доверия, выполнив следующее:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
Сначала с помощью --cache_creation_id 1 затем --cache_creation_id 2 и т. д. до 4
Теперь все обучено, и вы можете выполнить вывод с новой моделью :).
Если вы используете Diffdock Cocket в своем исследовании, пожалуйста, укажите следующую статью:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
Спасибо @jsilter за то, что упростили этот код в использовании и за создание интерфейса Huggingface!