Diffdock-Pocket é um modelo de ancoragem em todos os átomos que opera no nível do bolso. Ele implementa a difusão sobre os ângulos de torção da cadeia lateral e é otimizada para estruturas geradas computacionalmente. Neste repositório, você encontrará o código para treinar um modelo, executar inferência, visualizações e os pesos que usamos para alcançar os números apresentados no papel. Esse repositório é originalmente um garfo do Diffdock; portanto, alguns dos comandos podem parecer familiares - mas foi estendido, adaptado e alterado em tantos lugares que você não pode esperar de qualquer compatibilidade dos dois programas. Esteja ciente e não misture esses dois programas!
Se você deseja uma maneira fácil de executar a inferência, pode usar a interface Huggingface aqui.
Sinta -se à vontade para criar problemas ou PRs se tiver algum problema com este repositório! Por favor, considere o código de conduta.
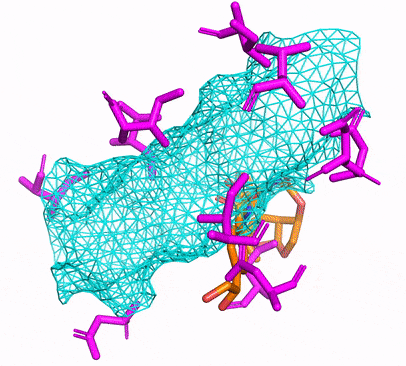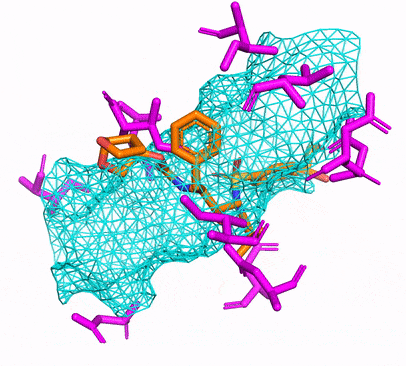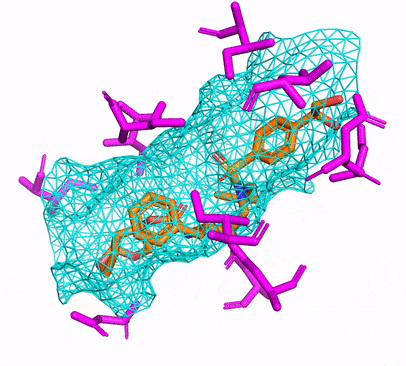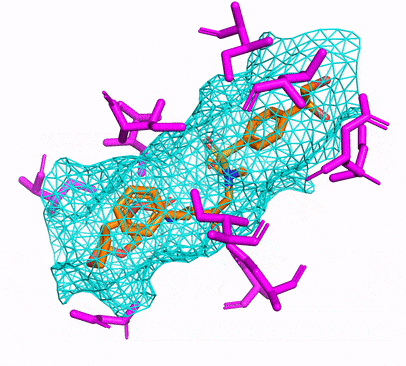
Você pode criar o ambiente CPU ou CUDA CONDA, simplesmente executando
conda env create -f environment[-cpu].yml
com, ou sem a bandeira da CPU. A versão CUDA é recomendada, pois é muito mais rápida.
Neste exemplo mínimo, usamos o complexo 3dpf e renove o ligante com Cahins laterais flexíveis. Desenhamos 40 amostras e as classificamos com o modelo de confiança. Todas as previsões serão armazenadas nos results/ .
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
Aqui, especificamos o sinalizador --keep_local_structures para não modificar o ligante. Com isso, o bolso e quais correntes laterais para modelar flexíveis são determinadas automaticamente.
Se esse comando for sem memória, você poderá diminuir o parâmetro --batch_size ou --samples_per_complex . A redução --batch_size não afetará a qualidade dos resultados, mas aumentará o tempo de execução.
Um comando semelhante para estruturas geradas computacionalmente seria:
python inference.py --protein_path example_data/3dpf_protein_esm.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --keep_local_structures --save_visualisation
Para realizar a minimização de energia na pose de ligante prevista-1 top-1, você pode usar o sinalizador --relax .
Se você conhece o seu bolso de ligação e quais cadeias laterais modelarem como flexíveis, pode especificá -las. Nesse caso, você também pode cair --keep_local_structures . Aconselhamos usar nosso modelo fornecido apenas com aminoácidos flexíveis que possuem pelo menos um átomo pesado em 3,5a de qualquer átomo de ligante pesado. Isso impede dados fora da distribuição.
python inference.py --protein_path example_data/3dpf_protein.pdb --ligand example_data/3dpf_ligand.sdf --batch_size 20 --samples_per_complex 40 --save_visualisation --pocket_center_x 9.7742 --pocket_center_y 27.2863 --pocket_center_z 14.6573 --flexible_sidechains A:160-A:193-A:197-A:198-A:222-A:224-A:227
Ou se você não tiver um ligante em mãos, também poderá usar a representação do Smiles.
--ligand [H]/N=C1/C(=O)C(=O)[C@@]1([H])[N@@+]1([H])C([H])([H])c2c([H])c(C([H])([H])N([H])C(=O)c3nc4sc5c(c4c(=O)n3[H])C([H])([H])C([H])([H])S(=O)(=O)C5([H])[H])c([H])c([H])c2C([H])([H])C1([H])[H]
No entanto, neste caso, você deve especificar seu centro de bolso e cadeias laterais flexíveis.
Se você estiver lidando com vários complexos, dê uma olhada no data/protein_ligand_example_csv com o FLAG --protein_ligand_csv data/protein_ligand_example_csv.csv . Isso permite especificar a inferência em vários complexos de uma só vez, onde você pode especificar o centro de bolso e as cadeias laterais flexíveis para cada complexo.
Ao escolher o centro de bolso, é melhor determinar os aminoácidos próximos ao local de ligação e calcular a média de suas coordenadas C-alfa. Em nosso treinamento, escolhemos todos os aminoácidos onde seu átomo de C-alfa fica a 5a de qualquer átomo pesado do ligante.
Estamos usando o PDBBind, que você pode baixar de várias fontes. No entanto, também fornecemos um conjunto de dados PDBBind pré-processado aqui, que você pode descompactar com unzip . Todas as proteínas foram fixadas com PDBFixer e alinhadas para minimizar o RMSD no bolso. Desembale -o para data/PDBBIND_atomCorrected .
Treine o modelo de grande pontuação:
python -m train --run_name big_score_model --test_sigma_intervals --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 5 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 60 --nv 10 --num_conv_layers 6 --distance_embed_dim 64 --cross_distance_embed_dim 64 --sigma_embed_dim 64 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 8 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 750 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
Os pesos do modelo são salvos no diretório workdir e podem ser usados para inferência.
Para treinar o modelo de confiança, você pode usar o modelo de pontuação anteriormente treinado para gerar as amostras. No entanto, optamos por treinar outro modelo de pontuação (menor) com uma tradução máxima de ligante máximo, para que o modelo de confiança veja amostras mais diversas e os dados de treinamento possam ser produzidos mais rapidamente.
Para treinar esse modelo de pontuação menor com maior tradução máxima, execute o seguinte:
python -m train --run_name small_score_model --test_sigma_intervals --esm_embeddings_path data/esm2_3billion_embeddings.pt --cache_path data/cache --log_dir workdir --lr 1e-3 --tr_sigma_min 0.1 --tr_sigma_max 15 --rot_sigma_min 0.03 --rot_sigma_max 1.55 --tor_sigma_min 0.03 --sidechain_tor_sigma_min 0.03 --batch_size 16 --ns 32 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scheduler plateau --scale_by_sigma --dropout 0.1 --sampling_alpha 1 --sampling_beta 1 --remove_hs --c_alpha_max_neighbors 24 --atom_max_neighbors 12 --receptor_radius 15 --num_dataloader_workers 1 --cudnn_benchmark --rot_alpha 1 --rot_beta 1 --tor_alpha 1 --tor_beta 1 --val_inference_freq 5 --use_ema --scheduler_patience 30 --n_epochs 500 --all_atom --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --pocket_reduction --pocket_buffer 10 --flexible_sidechains --flexdist 3.5 --flexdist_distance_metric prism --protein_file protein_esmfold_aligned_tr_fix --compare_true_protein --conformer_match_sidechains --conformer_match_score exp --match_max_rmsd 2 --use_original_conformer_fallback --use_original_conformer
O modelo de pontuação usado para gerar as amostras para treinar o modelo de confiança não precisa ser o mesmo que o modelo de pontuação usado com esse modelo de confiança durante a inferência.
Treine o modelo de confiança executando o seguinte:
python -m filtering.filtering_train --run_name confidence_model --original_model_dir workdir/small_score_model --ckpt best_ema_inference_epoch_model.pt --inference_steps 20 --samples_per_complex 7 --batch_size 16 --n_epochs 100 --lr 3e-4 --scheduler_patience 50 --ns 24 --nv 6 --num_conv_layers 5 --dynamic_max_cross --scale_by_sigma --dropout 0.1 --all_atoms --sh_lmax 1 --split_train data/splits/timesplit_no_lig_overlap_train --split_val data/splits/timesplit_no_lig_overlap_val_aligned --log_dir workdir --cache_path .cache/data_filtering data/PDBBIND_atomCorrected --remove_hs --c_alpha_max_neighbors 24 --receptor_radius 15 --esm_embeddings_path data/esm2_3billion_embeddings.pt --main_metric loss --main_metric_goal min --best_model_save_frequency 5 --rmsd_classification_cutoff 2 --sc_rmsd_classification_cutoff 1 --protein_file protein_esmfold_aligned_tr_fix --use_original_model_cache --pocket_reduction --pocket_buffer 10 --cache_creation_id 1 --cache_ids_to_combine 1 2 3 4
Primeiro com --cache_creation_id 1 Então --cache_creation_id 2 etc. até 4
Agora tudo está treinado e você pode obter inferência com seu novo modelo :).
Se você usar o Diffdock-Pocket em sua pesquisa, cite o seguinte artigo:
@article{plainer2023diffdockpocket,
author = {Plainer, Michael and Toth, Marcella and Dobers, Simon and St{"a}rk, Hannes and Corso, Gabriele and Marquet, C{'e}line and Barzilay, Regina},
title = {{DiffDock-Pocket}: Diffusion for Pocket-Level Docking with Sidechain Flexibility},
year = {2023},
maintitle = {Advances in Neural Information Processing Systems},
booktitle = {Machine Learning in Structural Biology},
}
Obrigado @jsilter por facilitar o uso deste código e por criar a interface Huggingface!