Observação
Bem-vindo ao nosso trabalho mais recente: Carellama (CARE ALPACA), um modelo médico de grande idioma que combina dezenas de conjuntos de dados médicos de ajuste médicos disponíveis e modelos de idiomas médicos disponíveis abertamente para promover o desenvolvimento rápido do Medical LLM: https://github.com/wangongsheng/carellama
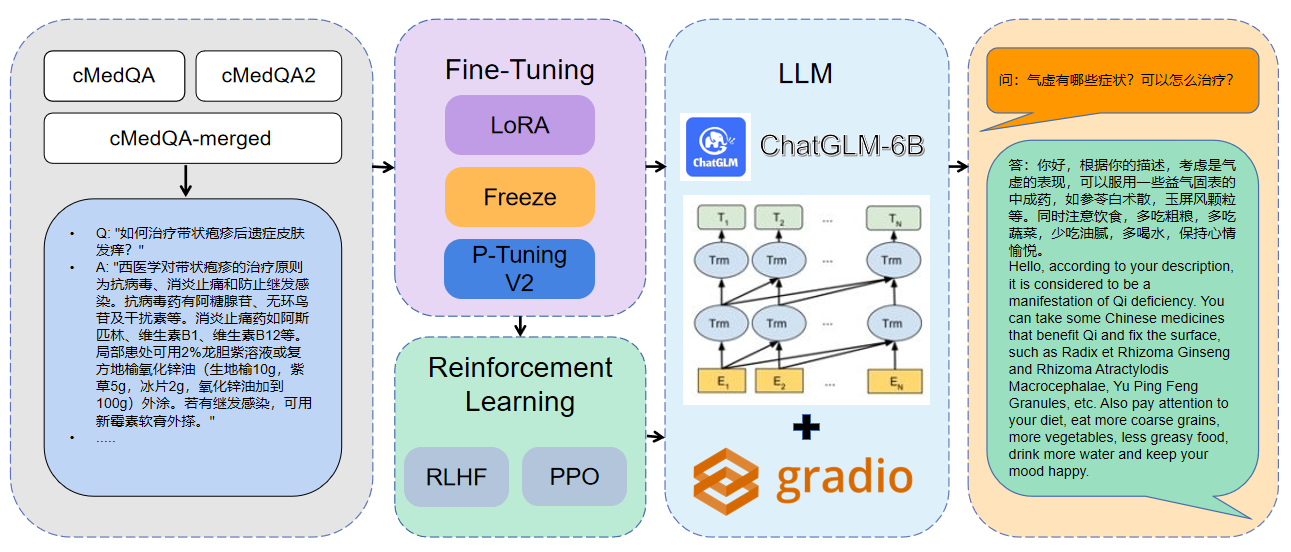
1 Os dados utilizados são cmedqa2
| projeto | Conjunto de dados | Modelo base |
|---|---|---|
| Chatmed | Consult contém 50w + consulta on -line + resposta chatgpt, TCM Tradition Chinese Medicine Diagnóstico e conjunto de dados de tratamento não foram publicados | Llama-7b |
| ChatDoctor | HealthCaremagic-100k contém 100k+ conjunto de dados de diálogo de pacientes com paciente real, o icliniq-10k contém 10K+ conjunto de dados de diálogo do paciente-doutor | Llama-7b |
| Med-Chatglm | Huatuo-Data, Huatuo-Liver-Câncer | Chatglm-6b |
| Huatuo-llama-Med-Chinese | Huatuo-Data, Huatuo-Liver-Câncer | Llama-7b |
| Doctorglm | CMD., Meddialog, DataSet de projeto de chatdoctor | Chatglm-6b |
| MedicalGpt-Zh | Dados não de código aberto | Chatglm-6b |
| Dr.llama | Lhama | |
| Medical_NLP 2 | - | - |
| CMCQA 3 | - | - |
| Qizhengpt | - | - |
| LLM-pré-fino-finente | - | - |
| PMC-Llama | - | Llama-7b |
| Bianque | - | - |
| Medalpaca | - | Llama-7b |
| MedicalGpt | - | - |
| LLM-pré-fino-finente | - | - |
| Shennong-tcm-llm | - | - |
| Sunsimiao | - | - |
| Cmlm-Zhongjing | - | - |
| Zhongjing | - | - |
| Ming | - | - |
| Medllm de disco | - | - |
pip install - r requirements . txt CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / finetune . py
- - do_train
- - dataset merged - cMedQA
- - finetuning_type lora
- - output_dir . / med - lora
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16 CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / finetune . py
- - do_train
- - dataset merged - cMedQA
- - finetuning_type freeze
- - output_dir . / med - freeze
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16 CUDA_VISIBLE_DEVICES = 1 python MedQA - ChatGLM / finetune . py
- - do_train - - dataset merged - cMedQA
- - finetuning_type p_tuning
- - output_dir . / med - p_tuning
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16Para obter mais informações sobre parâmetros, você pode visualizar a explicação detalhada dos documentos/parâmetros.md.
Treinamento distribuído de Multi-GPU:
# 配置分布式参数
accelerate config
# 分布式训练
accelerate launch src / finetune . py
- - do_train
- - dataset Huatuo , CMD , MedDialog , guanaco , cognition
- - finetuning_type lora
- - output_dir med - lora
- - per_device_train_batch_size 16
- - gradient_accumulation_steps 4
- - lr_scheduler_type cosine
- - logging_steps 10
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 3.0
- - fp16
- - ddp_find_unused_parameters False # 分布式训练时,LoRA微调需要添加防止报错
- - plot_loss CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / web_demo . py
- - checkpoint_dir med - lora /
( med - freez / )
( med - p_tuning / ) CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / infer . py
- - checkpoint_dir med - lora /
( med - freez / )
( med - p_tuning / )Mesclar o modelo:
CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / export_weights . py
- - finetuning_weights_path . / med - lora
- - save_weights_path . / save_loraCarregando o modelo mesclado:
CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / load_export_weights . py
- - save_weights_path . / save_lora| Método de ajuste fino | Peso do modelo | Duração do treinamento | Rodadas de treinamento |
|---|---|---|---|
| Lora | Medqa-chatglm-Lora | 28h | 10 |
| P2 de ajuste P. | MedQA-chatglm-ptuningv2 | 27h | 10 |
| Congelar | MedQA-ChatGLM-Freeze | 28h | 10 |
* O experimento foi realizado no sistema Linux, A100 (1x, 80 GB)
Os recursos relacionados a este projeto são apenas para pesquisa acadêmica e são estritamente proibidos para fins comerciais. Ao usar peças envolvendo código de terceiros, siga estritamente o protocolo de código aberto correspondente. O conteúdo gerado pelo modelo é afetado por fatores como cálculo do modelo, aleatoriedade e perdas de precisão quantitativa, e este projeto não pode garantir sua precisão. A maioria dos conjuntos de dados deste projeto é gerada por modelos e não pode ser usada como base para o diagnóstico médico real, mesmo que eles cumpram certos fatos médicos. Este projeto não assume nenhuma responsabilidade legal por qualquer saída de conteúdo pelo modelo, nem é responsável por quaisquer perdas que possam surgir do uso de recursos relevantes e resultados de saída.