Notiz
Willkommen zu unserer neuesten Arbeit: Carellama (Care Alpaca), ein medizinisches Großsprachmodell, das Dutzende von öffentlich verfügbaren medizinischen Finanz-Tuning-Datensätzen und offen verfügbare medizinische Großsprachenmodelle kombiniert, um die schnelle Entwicklung von medizinischen LLM zu fördern
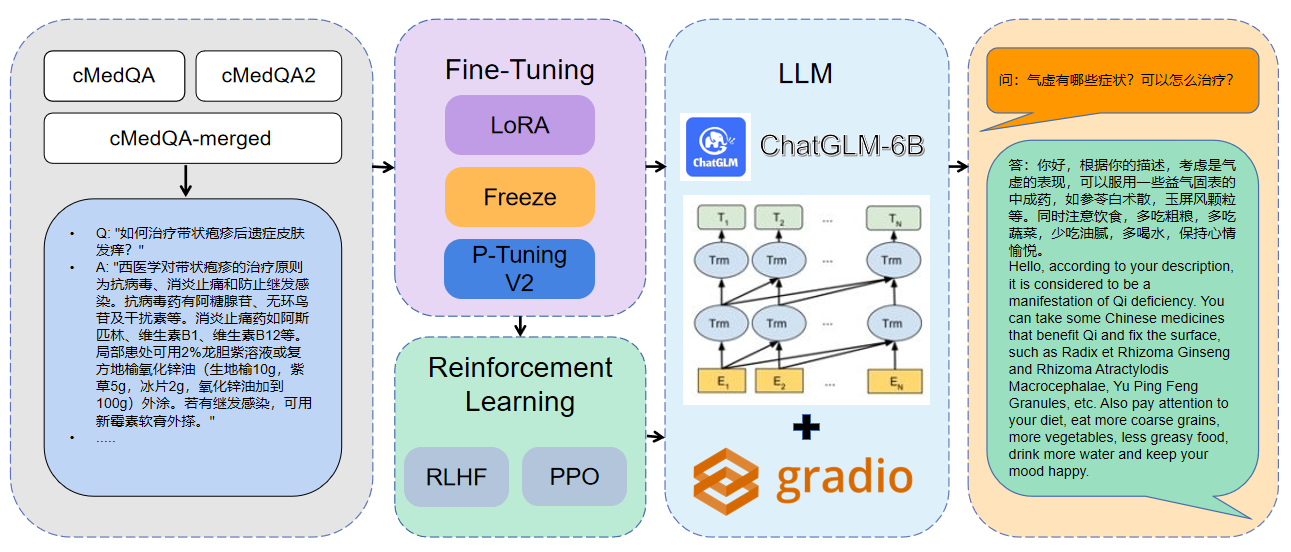
1 Die verwendeten Daten sind CMEDQA2
| Projekt | Datensatz | Basismodell |
|---|---|---|
| ChatMed | Consult enthält 50W + Online -Beratung + CHATGPT -Antwort, TCM Traditionelle chinesische Medizin -Diagnose- und Behandlungsdatensatz wurde nicht veröffentlicht | Lama-7b |
| Chatdoctor | HealthCaremagic-100K enthält 100K+ Real Patient-Doctor-Dialogdatensatz, ICLINIQ-10K enthält einen Dialogdialog-Datensatz von 10K+ Patienten-Doktor, GenMedGPT-5K enthält 5k+ Doktor-Patient-Dialog-Datensatz, das von GPT generiert wurde | Lama-7b |
| Med-chatglm | Huatuo-data, Huatuo-Liver-Krebs | Chatglm-6b |
| Huatuo-Llama-Med-Chinese | Huatuo-data, Huatuo-Liver-Krebs | Lama-7b |
| Doktorglm | Cmd., MedDialog, ChatDoctor -Projektdatensatz | Chatglm-6b |
| MedicalGpt-Zh | Daten nicht Open Source | Chatglm-6b |
| Dr.llama | Lama | |
| Medical_NLP 2 | - - | - - |
| CMCQA 3 | - - | - - |
| Qizhengpt | - - | - - |
| LLM-Vorstrain-Finetune | - - | - - |
| PMC-Llama | - - | Lama-7b |
| Bianque | - - | - - |
| Medaille | - - | Lama-7b |
| MedicalGpt | - - | - - |
| LLM-Vorstrain-Finetune | - - | - - |
| Shennong-tcm-llm | - - | - - |
| Sunsimiao | - - | - - |
| CMLM-Zhongjing | - - | - - |
| Zhongjing | - - | - - |
| Ming | - - | - - |
| Disc-Medllm | - - | - - |
pip install - r requirements . txt CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / finetune . py
- - do_train
- - dataset merged - cMedQA
- - finetuning_type lora
- - output_dir . / med - lora
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16 CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / finetune . py
- - do_train
- - dataset merged - cMedQA
- - finetuning_type freeze
- - output_dir . / med - freeze
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16 CUDA_VISIBLE_DEVICES = 1 python MedQA - ChatGLM / finetune . py
- - do_train - - dataset merged - cMedQA
- - finetuning_type p_tuning
- - output_dir . / med - p_tuning
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16Für weitere Parameterinformationen können Sie die detaillierte Erläuterung von DOCs/Parametern anzeigen.
Multi-GPU-verteiltes Training:
# 配置分布式参数
accelerate config
# 分布式训练
accelerate launch src / finetune . py
- - do_train
- - dataset Huatuo , CMD , MedDialog , guanaco , cognition
- - finetuning_type lora
- - output_dir med - lora
- - per_device_train_batch_size 16
- - gradient_accumulation_steps 4
- - lr_scheduler_type cosine
- - logging_steps 10
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 3.0
- - fp16
- - ddp_find_unused_parameters False # 分布式训练时,LoRA微调需要添加防止报错
- - plot_loss CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / web_demo . py
- - checkpoint_dir med - lora /
( med - freez / )
( med - p_tuning / ) CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / infer . py
- - checkpoint_dir med - lora /
( med - freez / )
( med - p_tuning / )Das Modell zusammenführen:
CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / export_weights . py
- - finetuning_weights_path . / med - lora
- - save_weights_path . / save_loraLaden des fusionierten Modells:
CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / load_export_weights . py
- - save_weights_path . / save_lora| Feinabstimmungsmethode | Modellgewicht | Trainingsdauer | Trainingsrunden |
|---|---|---|---|
| Lora | Medqa-chatglm-lora | 28h | 10 |
| P-Tuning v2 | Medqa-chatglm-ptuningv2 | 27h | 10 |
| Einfrieren | Medqa-Chatglm-Einfrieren | 28h | 10 |
* Das Experiment wurde am Linux -System A100 (1x, 80 GB) durchgeführt
Die Ressourcen im Zusammenhang mit diesem Projekt dienen nur für die akademische Forschung und sind für kommerzielle Zwecke strengstens untersagt. Wenn Sie Teile mit Code von Drittanbietern verwenden, folgen Sie bitte dem entsprechenden Open-Source-Protokoll ausschließlich. Der vom Modell erzeugte Inhalt wird von Faktoren wie Modellberechnung, Zufälligkeit und quantitativen Genauigkeitsverlusten beeinflusst, und dieses Projekt kann seine Genauigkeit nicht garantieren. Die meisten Datensätze dieses Projekts werden von Modellen generiert und können nicht als Grundlage für die tatsächliche medizinische Diagnose verwendet werden, selbst wenn sie bestimmten medizinischen Fakten entsprechen. Dieses Projekt setzt keine gesetzliche Haftung für eine Inhaltsausgabe durch das Modell über und haftet auch nicht für Verluste, die sich aus der Verwendung relevanter Ressourcen und Ausgabeergebnisse ergeben können.