Note
Bienvenue dans nos derniers travaux: Carellama (Care Alpaca), un modèle médical de grande langue qui combine des dizaines d'ensembles de données d'adaptation médicale accessibles au public et de modèles de grande langue médicale ouvertement disponibles pour promouvoir le développement rapide de Medical LLM: https://github.com/wangrongsheng/carellama
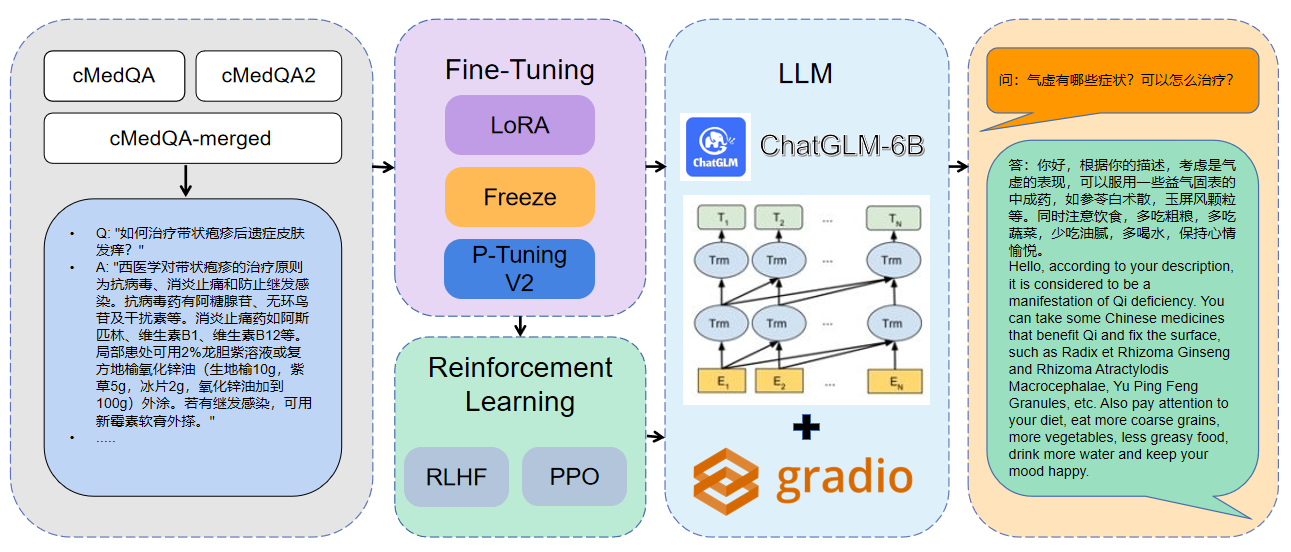
1 Les données utilisées sont CMEDQA2
| projet | Ensemble de données | Modèle de base |
|---|---|---|
| ChatMed | CONSULTES CONTANT 50W + CONSULTATION EN LIGNE + RÉPONSE DE CHATGPT, L'ensemble de données de diagnostic et de traitement traditionnel de médecine chinoise TCM n'a pas été publié | Lama-7b |
| Chatte | Ensemble de données sur le dialogue de dialogue Patient-Docaremagic-100K contient 100k + | Lama-7b |
| Chat-chat | Huatuo-Data, Huatuo-Liver-Cancer | Chatglm-6b |
| Huatuo-lelama-med-chinois | Huatuo-Data, Huatuo-Liver-Cancer | Lama-7b |
| Docteur | Cmd., MedDialog, ChatDoctor Project Dataset | Chatglm-6b |
| Medicalgpt-zh | Données non open source | Chatglm-6b |
| Dr.Lama | Lama | |
| Medical_nlp 2 | - | - |
| CMCQA 3 | - | - |
| Qizhengpt | - | - |
| LLM-pré-Finetune | - | - |
| PMC-Llama | - | Lama-7b |
| Bianque | - | - |
| médalpaca | - | Lama-7b |
| Médical | - | - |
| LLM-pré-Finetune | - | - |
| Shennong-TCM-llm | - | - |
| Soleil | - | - |
| Cmlm-zhongjing | - | - |
| Zhongjing | - | - |
| Ming | - | - |
| Disque-médllm | - | - |
pip install - r requirements . txt CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / finetune . py
- - do_train
- - dataset merged - cMedQA
- - finetuning_type lora
- - output_dir . / med - lora
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16 CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / finetune . py
- - do_train
- - dataset merged - cMedQA
- - finetuning_type freeze
- - output_dir . / med - freeze
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16 CUDA_VISIBLE_DEVICES = 1 python MedQA - ChatGLM / finetune . py
- - do_train - - dataset merged - cMedQA
- - finetuning_type p_tuning
- - output_dir . / med - p_tuning
- - per_device_train_batch_size 32
- - gradient_accumulation_steps 256
- - lr_scheduler_type cosine
- - logging_steps 500
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 10.0
- - fp16Pour plus d'informations sur les paramètres, vous pouvez afficher l'explication détaillée de Docs / Paramètres.md.
Formation distribuée multi-GPU:
# 配置分布式参数
accelerate config
# 分布式训练
accelerate launch src / finetune . py
- - do_train
- - dataset Huatuo , CMD , MedDialog , guanaco , cognition
- - finetuning_type lora
- - output_dir med - lora
- - per_device_train_batch_size 16
- - gradient_accumulation_steps 4
- - lr_scheduler_type cosine
- - logging_steps 10
- - save_steps 1000
- - learning_rate 5e-5
- - num_train_epochs 3.0
- - fp16
- - ddp_find_unused_parameters False # 分布式训练时,LoRA微调需要添加防止报错
- - plot_loss CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / web_demo . py
- - checkpoint_dir med - lora /
( med - freez / )
( med - p_tuning / ) CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / infer . py
- - checkpoint_dir med - lora /
( med - freez / )
( med - p_tuning / )Fusionner le modèle:
CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / export_weights . py
- - finetuning_weights_path . / med - lora
- - save_weights_path . / save_loraChargement du modèle fusionné:
CUDA_VISIBLE_DEVICES = 0 python MedQA - ChatGLM / load_export_weights . py
- - save_weights_path . / save_lora| Méthode de réglage fin | Modèle de poids | Durée de la formation | Tournées d'entraînement |
|---|---|---|---|
| Lora | Medqa-chatglm-lora | 28h | 10 |
| P-Tuning v2 | Medqa-chatglm-puningv2 | 27h | 10 |
| Geler | Medqa-chatglm-gezé | 28h | 10 |
* L'expérience a été réalisée sur le système Linux, A100 (1x, 80 Go)
Les ressources liées à ce projet concernent uniquement la recherche universitaire et sont strictement interdites à des fins commerciales. Lorsque vous utilisez des pièces impliquant du code tiers, veuillez suivre strictement le protocole open source correspondant. Le contenu généré par le modèle est affecté par des facteurs tels que le calcul du modèle, le hasard et les pertes de précision quantitative, et ce projet ne peut garantir sa précision. La plupart des ensembles de données de ce projet sont générés par des modèles et ne peuvent pas être utilisés comme base pour le diagnostic médical réel même s'ils sont conformes à certains faits médicaux. Ce projet n'assume aucune responsabilité juridique pour toute sortie de contenu par le modèle, et elle n'est pas responsable des pertes pouvant résulter de l'utilisation des ressources pertinentes et des résultats de sortie.