NOTA IMPORTANTE: A romã v1.0.0 é uma reescrita de romã em terra Embora a mesma funcionalidade seja suportada, a API é significativamente diferente. Consulte as pastas Tutoriais e Exemplos para obter ajuda a reescrever seu código.
ReadThEdocs | Tutoriais | Exemplos
A romã é uma biblioteca para modelagem probabilística definida por sua implementação e tratamento modular de todos os modelos como as distribuições de probabilidade que são. A implementação modular permite soltar facilmente distribuições normais em um modelo de mistura para criar um modelo de mistura gaussiano com a mesma facilidade que soltar uma gama e uma distribuição de Poisson em um modelo de mistura para criar uma mistura heterogênea. Mas isso não é tudo! Como cada modelo é tratado como uma distribuição de probabilidade, as redes bayesianas podem ser lançadas em uma mistura com a mesma facilidade que uma distribuição normal, e os modelos ocultos de Markov podem ser lançados em classificadores de Bayes para fazer um classificador sobre sequências. Juntos, essas duas opções de design permitem uma flexibilidade não vista em nenhum outro pacote de modelagem probabilístico.
Recentemente, a romã (v1.0.0) foi reescrita desde o início usando Pytorch para substituir o back -end desatualizado do Cython. Essa reescrita me deu a oportunidade de consertar muitas opções ruins de design que fiz como engenheiro de software da BB. Infelizmente, muitas dessas mudanças não são compatíveis com versões anteriores e interromperão os fluxos de trabalho. Por outro lado, essas mudanças aceleraram significativamente a maioria dos métodos, melhoraram e simplificaram o código, corrigiram muitos problemas levantados pela comunidade ao longo dos anos e tornaram significativamente mais fácil contribuir. Eu escrevi mais abaixo, mas você provavelmente está aqui agora porque seu código está quebrado e este é o TL; DR.
Gritos especiais para o NumFocus por apoiar este trabalho com uma bolsa de desenvolvimento especial.
pip install pomegranate
Se você precisar da última liberação do Cython antes da reescrita, use pip install pomegranate==0.14.8 . Pode ser necessário instalar manualmente uma versão do Cython antes da V3.
Esta reescrita foi motivada por quatro razões principais:
torch.nn.Moduletorch.masked.MaskedTensorNormalDistribution seja NormalFactorGraph agora é apoiado como cidadãos de primeira classe, com todos os métodos de previsão e treinamentoDenseHMM e SparseHMM que diferem na maneira como a matriz de transição é codificada, com os objetos DenseHMM sendo significativamente mais rápidos em gráficos verdadeiramente densosNaiveBayes foi removido permanentemente, pois é redundante com BayesClassifierMarkovNetwork ainda não foi implementado A maioria dos modelos e métodos na romã v1.0.0 é mais rápida que seus colegas nas versões anteriores. Isso geralmente é escalado por complexidade, onde se vê apenas pequenas acelerações para distribuições simples em pequenos conjuntos de dados, mas acelerações muito maiores para modelos mais complexos em conjuntos de big data, por exemplo, treinamento de modelos de Markov oculto ou inferência de rede bayesiana. A exceção notável por enquanto é que o aprendizado da estrutura de rede bayesiano, além da construção de árvores Chow-Liu, ainda é incompleta e não muito mais rápida. Nos exemplos abaixo, torchegranate refere -se ao repositório temporariamente usado para desenvolver romã v1.0.0 e pomegranate refere -se a romã v0.14.8.
Quem sabe o que está acontecendo aqui? Selvagem.
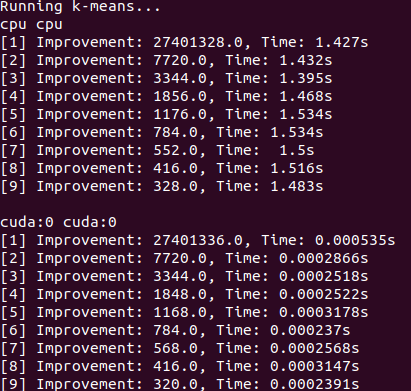
Matriz de transição densa (CPU)
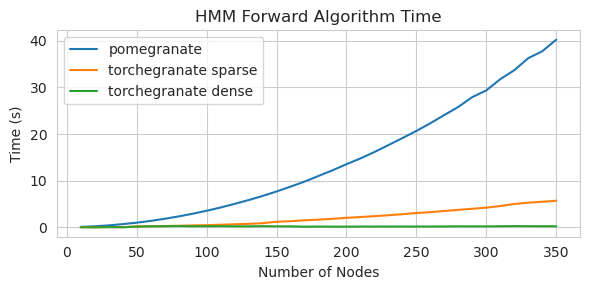
Matriz de transição esparsa (CPU)
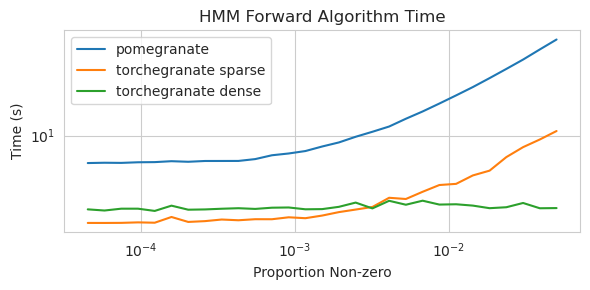
Treinamento de um modelo de 125 nó com uma matriz de densa de transição
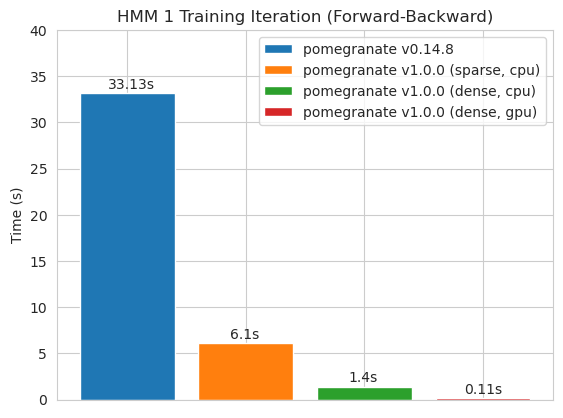
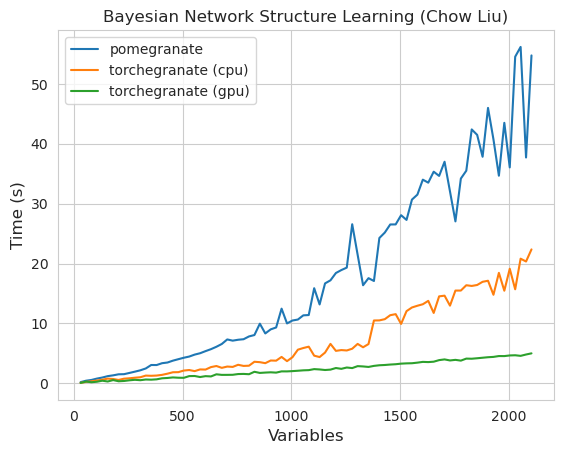
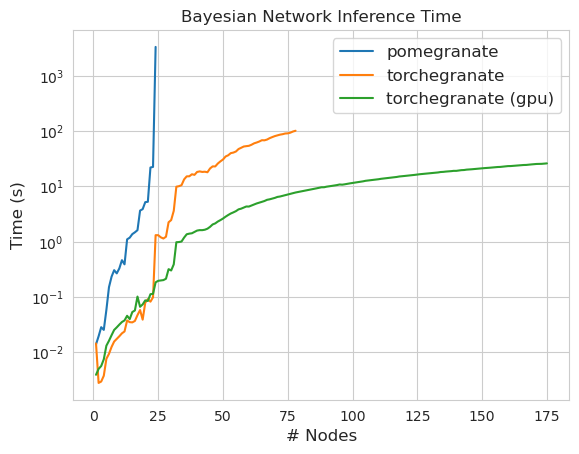
Nota Consulte a pasta Tutoriais para obter exemplos de código.
A mudança de um back -end do Cython para um back -end do Pytorch ativou ou expandiu um grande número de recursos. Como o reescrita é um invólucro fino sobre o Pytorch, à medida que os novos recursos são lançados para o Pytorch, eles podem ser aplicados aos modelos de romã sem a necessidade de um novo lançamento meu.
Todas as distribuições e métodos em romã agora têm suporte à GPU. Como cada distribuição é um objeto torch.nn.Module , o uso é idêntico a outro código escrito em Pytorch. Isso significa que o modelo e os dados devem ser movidos para a GPU pelo usuário. Por exemplo:
> >> X = torch . exp ( torch . randn ( 50 , 4 ))
# Will execute on the CPU
> >> d = Exponential (). fit ( X )
> >> d . scales
Parameter containing :
tensor ([ 1.8627 , 1.3132 , 1.7187 , 1.4957 ])
# Will execute on a GPU
> >> d = Exponential (). cuda (). fit ( X . cuda ())
> >> d . scales
Parameter containing :
tensor ([ 1.8627 , 1.3132 , 1.7187 , 1.4957 ], device = 'cuda:0' )Da mesma forma, todos os modelos são distribuições e, portanto, podem ser usados na GPU da mesma forma. Quando um modelo é movido para a GPU, todos os modelos associados a ele (por exemplo, distribuições) também são movidos para a GPU.
> >> X = torch . exp ( torch . randn ( 50 , 4 )). cuda ()
> >> model = GeneralMixtureModel ([ Exponential (), Exponential ()]). cuda ()
> >> model . fit ( X )
[ 1 ] Improvement : 1.26068115234375 , Time : 0.001134 s
[ 2 ] Improvement : 0.168121337890625 , Time : 0.001097 s
[ 3 ] Improvement : 0.037841796875 , Time : 0.001095 s
>> > model . distributions [ 0 ]. scales
Parameter containing :
>> > model . distributions [ 1 ]. scales
tensor ([ 0.9141 , 1.0835 , 2.7503 , 2.2475 ], device = 'cuda:0' )
Parameter containing :
tensor ([ 1.9902 , 2.3871 , 0.8984 , 1.2215 ], device = 'cuda:0' )Os modelos de romã podem, em teoria, operar nos mesmos regimes de precisão mista ou de baixa precisão que outros módulos de Pytorch. No entanto, como a romã usa operações mais complexas do que a maioria das redes neurais, isso às vezes não funciona ou ajuda na prática porque essas operações não foram otimizadas ou implementadas no regime de baixa precisão. Portanto, espero que esse recurso se torne mais útil ao longo do tempo.
> >> X = torch . randn ( 100 , 4 )
> >> d = Normal ( covariance_type = 'diag' )
> >>
>> > with torch . autocast ( 'cuda' , dtype = torch . bfloat16 ):
> >> d . fit ( X ) As distribuições de romã são todas as instâncias de torch.nn.Module e, portanto, a serialização é a mesma que qualquer outro modelo Pytorch.
Salvando:
> >> X = torch . exp ( torch . randn ( 50 , 4 )). cuda ()
> >> model = GeneralMixtureModel ([ Exponential (), Exponential ()], verbose = True )
> >> model . cuda ()
> >> model . fit ( X )
> >> torch . save ( model , "test.torch" )Carregando:
> >> model = torch . load ( "test.torch" )Note
torch.compileestá sob desenvolvimento ativo da equipe Pytorch e pode melhorar rapidamente. Por enquanto, pode ser necessário passar emcheck_data=Falseao inicializar modelos para evitar um problema de compatibilidade.
Em Pytorch v2.0.0, torch.compile foi introduzida como um invólucro flexível em torno de ferramentas que fundiriam operações, usariam gráficos CUDA e geralmente tentavam remover gargalos de E/S na execução da GPU. Como esses gargalos podem ser extremamente significativos nas configurações de dados de pequeno a médio porte que muitos usuários de romã se deparam, torch.compile parece que será extremamente valioso. Em vez de direcionar modelos inteiros, que principalmente compilam o método forward , você deve compilar métodos individuais de seus objetos.
# Create your object as normal
> >> mu = torch . exp ( torch . randn ( 100 ))
> >> d = Exponential ( mu ). cuda ()
# Create some data
> >> X = torch . exp ( torch . randn ( 1000 , 100 ))
> >> d . log_probability ( X )
# Compile the `log_probability` method!
> >> d . log_probability = torch . compile ( d . log_probability , mode = 'reduce-overhead' , fullgraph = True )
> >> d . log_probability ( X ) Infelizmente, tive dificuldade em fazer com que torch.compile funcione quando os métodos são chamados de maneira aninhada, por exemplo, ao compilar o método predict para um modelo de mistura que, dentro dele, chama o método log_probability de cada distribuição. Eu tentei organizar o código de uma maneira que evite alguns desses erros, mas como as mensagens de erro agora são opacas, tive alguma dificuldade.
A romã suporta o manuseio de dados com valores ausentes através torch.masked.MaskedTensor Objetos. Simplesmente, é preciso apenas colocar uma máscara sobre os valores que estão faltando.
> >> X = < your tensor with NaN for the missing values >
> >> mask = ~ torch . isnan ( X )
>> > X_masked = torch . masked . MaskedTensor ( X , mask = mask )
>> > d = Normal ( covariance_type = 'diag' ). fit ( X_masked )
>> > d . means
Parameter containing :
tensor ([ 0.2271 , 0.0290 , 0.0763 , 0.0135 ])Atualmente, todos os algoritmos tratam a falta como algo para ignorar. Como exemplo, ao calcular a média de uma coluna com valores ausentes, a média será simplesmente o valor médio dos valores presentes. Os valores ausentes não são imputados porque a imputação inadequada pode influenciar seus dados, produzir estimativas improváveis que distorcem distribuições e também diminuem a variação.
Como nem todas as operações ainda estão disponíveis para os tensores mascarados, as seguintes distribuições ainda não são suportadas para valores ausentes: Bernoulli, categórico, normal com covariância total, uniforme
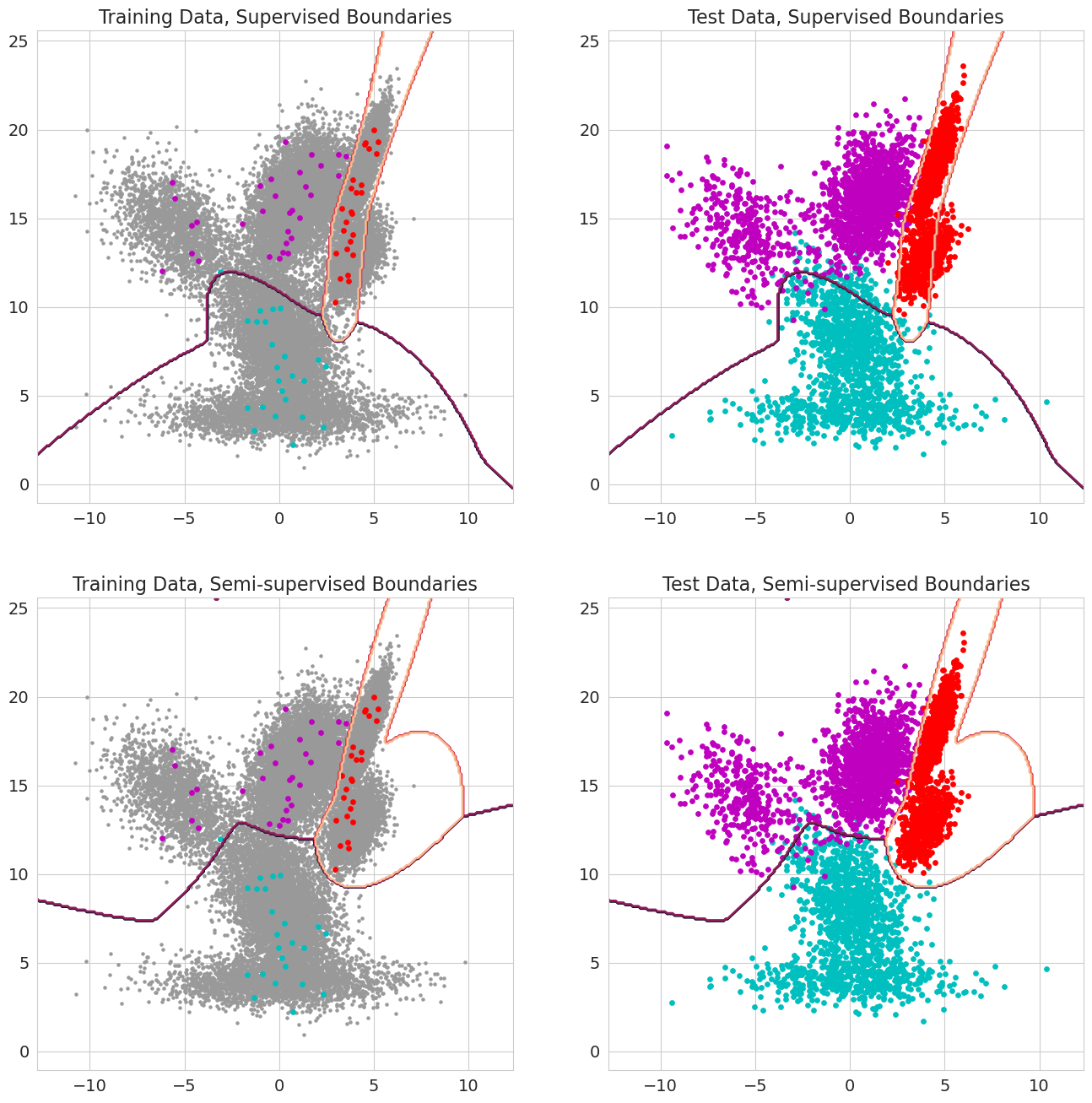
Um novo recurso no romã v1.0.0 é capaz de passar em probabilidades anteriores para cada observação para modelos de mistura, classificadores de Bayes e modelos ocultos de Markov. Essa é a probabilidade anterior de que uma observação pertence a um componente do modelo antes de avaliar a probabilidade e variar entre 0 e 1. Quando esses valores incluem um 1.0 para uma observação, é tratada como um rótulo, porque a probabilidade não é mais importante em termos de atribuição dessa observação a um estado. Portanto, pode-se usar essas probabilidades anteriores para realizar treinamento rotulado quando cada observação tiver um 1,0 para algum aprendizado semi-supervisionado de estado quando um subconjunto de observações (inclusive quando as seqüências são rotuladas apenas parcialmente para modelos ocultos de Markov) ou formas mais sofisticadas de ponderação quando os valores estão entre 0 e 1.