HINWEIS WICHTIG : Granatapfel v1.0.0 ist ein Grund-Up-Rewrite von Granatapfel mit Pytorch als Computer-Backend anstelle von Cython. Obwohl die gleiche Funktionalität unterstützt wird, unterscheidet sich die API erheblich. In den Ordnern von Tutorials und Beispielen finden Sie beim Umschreiben Ihres Codes.
Redethedocs | Tutorials | Beispiele
Granatapfel ist eine Bibliothek für die probabilistische Modellierung, die durch ihre modulare Implementierung und Behandlung aller Modelle als Wahrscheinlichkeitsverteilungen definiert ist. Die modulare Implementierung ermöglicht es, Normalverteilungen einfach in ein Mischmodell fallen zu lassen, um ein Gaußsche Mischungsmodell genauso einfach zu erstellen wie ein Gamma und eine Poisson -Verteilung in ein Mischungsmodell, um eine heterogene Mischung zu erzeugen. Aber das ist nicht alles! Da jedes Modell als Wahrscheinlichkeitsverteilung behandelt wird, können Bayes'sche Netzwerke genauso einfach in eine Mischung fallen gelassen werden wie eine Normalverteilung, und versteckte Markov -Modelle können in Bayes -Klassifikatoren fallen gelassen werden, um einen Klassifizierer über Sequenzen zu erstellen. Zusammen ermöglichen diese beiden Designoptionen eine Flexibilität, die in keinem anderen probabilistischen Modellierungspaket zu sehen ist.
Kürzlich wurde Granatapfel (v1.0.0) mit Pytorch von Grund auf neu geschrieben, um das veraltete Cython -Backend zu ersetzen. Dieses Umschreiben gab mir die Möglichkeit, viele schlechte Designoptionen zu beheben, die ich als BB -Software -Ingenieur getroffen habe. Leider sind viele dieser Änderungen nicht rückwärts kompatibel und stören die Workflows. Auf der anderen Seite haben diese Änderungen die meisten Methoden erheblich beschleunigt, den Code verbessert und vereinfacht, viele Probleme, die von der Community im Laufe der Jahre aufgeworfen wurden, behoben und es erheblich erleichtert, einen Beitrag zu leisten. Ich habe unten mehr geschrieben, aber Sie sind jetzt wahrscheinlich hier, weil Ihr Code gebrochen ist und dies der TL; dr.
Speziales Schieben an Numfocus für die Unterstützung dieser Arbeit mit einem speziellen Entwicklungszuschuss.
pip install pomegranate
Wenn Sie die letzte Cython -Version vor dem Umschreiben benötigen, verwenden Sie pip install pomegranate==0.14.8 . Möglicherweise müssen Sie eine Version von Cython vor V3 manuell installieren.
Dieses Umschreiben wurde durch vier Hauptgründe motiviert:
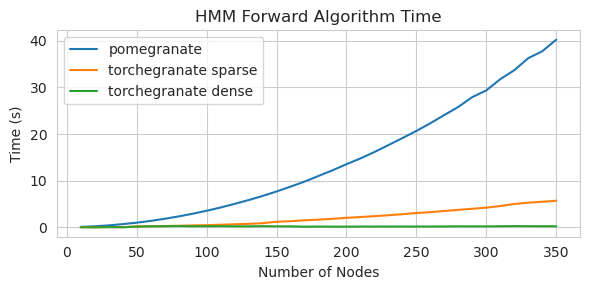
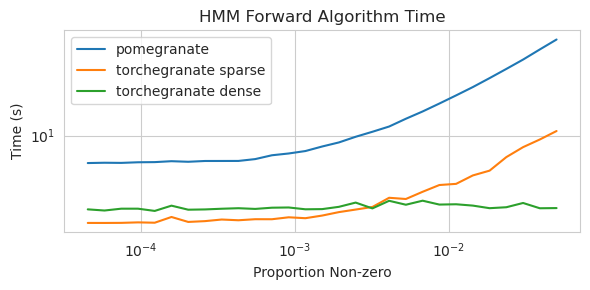
torch.nn.Moduletorch.masked.MaskedTensor -Objekte unterstütztNormalDistribution jetzt Normal istFactorGraph wird jetzt als erstklassige Bürger mit allen Vorhersage- und Trainingsmethoden unterstütztDenseHMM und SparseHMM -Modelle aufgeteilt, die sich in der Art der Übergangsmatrix unterscheiden, wobei DenseHMM -Objekte in wirklich dichten Graphen erheblich schneller sindNaiveBayes wurde dauerhaft entfernt, da es mit BayesClassifier überflüssig istMarkovNetwork wurde noch nicht implementiert Die meisten Modelle und Methoden in Granatapfel v1.0.0 sind schneller als ihre Gegenstücke in früheren Versionen. Dies skaliert im Allgemeinen nach Komplexität, wobei nur kleine Beschleunigungen für einfache Verteilungen an kleinen Datensätzen, aber viel größere Beschleunigungen für komplexere Modelle für Big -Data -Sets, z. B. ein Hidden Markov -Modelltraining oder Bayes'sche Netzwerkinferenz zu sehen sind. Die bemerkenswerte Ausnahme ist vorerst, dass das Lernen von Bayesian-Netzwerkstruktur als das Gebäude von Chow-Liu-Baum immer noch unvollständig und nicht viel schneller ist. In den folgenden Beispielen bezieht sich torchegranate auf das vorübergehende Repository zur Entwicklung von Granatapfel v1.0.0 und pomegranate auf Granatapfel V0.14.8.
Wer weiß, was hier passiert? Wild.
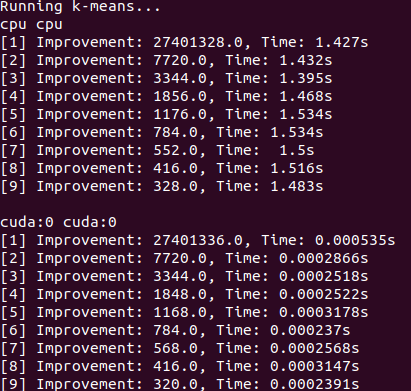
Dichte Übergangsmatrix (CPU)
Spärliche Übergangsmatrix (CPU)
Training eines 125 Knotenmodells mit einer dichten Übergangsmatrix
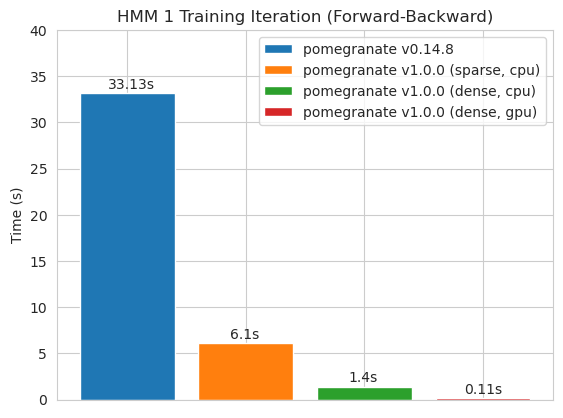
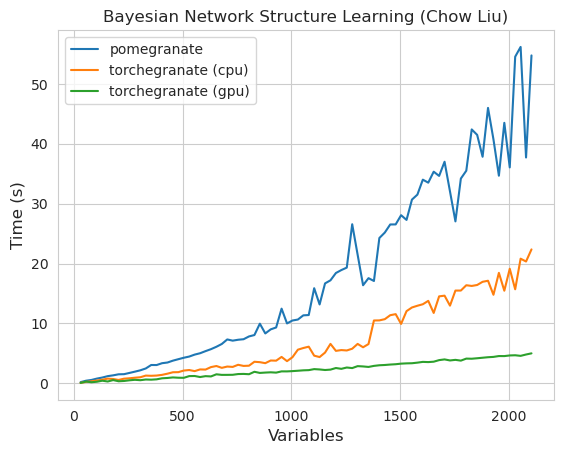
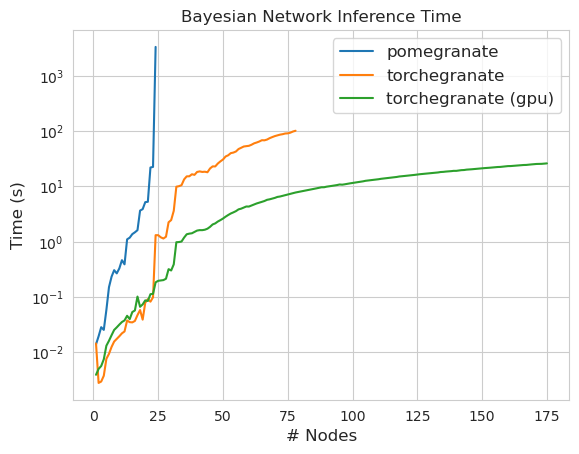
Hinweis finden Sie im Ordner Tutorials für Code -Beispiele.
Das Umschalten von einem Cython -Backend zu einem Pytorch -Backend hat eine große Anzahl von Funktionen aktiviert oder erweitert. Da das Umschreiben ein dünner Wrapper über Pytorch ist, können neue Funktionen für Pytorch veröffentlicht werden, sie können auf Granatapfelmodelle angewendet werden, ohne dass eine neue Version von mir erforderlich ist.
Alle Verteilungen und Methoden im Granatapfel haben jetzt GPU -Unterstützung. Da jede Verteilung ein torch.nn.Module -Objekt ist, ist die Verwendung identisch mit einem anderen in Pytorch geschriebenen Code. Dies bedeutet, dass sowohl das Modell als auch die Daten vom Benutzer an die GPU verschoben werden müssen. Zum Beispiel:
> >> X = torch . exp ( torch . randn ( 50 , 4 ))
# Will execute on the CPU
> >> d = Exponential (). fit ( X )
> >> d . scales
Parameter containing :
tensor ([ 1.8627 , 1.3132 , 1.7187 , 1.4957 ])
# Will execute on a GPU
> >> d = Exponential (). cuda (). fit ( X . cuda ())
> >> d . scales
Parameter containing :
tensor ([ 1.8627 , 1.3132 , 1.7187 , 1.4957 ], device = 'cuda:0' )Ebenso sind alle Modelle Verteilungen und können daher in ähnlicher Weise für die GPU verwendet werden. Wenn ein Modell in die GPU verschoben wird, werden alle damit verbundenen Modelle (z. B. Verteilungen) auch in die GPU verschoben.
> >> X = torch . exp ( torch . randn ( 50 , 4 )). cuda ()
> >> model = GeneralMixtureModel ([ Exponential (), Exponential ()]). cuda ()
> >> model . fit ( X )
[ 1 ] Improvement : 1.26068115234375 , Time : 0.001134 s
[ 2 ] Improvement : 0.168121337890625 , Time : 0.001097 s
[ 3 ] Improvement : 0.037841796875 , Time : 0.001095 s
>> > model . distributions [ 0 ]. scales
Parameter containing :
>> > model . distributions [ 1 ]. scales
tensor ([ 0.9141 , 1.0835 , 2.7503 , 2.2475 ], device = 'cuda:0' )
Parameter containing :
tensor ([ 1.9902 , 2.3871 , 0.8984 , 1.2215 ], device = 'cuda:0' )Granatapfelmodelle können theoretisch in den gleichen gemischten oder prezisionsarmen Regimen wie andere Pytorch-Module funktionieren. Da Granatapfel jedoch komplexere Operationen einsetzt als die meisten neuronalen Netze, funktioniert dies manchmal nicht in der Praxis, da diese Vorgänge im Regime mit niedrigem Präzision nicht optimiert oder implementiert wurden. Hoffentlich wird diese Funktion im Laufe der Zeit nützlicher.
> >> X = torch . randn ( 100 , 4 )
> >> d = Normal ( covariance_type = 'diag' )
> >>
>> > with torch . autocast ( 'cuda' , dtype = torch . bfloat16 ):
> >> d . fit ( X ) Granatapfelverteilungen sind alle Fälle von torch.nn.Module und so ist die Serialisierung mit jedem anderen Pytorch -Modell dieselbe.
Sparen:
> >> X = torch . exp ( torch . randn ( 50 , 4 )). cuda ()
> >> model = GeneralMixtureModel ([ Exponential (), Exponential ()], verbose = True )
> >> model . cuda ()
> >> model . fit ( X )
> >> torch . save ( model , "test.torch" )Laden:
> >> model = torch . load ( "test.torch" )HINWEIS
torch.compilewird vom Pytorch -Team aktiv entwickelt und kann sich schnell verbessern. Im Moment müssen Sie möglicherweise incheck_data=Falseübergeben.
In Pytorch v2.0.0 wurde torch.compile als flexible Wrapper um Werkzeuge eingeführt, die den Vorgängen zusammenfügen, CUDA -Diagramme verwenden und im Allgemeinen versuchen, E/A -Engpässe in der GPU -Ausführung zu entfernen. Da diese Engpässe in den Dateneinstellungen mit kleiner bis mittlerer Größe äußerst wichtig sein können, mit denen viele Granatapfel-Benutzer konfrontiert sind, scheint torch.compile , dass es äußerst wertvoll sein wird. Anstatt auf ganze Modelle zu zielen, die meistens nur die forward kompiliert, sollten Sie einzelne Methoden aus Ihren Objekten kompilieren.
# Create your object as normal
> >> mu = torch . exp ( torch . randn ( 100 ))
> >> d = Exponential ( mu ). cuda ()
# Create some data
> >> X = torch . exp ( torch . randn ( 1000 , 100 ))
> >> d . log_probability ( X )
# Compile the `log_probability` method!
> >> d . log_probability = torch . compile ( d . log_probability , mode = 'reduce-overhead' , fullgraph = True )
> >> d . log_probability ( X ) Leider hatte ich Schwierigkeiten, torch.compile zum Laufen zu bringen, wenn Methoden verschachtelt werden, z. B. beim Kompilieren der predict für ein Mischungsmodell, das darin die Methode log_probability jeder Verteilung aufruft. Ich habe versucht, den Code so zu organisieren, dass einige dieser Fehler vermieden werden, aber da die Fehlermeldungen im Moment undurchsichtig sind, hatte ich einige Schwierigkeiten.
Granatapfel unterstützt die Handhabung von Daten mit fehlenden Werten über torch.masked.MaskedTensor -Objekte. Einfach muss man einfach eine Maske über die fehlenden Werte legen.
> >> X = < your tensor with NaN for the missing values >
> >> mask = ~ torch . isnan ( X )
>> > X_masked = torch . masked . MaskedTensor ( X , mask = mask )
>> > d = Normal ( covariance_type = 'diag' ). fit ( X_masked )
>> > d . means
Parameter containing :
tensor ([ 0.2271 , 0.0290 , 0.0763 , 0.0135 ])Alle Algorithmen behandeln derzeit Fehlern als etwas zu ignorieren. Wenn beispielsweise der Mittelwert einer Spalte mit fehlenden Werten berechnet wird, ist der Mittelwert einfach der Durchschnittswert der gegenwärtigen Werte. Fehlende Werte werden nicht unterstellt, da eine unsachgemäße Imputation Ihre Daten verzerren, unwahrscheinliche Schätzungen erzeugen, die Verteilungen verzerren und auch die Varianz verkleinern.
Da noch nicht alle Operationen für Maskedtensoren verfügbar sind, werden die folgenden Verteilungen noch nicht für fehlende Werte unterstützt: Bernoulli, kategorisch, normal mit voller Kovarianz, Uniform
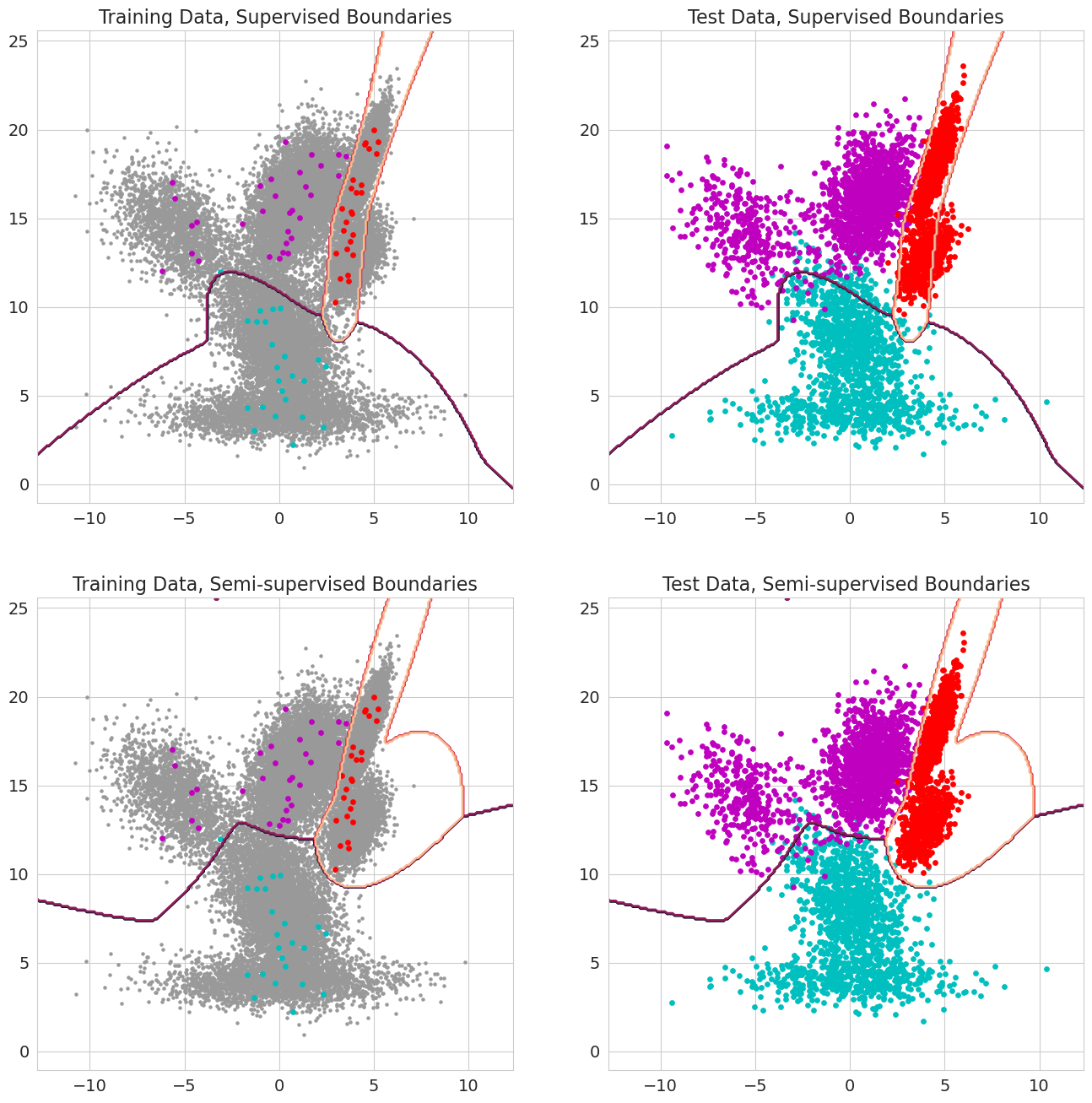
Ein neues Merkmal in Granatapfel V1.0.0 ist die Möglichkeit, frühere Wahrscheinlichkeiten für jede Beobachtung für Mischmodelle, Bayes -Klassifizierer und Hidden Markov -Modelle zu verabschieden. Dies ist die vorherige Wahrscheinlichkeit, dass eine Beobachtung zu einer Komponente des Modells gehört, bevor die Wahrscheinlichkeit bewertet wird, und sollte zwischen 0 und 1 liegen. Wenn diese Werte für eine Beobachtung eine 1,0 enthalten, wird sie als Etikett behandelt, da die Wahrscheinlichkeit nicht mehr wichtig ist, diese Beobachtung einem Zustand zuzuweisen. Daher kann man diese vorherigen Wahrscheinlichkeiten nutzen, um ein markiertes Training durchzuführen, wenn jede Beobachtung einen 1,0 für einen staatlichen, halb überbewerteten Lernen aufweist, wenn eine Untergruppe von Beobachtungen (einschließlich, wenn Sequenzen nur teilweise für versteckte Markov-Modelle markiert sind) oder ausgefeiltere Gewichtsformen, wenn die Werte zwischen 0 und 1 liegen.