Este repositório contém o código para executar treinamento , indexação e recuperação para modelos Splade. Ele também inclui tudo o que é necessário para lançar a avaliação no benchmark da Beir.
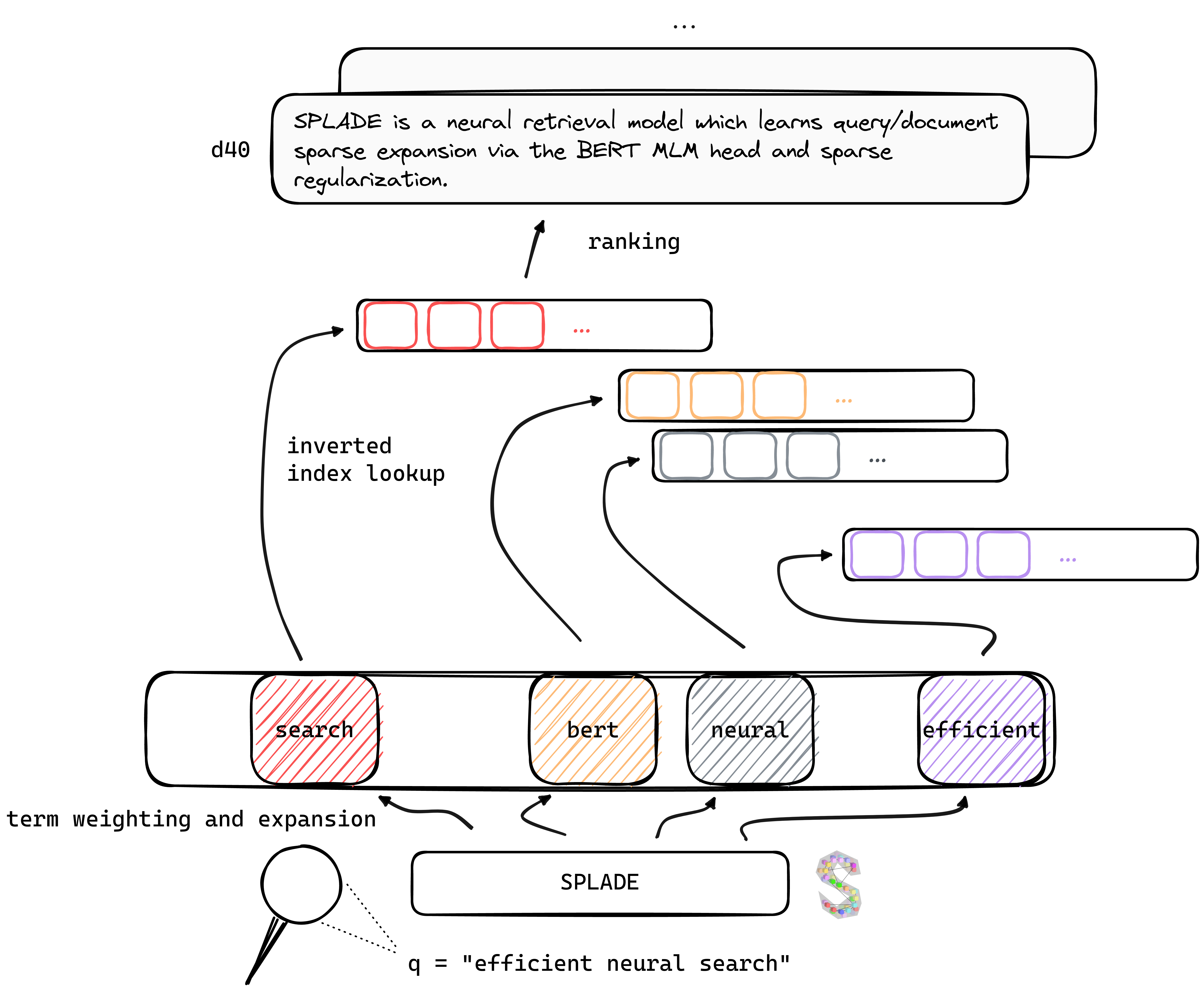
Tl; O DR Splade é um modelo de recuperação neural que aprende a expansão esparsa de consulta/documento através da cabeça do Bert MLM e da regularização esparsa. Representações escassas se beneficiam de várias vantagens em comparação com abordagens densas: uso eficiente do índice invertido, correspondência lexical explícita, interpretabilidade ... elas também parecem ser melhores em generalizar os dados fora do domínio (benchmark da Beir).
Ao se beneficiar dos recentes avanços no treinamento de retrievers neurais, nossos modelos V2 dependem de mineração, destilação e melhor inicialização de modelos de linguagem pré-treinados para aumentar ainda mais sua eficácia , tanto no domínio (MS MARCO) quanto na avaliação fora do domínio (benchmark BEIR).
Finalmente, ao introduzir várias modificações (regularização específica da consulta, codificadores disjuntos etc.), somos capazes de melhorar a eficiência , alcançando a latência em pé de igualdade com o BM25 sob as mesmas restrições de computação.
Os pesos para modelos treinados em várias configurações podem ser encontrados no site da Naver Labs Europe, além de abraçar o rosto. Lembre -se de que o splade é mais uma classe de modelos do que um modelo em si: dependendo da magnitude da regularização, podemos obter diferentes modelos (de muito escassos a modelos fazendo uma intensa expansão de consulta/documento) com diferentes propriedades e desempenho.
Splade: um spork que é afiado ao longo de uma borda ou ambas as bordas, permitindo que ela seja usada como uma faca, um garfo e uma colher.
Recomendamos começar de um ambiente novo e instalar os pacotes de conda_splade_env.yml .
conda create -n splade_env python=3.9
conda activate splade_env
conda env create -f conda_splade_env.yml
inference_splade.ipynb permite carregar e executar inferência com um modelo treinado, a fim de inspecionar o "saco de palavras de exploração" previsto. Fornecemos pesos para seis modelos principais:
| modelo | MRR@10 (Ms Marco Dev) |
|---|---|
naver/splade_v2_max ( v2 hf) | 34.0 |
naver/splade_v2_distil ( v2 hf) | 36.8 |
naver/splade-cocondenser-selfdistil ( Splade ++ , HF) | 37.6 |
naver/splade-cocondenser-ensembledistil ( splade ++ , HF) | 38.3 |
naver/efficient-splade-V-large-doc (HF) + naver/efficient-splade-V-large-query (HF) ( splade eficiente ) | 38.8 |
naver/efficient-splade-VI-BT-large-doc (HF) + efficient-splade-VI-BT-large-query (HF) ( Splade eficiente ) | 38.0 |
Também enviamos vários modelos aqui. Sinta -se à vontade para experimentá -los!
train.py ), index ( index.py ), recuperar ( retrieve.py ) (ou executar todas as etapas com os modelos Splade all.py ). Para simplificar a configuração, disponibilizamos todas as nossas pastas de dados, que podem ser baixadas aqui. Este link inclui consultas, documentos e dados negativos difíceis, permitindo o treinamento na configuração EnsembleDistil (consulte o papel V2BIS). Para outras configurações ( Simple , DistilMSE , SelfDistil ), você também precisa fazer o download:
Simple ) trigêmeos BM25 padrãoDistilMSE ) trigêmeos "Viena" para destilação de marginmseSelfDistil ) trigêmeos extraídos de spladeApós o download, você pode simplesmente não no diretório raiz e ele será colocado na pasta direita.
tar -xzvf file.tar.gz
Para executar todas as etapas (aqui nos dados dos brinquedos, ou seja, config_default.yaml ), vá no diretório raiz e execute:
conda activate splade_env
export PYTHONPATH= $PYTHONPATH : $( pwd )
export SPLADE_CONFIG_NAME= " config_default.yaml "
python3 -m splade.all
config.checkpoint_dir=experiments/debug/checkpoint
config.index_dir=experiments/debug/index
config.out_dir=experiments/debug/outFornecemos exemplos adicionais que podem ser conectados no código acima. Consulte conf/readme.md para obter detalhes sobre como alterar as configurações de experimento.
python3 -m splade.train (o mesmo para indexação ou recuperação)SPLADE_CONFIG_FULLPATH=/path/to/checkpoint/dir/config.yaml python3 -m splade.create_anserini +quantization_factor_document=100 +quantization_factor_query=100/conf . Por exemplo, para executar a configuração SelfDistil :SPLADE_CONFIG_NAME=config_splade++_selfdistil.yamlpython3 -m splade.all config.regularizer.FLOPS.lambda_q=0.06 config.regularizer.FLOPS.lambda_d=0.02 Fornecemos várias configurações básicas que correspondem aos experimentos nos documentos V2BIs e "eficiência". Observe que eles são adequados para a configuração de hardware, ou seja, 4 GPUs Tesla V100 com memória de 32 GB. Para treinar modelos com por exemplo, uma GPU, você precisa diminuir o tamanho do lote para treinamento e avaliação. Observe também que, como o intervalo para a perda pode mudar com um tamanho de lote diferente, os lambdas correspondentes para regularização podem precisar ser adaptados. No entanto, fornecemos uma configuração mono-gpu config_splade++_cocondenser_ensembledistil_monogpu.yaml para a qual obtemos 37.2 mrr@10, treinados em uma única GPU de 16 GB.
A indexação (e a recuperação) pode ser feita usando nossa implementação (baseada em NUMBA) do índice invertido ou Anserini. Vamos executar essas etapas usando um modelo disponível ( naver/splade-cocondenser-ensembledistil ).
conda activate splade_env
export PYTHONPATH= $PYTHONPATH : $( pwd )
export SPLADE_CONFIG_NAME= " config_splade++_cocondenser_ensembledistil "
python3 -m splade.index
init_dict.model_type_or_dir=naver/splade-cocondenser-ensembledistil
config.pretrained_no_yamlconfig=true
config.index_dir=experiments/pre-trained/index
python3 -m splade.retrieve
init_dict.model_type_or_dir=naver/splade-cocondenser-ensembledistil
config.pretrained_no_yamlconfig=true
config.index_dir=experiments/pre-trained/index
config.out_dir=experiments/pre-trained/out
# pretrained_no_yamlconfig indicates that we solely rely on a HF-valid model pathretrieve_evaluate=msmarco como argumento de splade.retrieve .Você pode criar da mesma forma os arquivos que serão ingeridos por Anserini:
python3 -m splade.create_anserini
init_dict.model_type_or_dir=naver/splade-cocondenser-ensembledistil
config.pretrained_no_yamlconfig=true
config.index_dir=experiments/pre-trained/index
+quantization_factor_document=100
+quantization_factor_query=100 Ele criará a coleção JSON ( docs_anserini.jsonl ), bem como as consultas ( queries_anserini.tsv ) necessárias para Anserini. Você só precisa seguir a regressão para se divirta aqui para indexar e recuperar.
Você também pode executar a avaliação em Beir, por exemplo:
conda activate splade_env
export PYTHONPATH= $PYTHONPATH : $( pwd )
export SPLADE_CONFIG_FULLPATH= " /path/to/checkpoint/dir/config.yaml "
for dataset in arguana fiqa nfcorpus quora scidocs scifact trec-covid webis-touche2020 climate-fever dbpedia-entity fever hotpotqa nq
do
python3 -m splade.beir_eval
+beir.dataset= $dataset
+beir.dataset_path=data/beir
config.index_retrieve_batch_size=100
done Fornecemos em efficient_splade_pisa/README.md as etapas para avaliar modelos eficientes com PISA.
Cite nosso trabalho como:
@inbook{10.1145/3404835.3463098,
author = {Formal, Thibault and Piwowarski, Benjamin and Clinchant, St'{e}phane},
title = {SPLADE: Sparse Lexical and Expansion Model for First Stage Ranking},
year = {2021},
isbn = {9781450380379},
publisher = {Association for Computing Machinery},
address = {New York, NY, USA},
url = {https://doi.org/10.1145/3404835.3463098},
booktitle = {Proceedings of the 44th International ACM SIGIR Conference on Research and Development in Information Retrieval},
pages = {2288–2292},
numpages = {5}
}
@misc{https://doi.org/10.48550/arxiv.2109.10086,
doi = {10.48550/ARXIV.2109.10086},
url = {https://arxiv.org/abs/2109.10086},
author = {Formal, Thibault and Lassance, Carlos and Piwowarski, Benjamin and Clinchant, Stéphane},
keywords = {Information Retrieval (cs.IR), Artificial Intelligence (cs.AI), Computation and Language (cs.CL), FOS: Computer and information sciences, FOS: Computer and information sciences},
title = {SPLADE v2: Sparse Lexical and Expansion Model for Information Retrieval},
publisher = {arXiv},
year = {2021},
copyright = {Creative Commons Attribution Non Commercial Share Alike 4.0 International}
}
@inproceedings{10.1145/3477495.3531857,
author = {Formal, Thibault and Lassance, Carlos and Piwowarski, Benjamin and Clinchant, St'{e}phane},
title = {From Distillation to Hard Negative Sampling: Making Sparse Neural IR Models More Effective},
year = {2022},
isbn = {9781450387323},
publisher = {Association for Computing Machinery},
address = {New York, NY, USA},
url = {https://doi.org/10.1145/3477495.3531857},
doi = {10.1145/3477495.3531857},
abstract = {Neural retrievers based on dense representations combined with Approximate Nearest Neighbors search have recently received a lot of attention, owing their success to distillation and/or better sampling of examples for training -- while still relying on the same backbone architecture. In the meantime, sparse representation learning fueled by traditional inverted indexing techniques has seen a growing interest, inheriting from desirable IR priors such as explicit lexical matching. While some architectural variants have been proposed, a lesser effort has been put in the training of such models. In this work, we build on SPLADE -- a sparse expansion-based retriever -- and show to which extent it is able to benefit from the same training improvements as dense models, by studying the effect of distillation, hard-negative mining as well as the Pre-trained Language Model initialization. We furthermore study the link between effectiveness and efficiency, on in-domain and zero-shot settings, leading to state-of-the-art results in both scenarios for sufficiently expressive models.},
booktitle = {Proceedings of the 45th International ACM SIGIR Conference on Research and Development in Information Retrieval},
pages = {2353–2359},
numpages = {7},
keywords = {neural networks, indexing, sparse representations, regularization},
location = {Madrid, Spain},
series = {SIGIR '22}
}
@inproceedings{10.1145/3477495.3531833,
author = {Lassance, Carlos and Clinchant, St'{e}phane},
title = {An Efficiency Study for SPLADE Models},
year = {2022},
isbn = {9781450387323},
publisher = {Association for Computing Machinery},
address = {New York, NY, USA},
url = {https://doi.org/10.1145/3477495.3531833},
doi = {10.1145/3477495.3531833},
abstract = {Latency and efficiency issues are often overlooked when evaluating IR models based on Pretrained Language Models (PLMs) in reason of multiple hardware and software testing scenarios. Nevertheless, efficiency is an important part of such systems and should not be overlooked. In this paper, we focus on improving the efficiency of the SPLADE model since it has achieved state-of-the-art zero-shot performance and competitive results on TREC collections. SPLADE efficiency can be controlled via a regularization factor, but solely controlling this regularization has been shown to not be efficient enough. In order to reduce the latency gap between SPLADE and traditional retrieval systems, we propose several techniques including L1 regularization for queries, a separation of document/query encoders, a FLOPS-regularized middle-training, and the use of faster query encoders. Our benchmark demonstrates that we can drastically improve the efficiency of these models while increasing the performance metrics on in-domain data. To our knowledge, we propose the first neural models that, under the same computing constraints, achieve similar latency (less than 4ms difference) as traditional BM25, while having similar performance (less than 10% MRR@10 reduction) as the state-of-the-art single-stage neural rankers on in-domain data.},
booktitle = {Proceedings of the 45th International ACM SIGIR Conference on Research and Development in Information Retrieval},
pages = {2220–2226},
numpages = {7},
keywords = {splade, sparse representations, latency, information retrieval},
location = {Madrid, Spain},
series = {SIGIR '22}
}
Sinta -se à vontade para entrar em contato conosco via Twitter ou por Mail @ [email protected]!
Copyright Splade (C) 2021-Apresentante Naver Corp.
Splade é licenciado sob uma licença internacional Creative Commons Attribution-NonCommercial-Sharealike 4.0. (Veja a licença)
Você deveria ter recebido uma cópia da licença junto com este trabalho. Caso contrário, consulte http://creativecommons.org/license/by-nnc-la/4.0/.