Dieses Repository enthält den Code zur Durchführung von Schulungen , Indizieren und Abrufen für Splade -Modelle. Es enthält auch alles, was für die Einführung der Bewertung am BEIR -Benchmark erforderlich ist.
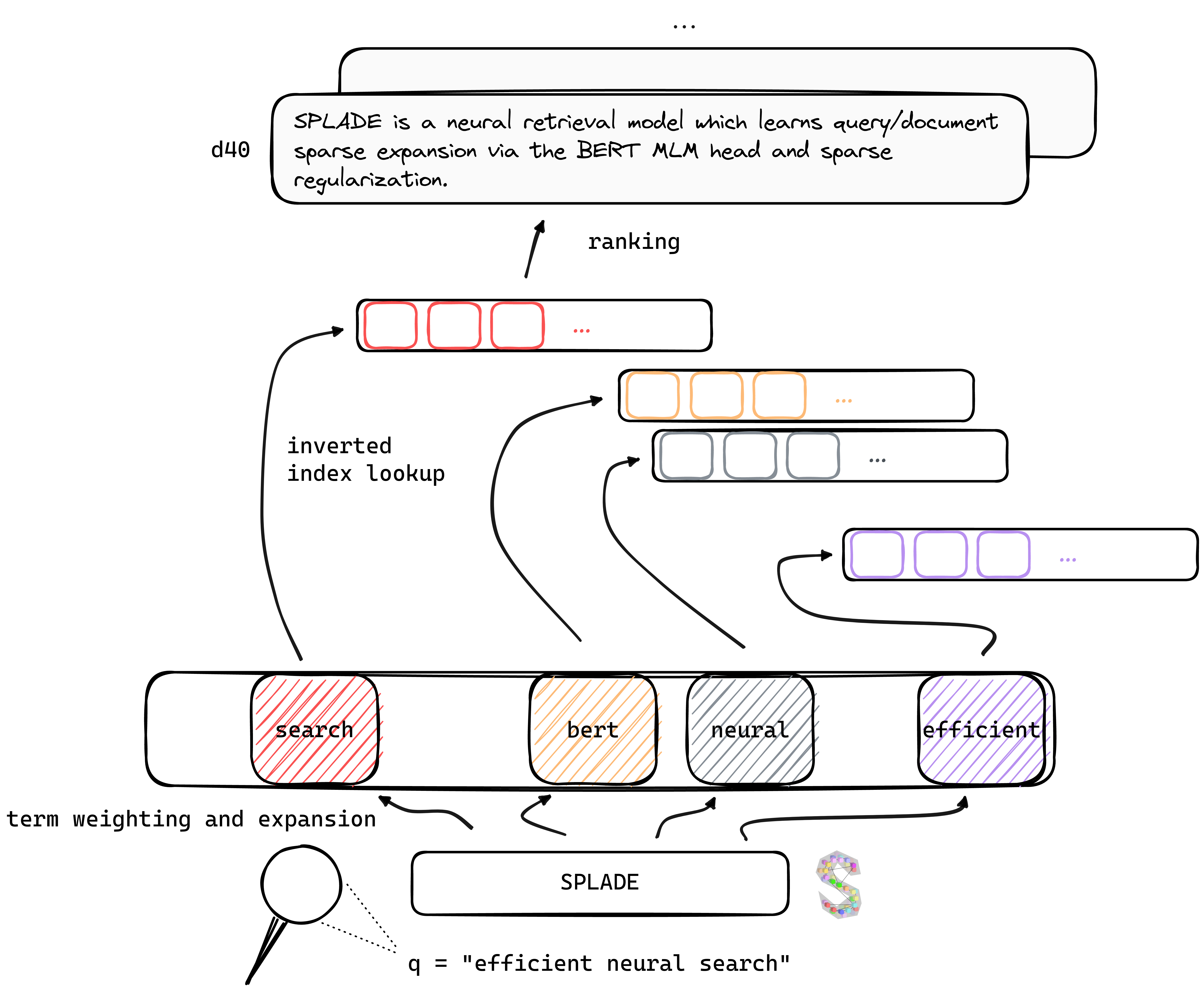
Tl; Dr. Splade ist ein neuronales Abrufmodell, das die spärliche Expansion von Abfragen und Dokumenten über den Bert MLM -Kopf und die spärliche Regularisierung lernt. Spärliche Darstellungen profitieren von mehreren Vorteilen im Vergleich zu dichten Ansätzen: Effizienter Einsatz von invertiertem Index, explizite lexikalische Übereinstimmung, Interpretierbarkeit ... Sie scheinen auch besser auf Daten außerhalb der Domänen zu verallgemeinern (Beir-Benchmark).
Unsere V2- Modelle profitieren von den jüngsten Fortschritten bei der Ausbildung neuronaler Retriever und stützen sich auf hartnegatives Mining, Destillation und eine bessere Initialisierung des vorgebliebenen Sprachmodells, um ihre Wirksamkeit sowohl bei der In-Domäne (MS Marco) als auch auf der Auswertung außerhalb der Domäne (BEIR-Benchmark) weiter zu erhöhen.
Durch die Einführung mehrerer Modifikationen (Abfragespezifische Regularisierung, Disjunkt -Encoder usw.) können wir die Effizienz verbessern und die Latenz mit BM25 unter denselben Rechenbeschränkungen erreichen.
Gewichte für Modelle, die unter verschiedenen Umgebungen trainiert wurden, finden Sie auf der Website von Naver Labs Europe sowie auf dem Umarmungsgesicht. Bitte beachten Sie, dass Splade eher eine Klasse von Modellen als ein Modell an sich ist: Abhängig von der Regularisierungsgröße können wir unterschiedliche Modelle (von sehr spärlich bis zu Modellen, die eine intensive Abfrage-/DOC -Erweiterung durchführen) mit unterschiedlichen Eigenschaften und Leistung erhalten.
Splade: Ein Spork, der an einer Kante oder bei beiden Kanten scharf ist und es ermöglicht, als Messer, Gabel und Löffel verwendet zu werden.
Wir empfehlen, in einer neuen Umgebung aus zu beginnen und die Pakete von conda_splade_env.yml zu installieren.
conda create -n splade_env python=3.9
conda activate splade_env
conda env create -f conda_splade_env.yml
inference_splade.ipynb ermöglicht es Ihnen, mit einem geschulten Modell Schlussfolgerungen zu laden und durchzuführen, um die vorhergesagten "Sack-of-Expanded-Wörter" zu inspizieren. Wir bieten Gewichte für sechs Hauptmodelle:
| Modell | MRR@10 (Frau Marco Dev) |
|---|---|
naver/splade_v2_max ( v2 hf) | 34.0 |
naver/splade_v2_distil ( v2 hf) | 36.8 |
naver/splade-cocondenser-selfdistil ( Splade ++ , HF) | 37.6 |
naver/splade-cocondenser-ensembledistil ( Splade ++ , HF) | 38.3 |
naver/efficient-splade-V-large-doc (HF) + naver/efficient-splade-V-large-query (HF) ( effiziente Splade ) | 38,8 |
naver/efficient-splade-VI-BT-large-doc (HF) + efficient-splade-VI-BT-large-query (HF) ( effiziente Splade ) | 38.0 |
Wir haben hier auch verschiedene Modelle hochgeladen. Fühlen Sie sich frei, sie auszuprobieren!
train.py ), Index ( index.py ), abrufen ( retrieve.py ) (oder jeden Schritt mit all.py ) Splade -Modellen. Um die Einrichtung zu vereinfachen, haben wir alle unsere Datenordner zur Verfügung gestellt, die hier heruntergeladen werden können. Dieser Link enthält Abfragen, Dokumente und harte negative Daten, die ein Training unter der EnsembleDistil ermöglichen (siehe V2BIS -Papier). Für andere Einstellungen ( Simple , DistilMSE , SelfDistil ) müssen Sie auch herunterladen:
Simple ) Standard -BM25 -TriplettsDistilMSE ) "Wien" -Tripletts für die Marginmse -DestillationSelfDistil ) Tripletts aus Splade abgebautNach dem Herunterladen können Sie einfach im Stammverzeichnis enttar werden, und es wird in den richtigen Ordner platziert.
tar -xzvf file.tar.gz
Um alle Schritte auszuführen (hier auf Spielzeugdaten, dh config_default.yaml ), gehen Sie in das Stammverzeichnis und führen Sie aus:
conda activate splade_env
export PYTHONPATH= $PYTHONPATH : $( pwd )
export SPLADE_CONFIG_NAME= " config_default.yaml "
python3 -m splade.all
config.checkpoint_dir=experiments/debug/checkpoint
config.index_dir=experiments/debug/index
config.out_dir=experiments/debug/outWir geben zusätzliche Beispiele an, die im obigen Code eingesteckt werden können. In Conf/Readme.md finden Sie Details zum Ändern der Experiment -Einstellungen.
python3 -m splade.train ausführen (gleiche für die Indexierung oder Abruf)SPLADE_CONFIG_FULLPATH=/path/to/checkpoint/dir/config.yaml python3 -m splade.create_anserini +quantization_factor_document=100 +quantization_factor_query=100/conf verfügbar. Zum Beispiel die SelfDistil Einstellung ausführen:SPLADE_CONFIG_NAME=config_splade++_selfdistil.yamlpython3 -m splade.all config.regularizer.FLOPS.lambda_q=0.06 config.regularizer.FLOPS.lambda_d=0.02 Parameter (z. B. lambdas) außerhalb der Konfiguration weiter zu ändern Wir bieten mehrere Basiskonfigurationen, die den Experimenten in den Papieren von V2BIS und "Effizienz" entsprechen. Bitte beachten Sie, dass diese für unsere Hardware -Einstellung geeignet sind, dh 4 GPUs Tesla V100 mit 32 GB Speicher. Um Modelle mit z. B. einer GPU zu trainieren, müssen Sie die Chargengröße für das Training und die Bewertung verringern. Beachten Sie auch, dass sich entsprechende Lambdas für die Regularisierung möglicherweise angepasst werden müssen, da sich der Bereich für den Verlust mit einer anderen Chargengröße ändern kann. Wir bieten jedoch eine Mono-GPU-Konfiguration config_splade++_cocondenser_ensembledistil_monogpu.yaml für die wir 37,2 MRR@10 erhalten, die auf einer einzigen 16 GB GPU trainiert wird.
Die Indexierung (und das Abrufen) kann entweder mit unserer (Numba-basierten) Implementierung von invertiertem Index oder Anserini durchgeführt werden. Führen Sie diese Schritte mit einem verfügbaren Modell aus ( naver/splade-cocondenser-ensembledistil ).
conda activate splade_env
export PYTHONPATH= $PYTHONPATH : $( pwd )
export SPLADE_CONFIG_NAME= " config_splade++_cocondenser_ensembledistil "
python3 -m splade.index
init_dict.model_type_or_dir=naver/splade-cocondenser-ensembledistil
config.pretrained_no_yamlconfig=true
config.index_dir=experiments/pre-trained/index
python3 -m splade.retrieve
init_dict.model_type_or_dir=naver/splade-cocondenser-ensembledistil
config.pretrained_no_yamlconfig=true
config.index_dir=experiments/pre-trained/index
config.out_dir=experiments/pre-trained/out
# pretrained_no_yamlconfig indicates that we solely rely on a HF-valid model pathretrieve_evaluate=msmarco als Argument von splade.retrieve .In ähnlicher Weise können Sie die Dateien erstellen, die von Anserini aufgenommen werden:
python3 -m splade.create_anserini
init_dict.model_type_or_dir=naver/splade-cocondenser-ensembledistil
config.pretrained_no_yamlconfig=true
config.index_dir=experiments/pre-trained/index
+quantization_factor_document=100
+quantization_factor_query=100 Es erstellt die JSON -Sammlung ( docs_anserini.jsonl ) sowie die Abfragen ( queries_anserini.tsv ), die für Anserini benötigt werden. Sie müssen dann nur die Regression für Splade hier folgen, um zu indizieren und abzurufen.
Sie können beispielsweise auch die Bewertung auf Beir.
conda activate splade_env
export PYTHONPATH= $PYTHONPATH : $( pwd )
export SPLADE_CONFIG_FULLPATH= " /path/to/checkpoint/dir/config.yaml "
for dataset in arguana fiqa nfcorpus quora scidocs scifact trec-covid webis-touche2020 climate-fever dbpedia-entity fever hotpotqa nq
do
python3 -m splade.beir_eval
+beir.dataset= $dataset
+beir.dataset_path=data/beir
config.index_retrieve_batch_size=100
done Wir stellen in efficient_splade_pisa/README.md die Schritte zur Bewertung effizienter Splade -Modelle mit PISA zur Verfügung.
Bitte zitieren Sie unsere Arbeit als:
@inbook{10.1145/3404835.3463098,
author = {Formal, Thibault and Piwowarski, Benjamin and Clinchant, St'{e}phane},
title = {SPLADE: Sparse Lexical and Expansion Model for First Stage Ranking},
year = {2021},
isbn = {9781450380379},
publisher = {Association for Computing Machinery},
address = {New York, NY, USA},
url = {https://doi.org/10.1145/3404835.3463098},
booktitle = {Proceedings of the 44th International ACM SIGIR Conference on Research and Development in Information Retrieval},
pages = {2288–2292},
numpages = {5}
}
@misc{https://doi.org/10.48550/arxiv.2109.10086,
doi = {10.48550/ARXIV.2109.10086},
url = {https://arxiv.org/abs/2109.10086},
author = {Formal, Thibault and Lassance, Carlos and Piwowarski, Benjamin and Clinchant, Stéphane},
keywords = {Information Retrieval (cs.IR), Artificial Intelligence (cs.AI), Computation and Language (cs.CL), FOS: Computer and information sciences, FOS: Computer and information sciences},
title = {SPLADE v2: Sparse Lexical and Expansion Model for Information Retrieval},
publisher = {arXiv},
year = {2021},
copyright = {Creative Commons Attribution Non Commercial Share Alike 4.0 International}
}
@inproceedings{10.1145/3477495.3531857,
author = {Formal, Thibault and Lassance, Carlos and Piwowarski, Benjamin and Clinchant, St'{e}phane},
title = {From Distillation to Hard Negative Sampling: Making Sparse Neural IR Models More Effective},
year = {2022},
isbn = {9781450387323},
publisher = {Association for Computing Machinery},
address = {New York, NY, USA},
url = {https://doi.org/10.1145/3477495.3531857},
doi = {10.1145/3477495.3531857},
abstract = {Neural retrievers based on dense representations combined with Approximate Nearest Neighbors search have recently received a lot of attention, owing their success to distillation and/or better sampling of examples for training -- while still relying on the same backbone architecture. In the meantime, sparse representation learning fueled by traditional inverted indexing techniques has seen a growing interest, inheriting from desirable IR priors such as explicit lexical matching. While some architectural variants have been proposed, a lesser effort has been put in the training of such models. In this work, we build on SPLADE -- a sparse expansion-based retriever -- and show to which extent it is able to benefit from the same training improvements as dense models, by studying the effect of distillation, hard-negative mining as well as the Pre-trained Language Model initialization. We furthermore study the link between effectiveness and efficiency, on in-domain and zero-shot settings, leading to state-of-the-art results in both scenarios for sufficiently expressive models.},
booktitle = {Proceedings of the 45th International ACM SIGIR Conference on Research and Development in Information Retrieval},
pages = {2353–2359},
numpages = {7},
keywords = {neural networks, indexing, sparse representations, regularization},
location = {Madrid, Spain},
series = {SIGIR '22}
}
@inproceedings{10.1145/3477495.3531833,
author = {Lassance, Carlos and Clinchant, St'{e}phane},
title = {An Efficiency Study for SPLADE Models},
year = {2022},
isbn = {9781450387323},
publisher = {Association for Computing Machinery},
address = {New York, NY, USA},
url = {https://doi.org/10.1145/3477495.3531833},
doi = {10.1145/3477495.3531833},
abstract = {Latency and efficiency issues are often overlooked when evaluating IR models based on Pretrained Language Models (PLMs) in reason of multiple hardware and software testing scenarios. Nevertheless, efficiency is an important part of such systems and should not be overlooked. In this paper, we focus on improving the efficiency of the SPLADE model since it has achieved state-of-the-art zero-shot performance and competitive results on TREC collections. SPLADE efficiency can be controlled via a regularization factor, but solely controlling this regularization has been shown to not be efficient enough. In order to reduce the latency gap between SPLADE and traditional retrieval systems, we propose several techniques including L1 regularization for queries, a separation of document/query encoders, a FLOPS-regularized middle-training, and the use of faster query encoders. Our benchmark demonstrates that we can drastically improve the efficiency of these models while increasing the performance metrics on in-domain data. To our knowledge, we propose the first neural models that, under the same computing constraints, achieve similar latency (less than 4ms difference) as traditional BM25, while having similar performance (less than 10% MRR@10 reduction) as the state-of-the-art single-stage neural rankers on in-domain data.},
booktitle = {Proceedings of the 45th International ACM SIGIR Conference on Research and Development in Information Retrieval},
pages = {2220–2226},
numpages = {7},
keywords = {splade, sparse representations, latency, information retrieval},
location = {Madrid, Spain},
series = {SIGIR '22}
}
Fühlen Sie sich frei, uns über Twitter oder per E -Mail @ [email protected] zu kontaktieren!
Splade Copyright (C) 2021-Präsenz Naver Corp.
Splade ist unter einer kreativen Commons Attribution-Noncommercial-Sharealike 4.0 International Lizenz lizenziert. (Siehe Lizenz)
Sie sollten zusammen mit dieser Arbeit eine Kopie der Lizenz erhalten haben. Wenn nicht, siehe http://creatvecommons.org/licenses/by-nc-sa/4.0/.