Baik Cblaster dan Clinker sekarang dapat digunakan tanpa instalasi di Cagecat Websver.
Generator figur perbandingan cluster gen
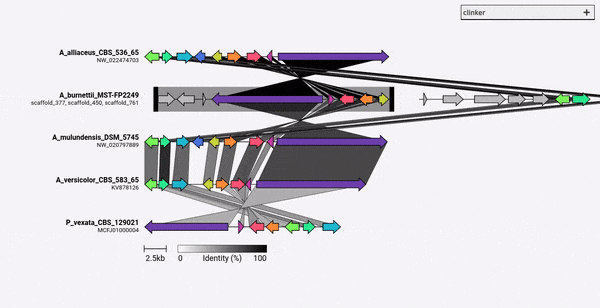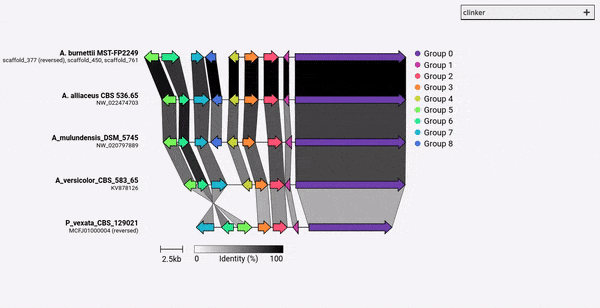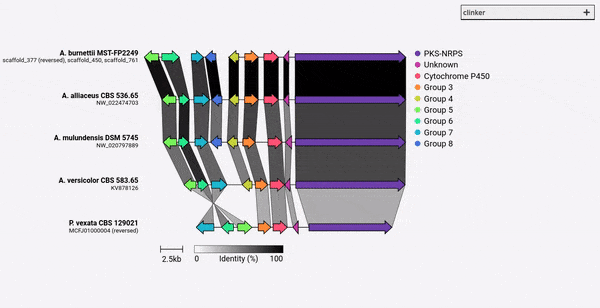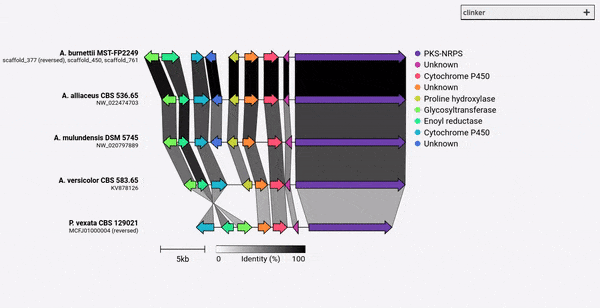
Clinker adalah saluran pipa untuk dengan mudah menghasilkan angka perbandingan kluster gen berkualitas publikasi.
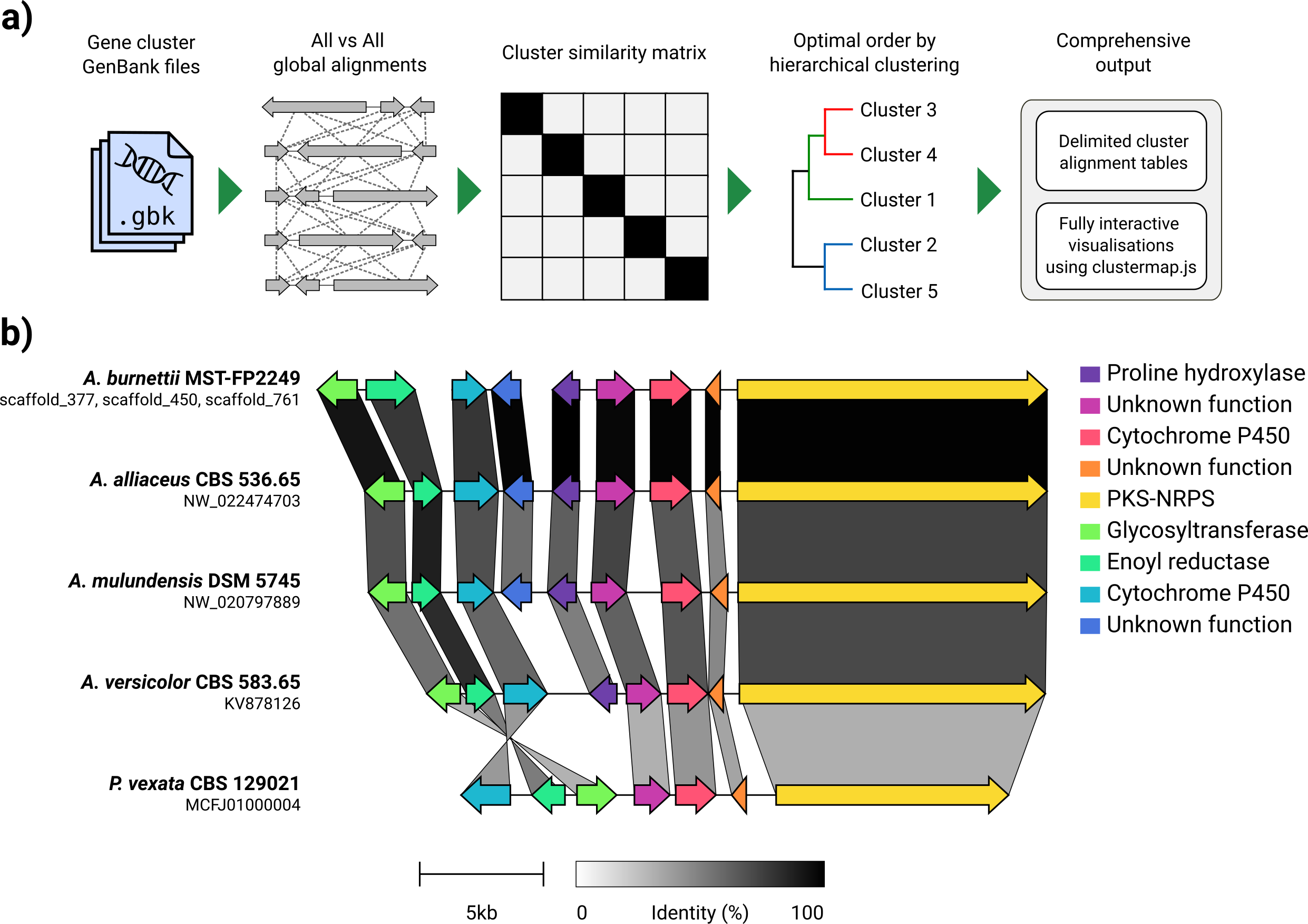
Diberikan satu set file GenBank, klinker akan secara otomatis mengekstrak terjemahan protein, melakukan keberpihakan global antara sekuens di setiap cluster, menentukan urutan tampilan optimal berdasarkan kesamaan cluster, dan menghasilkan visualisasi interaktif (menggunakan clustermap.js) yang dapat diubah secara ekstensif sebelumnya diekspor sebagai file SVG.
Clinker dirancang terutama sebagai cara sederhana untuk memvisualisasikan kelompok kelompok gen biosintetik homolog, yang biasanya merupakan daerah genomik kecil dengan tidak banyak gen (seperti pada contoh GIF). Ini melakukan penyelarasan berpasangan dari semua gen di semua file input menggunakan aligner yang dibangun ke dalam Biopython, kemudian menghasilkan dokumen SVG interaktif di browser. Tahap penyelarasan akan skala sangat buruk untuk beberapa genom dengan banyak gen, dan visualisasi yang dihasilkan juga akan sangat lambat mengingat berapa banyak elemen SVG yang akan dikandungnya. Jika Anda ingin menyelaraskan seluruh genom, Anda mungkin akan lebih baik dilayani menggunakan alat yang dibangun untuk tujuan itu (misalnya Cactus).
Clinker dapat dipasang langsung melalui PIP:
pip install clinker
Dengan mengkloning kode sumber dari GitHub:
git clone https://github.com/gamcil/clinker.git
cd clinker
pip install .
Atau, melalui conda:
conda create -n clinker -c conda-forge -c bioconda clinker-py
conda activate clinker
Jika Anda menemukan Clinker berguna, silakan kutip:
clinker & clustermap.js: Automatic generation of gene cluster comparison figures.
Gilchrist, C.L.M., Chooi, Y.-H., 2020.
Bioinformatics. doi: https://doi.org/10.1093/bioinformatics/btab007
Menjalankan Clinker bisa sesederhana:
clinker clusters/*.gbk
Ini akan dibaca di semua file GenBank di dalam folder, menyelaraskannya, dan mencetak keberpihakan ke terminal. Untuk menghasilkan visualisasi, gunakan argumen -p/--plot :
clinker clusters/*.gbk -p <optional: file name to save static HTML>
Clinker juga dapat mengurai file GFF3:
clinker cluster1.gff3 cluster2.gff3 -p
Catatan: File Fasta yang sesuai dengan nama yang sama (ekstensi ".fa", ".fsa", ".fna", ".fasta" atau ".faa") harus ditemukan di direktori yang sama dengan GFF3, yaitu cluster1.fa dan cluster2.fa .
Lihat -h/--help UNTUK INFORMASI LEBIH LANJUT:
usage: clinker [-h] [--version] [-r RANGES [RANGES ...]] [-gf GENE_FUNCTIONS] [-na] [-i IDENTITY] [-j JOBS] [-s SESSION] [-ji JSON_INDENT] [-f] [-o OUTPUT] [-p [PLOT]] [-dl DELIMITER] [-dc DECIMALS] [-hl] [-ha] [-mo MATRIX_OUT] [-ufo] [files ...]
clinker: Automatic creation of publication-ready gene cluster comparison figures.
clinker generates gene cluster comparison figures from GenBank files. It performs pairwise local or global alignments between every sequence in every unique pair of clusters and generates interactive, to-scale comparison figures using the clustermap.js library.
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
Input options:
files Gene cluster GenBank files
-r RANGES [RANGES ...], --ranges RANGES [RANGES ...]
Scaffold extraction ranges. If a range is specified, only features within the range will be extracted from the scaffold. Ranges should be formatted like: scaffold:start-end (e.g. scaffold_1:15000-40000)
-gf GENE_FUNCTIONS, --gene_functions GENE_FUNCTIONS
2-column CSV file containing gene functions, used to build gene groups from same function instead of sequence similarity (e.g. GENE_001,PKS-NRPS).
Alignment options:
-na, --no_align Do not align clusters
-i IDENTITY, --identity IDENTITY
Minimum alignment sequence identity [default: 0.3]
-j JOBS, --jobs JOBS Number of alignments to run in parallel (0 to use the number of CPUs) [default: 0]
Output options:
-s SESSION, --session SESSION
Path to clinker session
-ji JSON_INDENT, --json_indent JSON_INDENT
Number of spaces to indent JSON [default: none]
-f, --force Overwrite previous output file
-o OUTPUT, --output OUTPUT
Save alignments to file
-p [PLOT], --plot [PLOT]
Plot cluster alignments using clustermap.js. If a path is given, clinker will generate a portable HTML file at that path. Otherwise, the plot will be served dynamically using Python's HTTP server.
-dl DELIMITER, --delimiter DELIMITER
Character to delimit output by [default: human readable]
-dc DECIMALS, --decimals DECIMALS
Number of decimal places in output [default: 2]
-hl, --hide_link_headers
Hide alignment column headers
-ha, --hide_aln_headers
Hide alignment cluster name headers
-mo MATRIX_OUT, --matrix_out MATRIX_OUT
Save cluster similarity matrix to file
Visualisation options:
-ufo, --use_file_order
Display clusters in order of input files
Example usage
-------------
Align clusters, plot results and print scores to screen:
$ clinker files/*.gbk
Only save gene-gene links when identity is over 50%:
$ clinker files/*.gbk -i 0.5
Save an alignment session for later:
$ clinker files/*.gbk -s session.json
Save alignments to file, in comma-delimited format, with 4 decimal places:
$ clinker files/*.gbk -o alignments.csv -dl "," -dc 4
Generate visualisation:
$ clinker files/*.gbk -p
Save visualisation as a static HTML document:
$ clinker files/*.gbk -p plot.html
Cameron Gilchrist, 2020
Secara default, Clinker secara otomatis memberikan nama dan warna untuk setiap kelompok gen homolog. Anda sebaliknya dapat pra-penugasan nama ( -gf/--gene_functions .
GENE_001,Cytochrome P450
GENE_002,Cytochrome P450
GENE_003,Methyltransferase
GENE_004,Methyltransferase
Ini akan menghasilkan dua kelompok, sitokrom P450 (gen_001 dan 002), dan methyltransferase (gen_003, gen_004). Jika ada gen homolog lainnya yang diidentifikasi, mereka akan secara otomatis ditambahkan ke kelompok -kelompok ini.
Pada Clinker V0.0.28, Anda sekarang dapat menentukan warna untuk gen yang ditentukan oleh argumen -gf/--gene_functions . Untuk melakukan ini, gunakan argumen -cm/--colour_map yang juga mengambil file CSV 2-kolom yang berisi nama grup dan kode warna heksadesimal seperti:
Cytochrome P450,#FF0000
Methyltransferase,#0000FF