GPL est une méthode d'adaptation de domaine non supervisée pour la formation de retrievers dense. Il est basé sur la génération de requêtes et le pseudo-étiquetage avec de puissants encodeurs croisés. Pour former un modèle adapté au domaine, il n'a besoin que du corpus cible non étiqueté et peut obtenir une amélioration significative par rapport aux modèles zéro-shot.
Pour plus d'informations, consultez notre publication:
Pour la reproduction, veuillez vous référer à cette branche instantanée.
On peut soit installer GPL via pip
pip install gpl ou via git clone
git clone https://github.com/UKPLab/gpl.git && cd gpl
pip install -e .En attendant, assurez-vous que la version correcte de Pytorch a été installée selon votre version CUDA.
GPL accepte les données dans le format beir. Par exemple, nous pouvons télécharger l'ensemble de données FIQA hébergé par Beir:
wget https://public.ukp.informatik.tu-darmstadt.de/thakur/BEIR/datasets/fiqa.zip
unzip fiqa.zip
head -n 2 fiqa/corpus.jsonl # One can check this data format. Actually GPL only need this `corpus.jsonl` as data input for training. Ensuite, nous pouvons soit utiliser la fonction python -m pour exécuter directement la formation GPL:
export dataset= " fiqa "
python -m gpl.train
--path_to_generated_data " generated/ $dataset "
--base_ckpt " distilbert-base-uncased "
--gpl_score_function " dot "
--batch_size_gpl 32
--gpl_steps 140000
--new_size -1
--queries_per_passage -1
--output_dir " output/ $dataset "
--evaluation_data " ./ $dataset "
--evaluation_output " evaluation/ $dataset "
--generator " BeIR/query-gen-msmarco-t5-base-v1 "
--retrievers " msmarco-distilbert-base-v3 " " msmarco-MiniLM-L-6-v3 "
--retriever_score_functions " cos_sim " " cos_sim "
--cross_encoder " cross-encoder/ms-marco-MiniLM-L-6-v2 "
--qgen_prefix " qgen "
--do_evaluation
# --use_amp # Use this for efficient training if the machine supports AMP
# One can run `python -m gpl.train --help` for the information of all the arguments
# To reproduce the experiments in the paper, set `base_ckpt` to "GPL/msmarco-distilbert-margin-mse" (https://huggingface.co/GPL/msmarco-distilbert-margin-mse)ou importez la méthode en entraînement de GPL dans un script Python:
import gpl
dataset = 'fiqa'
gpl . train (
path_to_generated_data = f"generated/ { dataset } " ,
base_ckpt = "distilbert-base-uncased" ,
# base_ckpt='GPL/msmarco-distilbert-margin-mse',
# The starting checkpoint of the experiments in the paper
gpl_score_function = "dot" ,
# Note that GPL uses MarginMSE loss, which works with dot-product
batch_size_gpl = 32 ,
gpl_steps = 140000 ,
new_size = - 1 ,
# Resize the corpus to `new_size` (|corpus|) if needed. When set to None (by default), the |corpus| will be the full size. When set to -1, the |corpus| will be set automatically: If QPP * |corpus| <= 250K, |corpus| will be the full size; else QPP will be set 3 and |corpus| will be set to 250K / 3
queries_per_passage = - 1 ,
# Number of Queries Per Passage (QPP) in the query generation step. When set to -1 (by default), the QPP will be chosen automatically: If QPP * |corpus| <= 250K, then QPP will be set to 250K / |corpus|; else QPP will be set 3 and |corpus| will be set to 250K / 3
output_dir = f"output/ { dataset } " ,
evaluation_data = f"./ { dataset } " ,
evaluation_output = f"evaluation/ { dataset } " ,
generator = "BeIR/query-gen-msmarco-t5-base-v1" ,
retrievers = [ "msmarco-distilbert-base-v3" , "msmarco-MiniLM-L-6-v3" ],
retriever_score_functions = [ "cos_sim" , "cos_sim" ],
# Note that these two retriever model work with cosine-similarity
cross_encoder = "cross-encoder/ms-marco-MiniLM-L-6-v2" ,
qgen_prefix = "qgen" ,
# This prefix will appear as part of the (folder/file) names for query-generation results: For example, we will have "qgen-qrels/" and "qgen-queries.jsonl" by default.
do_evaluation = True ,
# use_amp=True # One can use this flag for enabling the efficient float16 precision
)On peut également se référer à cet exemple de jouet sur Google Colab pour mieux comprendre le fonctionnement du code.
Le flux de travail de GPL est montré comme suit: 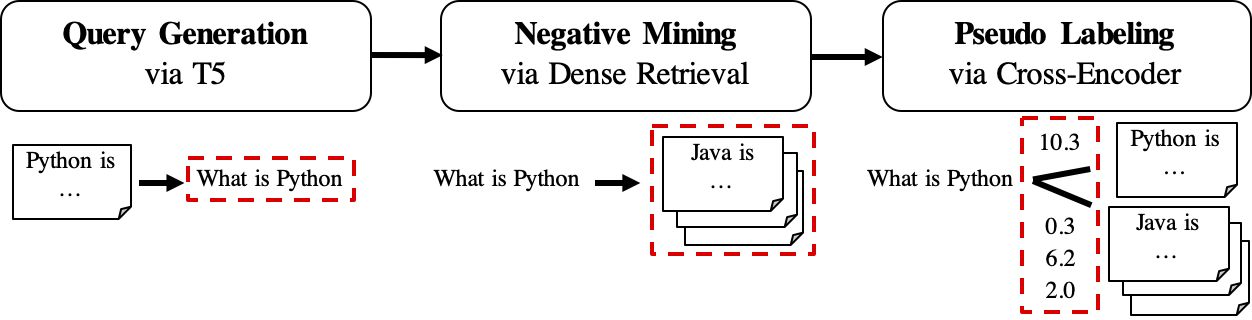
queries_per_passage pour chaque passage du corpus non étiqueté. Les paires de passages de requête sont considérées comme des exemples positifs pour la formation.Fichiers de résultat (sous Path
$path_to_generated_data): (1)${qgen}-qrels/train.tsv, (2)${qgen}-queries.jsonlet également (3)corpus.jsonl(copié à partir de$evaluation_data/);
retrievers d'arguments en tant que mineur négatif.Fichier de résultat (sous Path
$path_to_generated_data): hard-negatif.jsonl;
Fichier de résultat (sous Path
$path_to_generated_data):gpl-training-data.tsv. Il contient des tuples (gpl_steps*batch_size_gpl) au total.
Jusqu'à présent, nous avons les données de formation réelles prêtes. On peut consulter des données d'échantillon / générées / FIQA pour un exemple rapide sur le format de données. La toute dernière étape consiste à appliquer la perte de marginmse pour enseigner à l'étudiant Retriever pour imiter les scores de marge, CE (requête, positif) - CE (requête, négatif) étiqueté par le modèle des enseignants (Cross-Encoder, CE). Et bien sûr, l'étape de marginmse est incluse dans GPL et se fera automatiquement :). Notez que MarginMSE fonctionne avec le produit de points et donc les modèles finaux formés avec GPL fonctionnent avec le produit de points .
PS: Les --retrievers sont destinés à l'extraction négative. Ils peuvent être des retrievers denses formés sur le domaine général (par exemple MS Marco) et n'ont pas besoin d'être forts pour la tâche / domaine cible . Veuillez vous référer au document pour plus de détails (cf. Tableau 7).
On peut également remplacer / placer les données personnalisées pour toute étape intermédiaire sous le chemin de chemin $path_to_generated_data avec la même mode. GPL ignorera les étapes intermédiaires en utilisant ces données fournies.
En tant que flux de travail typique, on ne peut avoir que le corpus (anglais) et vouloir un bon modèle bien performé pour ce corpus. Pour suivre une formation GPL dans une telle condition, il suffit de ces étapes:
corpus.jsonl sous un dossier, par exemple, nommé «généré» pour le chargement des données et la génération de données par GPL;python -m gpl.train
--path_to_generated_data " generated "
--output_dir " output "
--new_size -1
--queries_per_passage -1Nous publions maintenant les modèles GPL pré-formés via le https://huggingface.co/gpl. Il existe actuellement cinq types de modèles:
GPL/${dataset}-msmarco-distilbert-gpl : modèle avec ordre de formation de (1) marginmse sur msmarco -> (2) gpl sur ${dataset} ;GPL/${dataset}-tsdae-msmarco-distilbert-gpl : modèle avec ordre de formation de (1) tsdae sur ${dataset} -> (2) marginmse sur msmarco -> (3) gpl on ${dataset} ;GPL/msmarco-distilbert-margin-mse : Modèle formé sur MSMARCO avec MarginMSE;GPL/${dataset}-tsdae-msmarco-distilbert-margin-mse : modèle avec ordre de formation de (1) tsdae sur $ {ensemble de données} -> (2) marginmse sur msmarco;GPL/${dataset}-distilbert-tas-b-gpl-self_miner : à partir du modèle TAS-B, les modèles ont été formés avec GPL sur le corpus cible ${dataset} avec le modèle de base lui-même comme le mineur négatif (ici noté "self_min"). Les modèles 1. Et 2. Ont été en fait formés au-dessus des modèles 3. Et 4. Resp. Tous les modèles GPL ont été formés le paramètre automatique de new_size et queries_per_passage (en les définissant sur -1 ). Ce paramètre automatique peut maintenir les performances tout en étant efficaces. Pour plus de détails, veuillez vous référer à la section 4.1 dans le document.
Parmi ces modèles, GPL/${dataset}-distilbert-tas-b-gpl-self_miner les meilleurs fonctionnent le mieux sur la référence Beir: 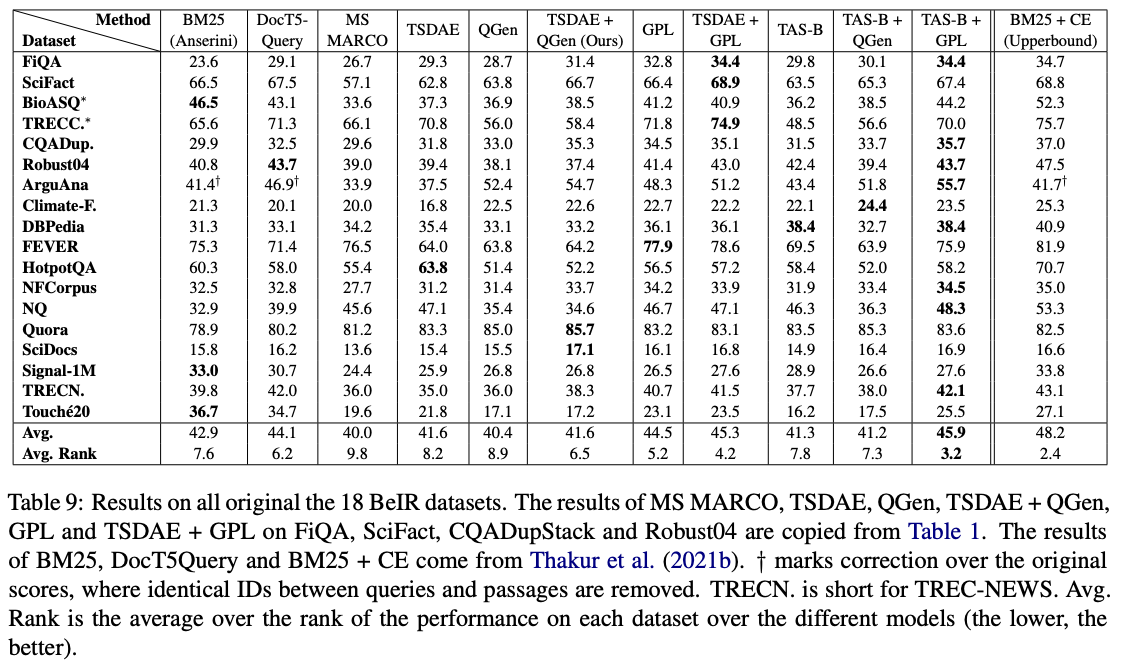
Pour reproduire les résultats avec les mêmes versions de package utilisées dans les expériences, veuillez vous référer au fichier environnemental Conda, Environment.yml.
Nous libérons maintenant les données générées utilisées dans les expériences du papier GPL:
Veuillez noter que les 4 ensembles de données de bioasq , robust04 , trec-news et signal1m ne sont disponibles qu'après l'enregistrement avec les autorités officielles d'origine. Nous publions uniquement les ID de document pour ces corpus avec le nom de fichier corpus.doc_ids.txt . Pour plus de détails, veuillez vous référer au référentiel Beir.
Si vous utilisez le code pour l'évaluation, n'hésitez pas à citer notre publication GPL: étiquetage de pseudo génératif pour l'adaptation de domaine non supervisée de la récupération dense:
@article { wang2021gpl ,
title = " GPL: Generative Pseudo Labeling for Unsupervised Domain Adaptation of Dense Retrieval " ,
author = " Kexin Wang and Nandan Thakur and Nils Reimers and Iryna Gurevych " ,
journal = " arXiv preprint arXiv:2112.07577 " ,
month = " 4 " ,
year = " 2021 " ,
url = " https://arxiv.org/abs/2112.07577 " ,
}Personne de contact et contributeur principal: Kexin Wang, [email protected]
https://www.ukp.tu-darmstadt.de/
https://www.tu-darmstadt.de/
N'hésitez pas à nous envoyer un e-mail ou à signaler un problème, si quelque chose est cassé (et il ne devrait pas l'être) ou si vous avez d'autres questions.
Ce référentiel contient des logiciels expérimentaux et est publié dans le seul but de donner des détails de fond supplémentaires sur la publication respective.