GPL ist eine unbeaufsichtigte Domänenanpassungsmethode für die Schulung dichter Retriever. Es basiert auf der Erzeugung von Abfragen und einer Pseudo-Kennzeichnung mit leistungsstarken Cross-Codern. Um ein domänen angepasster Modell zu trainieren, benötigt es nur das unbeschriebene Zielkorpus und kann eine signifikante Verbesserung gegenüber Null-Shot-Modellen erzielen.
Weitere Informationen finden Sie in unserer Veröffentlichung:
Für die Fortpflanzung finden Sie in diesem Snapshot -Zweig.
Man kann entweder GPL über pip installieren
pip install gpl oder über git clone
git clone https://github.com/UKPLab/gpl.git && cd gpl
pip install -e .Bitte stellen Sie bitte sicher, dass die richtige Version von Pytorch gemäß Ihrer CUDA -Version installiert wurde.
GPL akzeptiert Daten im Beir-Format. Zum Beispiel können wir den von BIRREIN gehosteten FIQA -Datensatz herunterladen:
wget https://public.ukp.informatik.tu-darmstadt.de/thakur/BEIR/datasets/fiqa.zip
unzip fiqa.zip
head -n 2 fiqa/corpus.jsonl # One can check this data format. Actually GPL only need this `corpus.jsonl` as data input for training. Dann können wir entweder die python -m -Funktion verwenden, um das GPL -Training direkt auszuführen:
export dataset= " fiqa "
python -m gpl.train
--path_to_generated_data " generated/ $dataset "
--base_ckpt " distilbert-base-uncased "
--gpl_score_function " dot "
--batch_size_gpl 32
--gpl_steps 140000
--new_size -1
--queries_per_passage -1
--output_dir " output/ $dataset "
--evaluation_data " ./ $dataset "
--evaluation_output " evaluation/ $dataset "
--generator " BeIR/query-gen-msmarco-t5-base-v1 "
--retrievers " msmarco-distilbert-base-v3 " " msmarco-MiniLM-L-6-v3 "
--retriever_score_functions " cos_sim " " cos_sim "
--cross_encoder " cross-encoder/ms-marco-MiniLM-L-6-v2 "
--qgen_prefix " qgen "
--do_evaluation
# --use_amp # Use this for efficient training if the machine supports AMP
# One can run `python -m gpl.train --help` for the information of all the arguments
# To reproduce the experiments in the paper, set `base_ckpt` to "GPL/msmarco-distilbert-margin-mse" (https://huggingface.co/GPL/msmarco-distilbert-margin-mse)oder importieren Sie die Training -Methode von GPL in einem Python -Skript:
import gpl
dataset = 'fiqa'
gpl . train (
path_to_generated_data = f"generated/ { dataset } " ,
base_ckpt = "distilbert-base-uncased" ,
# base_ckpt='GPL/msmarco-distilbert-margin-mse',
# The starting checkpoint of the experiments in the paper
gpl_score_function = "dot" ,
# Note that GPL uses MarginMSE loss, which works with dot-product
batch_size_gpl = 32 ,
gpl_steps = 140000 ,
new_size = - 1 ,
# Resize the corpus to `new_size` (|corpus|) if needed. When set to None (by default), the |corpus| will be the full size. When set to -1, the |corpus| will be set automatically: If QPP * |corpus| <= 250K, |corpus| will be the full size; else QPP will be set 3 and |corpus| will be set to 250K / 3
queries_per_passage = - 1 ,
# Number of Queries Per Passage (QPP) in the query generation step. When set to -1 (by default), the QPP will be chosen automatically: If QPP * |corpus| <= 250K, then QPP will be set to 250K / |corpus|; else QPP will be set 3 and |corpus| will be set to 250K / 3
output_dir = f"output/ { dataset } " ,
evaluation_data = f"./ { dataset } " ,
evaluation_output = f"evaluation/ { dataset } " ,
generator = "BeIR/query-gen-msmarco-t5-base-v1" ,
retrievers = [ "msmarco-distilbert-base-v3" , "msmarco-MiniLM-L-6-v3" ],
retriever_score_functions = [ "cos_sim" , "cos_sim" ],
# Note that these two retriever model work with cosine-similarity
cross_encoder = "cross-encoder/ms-marco-MiniLM-L-6-v2" ,
qgen_prefix = "qgen" ,
# This prefix will appear as part of the (folder/file) names for query-generation results: For example, we will have "qgen-qrels/" and "qgen-queries.jsonl" by default.
do_evaluation = True ,
# use_amp=True # One can use this flag for enabling the efficient float16 precision
)Man kann sich auch auf dieses Spielzeugbeispiel in Google Colab beziehen, um besser zu verstehen, wie der Code funktioniert.
Der Workflow von GPL wird wie folgt angezeigt: 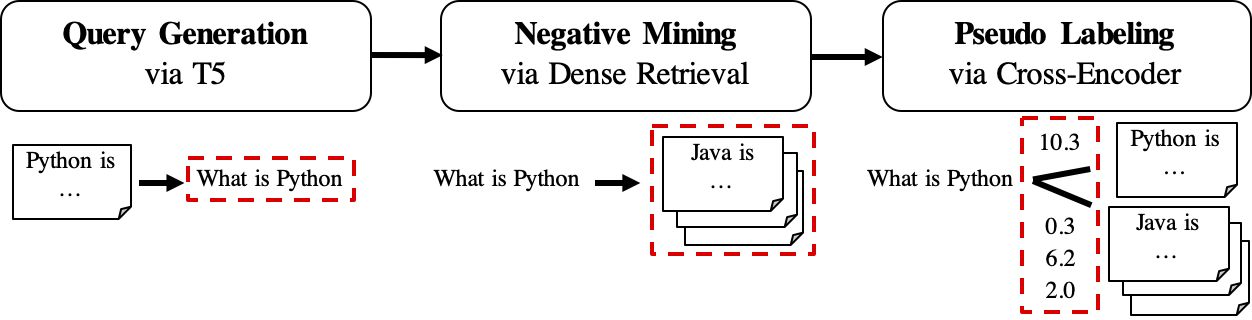
queries_per_passage für jede Passage im unbeschriebenen Korpus zu generieren. Die Abfragepassagepaare werden als positive Beispiele für das Training angesehen.Ergebnisdateien (unter Pfad
$path_to_generated_data): (1)${qgen}-qrels/train.tsv, (2)${qgen}-queries.jsonlund auch (3)corpus.jsonl(kopiert aus$evaluation_data/);
retrievers als negativen Miner.Ergebnisdatei (unter Pfad
$path_to_generated_data): hard-negative.jsonl;
Ergebnisdatei (unter Pfad
$path_to_generated_data):gpl-training-data.tsv. Es enthält insgesamt (gpl_steps*batch_size_gpl) Tupel.
Bisher haben wir die tatsächlichen Trainingsdaten bereit. Man kann die Probendaten/generierte/fiqa für ein kurzes Beispiel zum Datenformat betrachten. Der allerletzte Schritt besteht darin, den Marginmse -Verlust anzuwenden, um dem Schüler Retriever beizubringen, die Margin -Scores, CE (Abfrage, positiv) - CE (Abfrage, negativ), die vom Lehrermodell (Crosscoder, CE) gekennzeichnet ist. Und natürlich ist der Marginmse -Schritt in GPL enthalten und wird automatisch durchgeführt :). Beachten Sie, dass Marginmse mit DOT-Produkt und damit die endgültigen Modelle mit GPL mit DOT-Produkt funktioniert .
PS: Die --retrievers sind für negativen Bergbau. Sie können alle dichten Retrievers sein, die auf der allgemeinen Domäne trainiert werden (z. B. Marco) und müssen für die Zielaufgabe/die Zielaufgabe nicht stark sein . Weitere Informationen finden Sie im Papier (vgl. Tabelle 7).
Man kann die benutzerdefinierten Daten auch für jeden Zwischenschritt unter dem Pfad $path_to_generated_data mit derselben Namensführung ersetzen/setzen. GPL überspringt die Zwischenschritte, indem diese bereitgestellten Daten verwendet werden.
Als typischer Workflow hat man möglicherweise nur den (englischen) unlabelnen Korpus und möchte ein gutes Modell, das für diesen Korpus gut abschneidet. Um das GPL -Training unter solch einer Bedingung durchzuführen, braucht man nur die folgenden Schritte:
corpus.jsonl unter einen Ordner, z.python -m gpl.train
--path_to_generated_data " generated "
--output_dir " output "
--new_size -1
--queries_per_passage -1Wir veröffentlichen nun die vorgeborenen GPL-Modelle über die https://huggingface.co/gpl. Derzeit gibt es fünf Arten von Modellen:
GPL/${dataset}-msmarco-distilbert-gpl : Modell mit Trainingsreihenfolge von (1) marginmse auf msmarco-> (2) GPL auf ${dataset} ;GPL/${dataset}-tsdae-msmarco-distilbert-gpl : Modell mit Trainingsreihenfolge von (1) tsdae auf ${dataset} -> (2) marginmse auf msmarco-> (3) GPL auf ${dataset} ;GPL/msmarco-distilbert-margin-mse : Modell, das auf MSmarco mit Marginmse ausgebildet ist;GPL/${dataset}-tsdae-msmarco-distilbert-margin-mse : Modell mit Trainingsreihenfolge von (1) tsdae auf $ {dataset}-> (2) marginmse auf MSMARCO;GPL/${dataset}-distilbert-tas-b-gpl-self_miner : Ausgehend vom Tas-B-Modell wurden die Modelle mit GPL auf dem Zielkorpus ${dataset} mit dem Basismodell selbst als negativer Miner trainiert (hier als "self_Miner"). Die Modelle 1. und 2. wurden tatsächlich über die Modelle der Modelle 3. und 4. Alle GPL -Modelle wurden durch die automatische Einstellung von new_size und queries_per_passage (indem sie auf -1 festgelegt) geschult. Diese automatische Einstellung kann die Leistung behalten und gleichzeitig effizient sind. Weitere Informationen finden Sie im Abschnitt 4.1 im Papier.
Unter diesen Modellen funktioniert GPL/${dataset}-distilbert-tas-b-gpl-self_miner am besten für den BEIR-Benchmark: 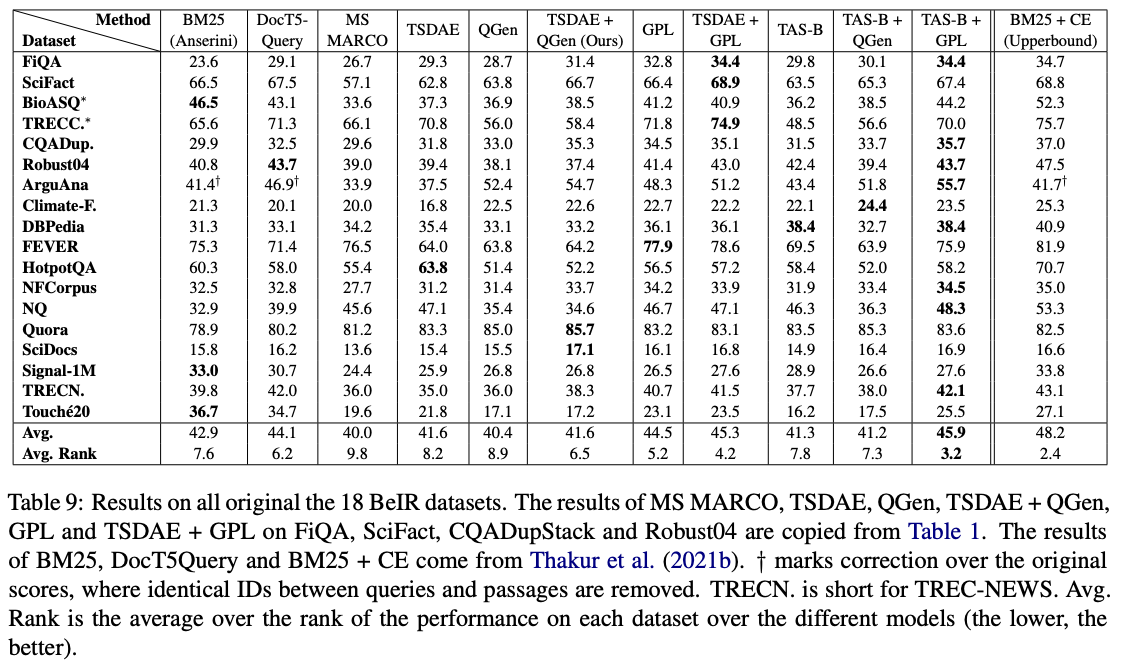
Um die Ergebnisse mit denselben Paketversionen zu reproduzieren, die in den Experimenten verwendet werden, finden Sie in der Conda Environment -Datei Environment.yml.
Wir geben nun die generierten Daten frei, die in den Experimenten des GPL -Papiers verwendet werden:
Bitte beachten Sie, dass die 4 Datensätze von bioasq , robust04 , trec-news und signal1m erst nach der Registrierung bei den ursprünglichen offiziellen Behörden verfügbar sind. Wir veröffentlichen nur die Dokument -IDs für diese Korpora mit dem Dateinamen corpus.doc_ids.txt . Weitere Informationen finden Sie im Beir -Repository.
Wenn Sie den Code für die Bewertung verwenden, können Sie unsere Veröffentlichung GPL: Generative Pseudo -Kennzeichnung für eine unbeaufsichtigte Domänenanpassung des dichten Abrufs zitieren:
@article { wang2021gpl ,
title = " GPL: Generative Pseudo Labeling for Unsupervised Domain Adaptation of Dense Retrieval " ,
author = " Kexin Wang and Nandan Thakur and Nils Reimers and Iryna Gurevych " ,
journal = " arXiv preprint arXiv:2112.07577 " ,
month = " 4 " ,
year = " 2021 " ,
url = " https://arxiv.org/abs/2112.07577 " ,
}Kontaktperson und Hauptmitarbeiter: Kexin Wang, [email protected]
https://www.ukp.tu-darmstadt.de/
https://www.tu-darmstadt.de/
Zögern Sie nicht, uns eine E-Mail zu senden oder ein Problem zu melden, wenn etwas kaputt ist (und dies nicht sein sollte) oder wenn Sie weitere Fragen haben.
Dieses Repository enthält experimentelle Software und wird für den einzigen Zweck veröffentlicht, zusätzliche Hintergrunddetails zur jeweiligen Veröffentlichung anzugeben.