radian
1.0.0
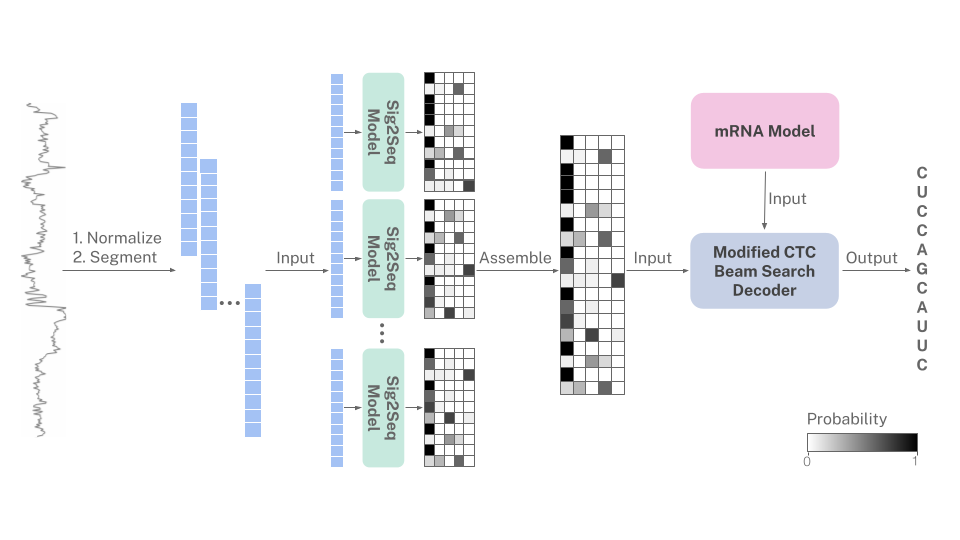
r na l a nguage informe d Decod i ng n a n a nopore sig n als
使用mRNA語言模型的納米孔直接RNA BaseCaller。
由於RNA始終從3'至5'方向進行測序,因此納米孔信號隱式編碼mRNA中的核苷酸偏置。當信號預測模棱兩可時,該基本符號使用人類mRNA語言的概率模型來指導基本。 mRNA模型納入了修改的CTC梁搜索解碼算法中。
預印本:https://www.biorxiv.org/content/10.1101/2022.10.19.512968V1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
我們提供一個Fast5文件,其中包含5個讀取數據/reads.fast5中的讀取。
basecall in in <out_dir>中的單個或多-Fast5文件:
cd radian
mkdir out_dir
python3 basecall.py data out_dir