radian
1.0.0
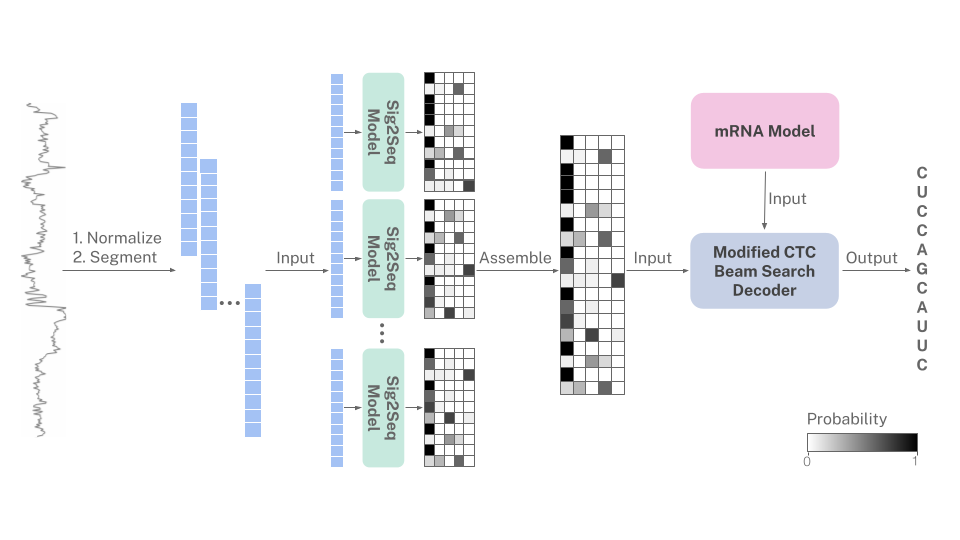
r na l a nguage informe d decod i ng ของ n a nopore sig n als
Nanopore Direct RNA basecaller ที่ใช้แบบจำลองของภาษา mRNA
เนื่องจาก RNA นั้นถูกจัดลำดับจากทิศทาง 3 'ถึง 5' เสมอสัญญาณนาโนโพรงจะเข้ารหัสอคตินิวคลีโอไทด์โดยปริยายใน mRNA basecaller นี้ใช้แบบจำลองความน่าจะเป็นของภาษา mRNA ของมนุษย์เพื่อเป็นแนวทางในการเรียกเก็บเงินเมื่อการทำนายสัญญาณไม่ชัดเจน โมเดล mRNA นั้นรวมอยู่ในอัลกอริทึมการถอดรหัสลำแสง CTC ที่แก้ไขแล้ว
preprint : https://www.biorxiv.org/content/10.1101/2022.10.19.512968V1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
เราจัดเตรียมไฟล์ Fast5 ที่มีการอ่าน 5 ครั้งสำหรับการทดสอบใน data/reads.fast5
หากต้องการ basecall ไฟล์เดียวหรือ multi-fast5 ในและส่งออก fasta เป็น <out_dir>::
cd radian
mkdir out_dir
python3 basecall.py data out_dir