R na l nguage informe d decod ng n a nopore sig n als
Nanopore Direct RNA BaseCaller, который использует модель языка мРНК.
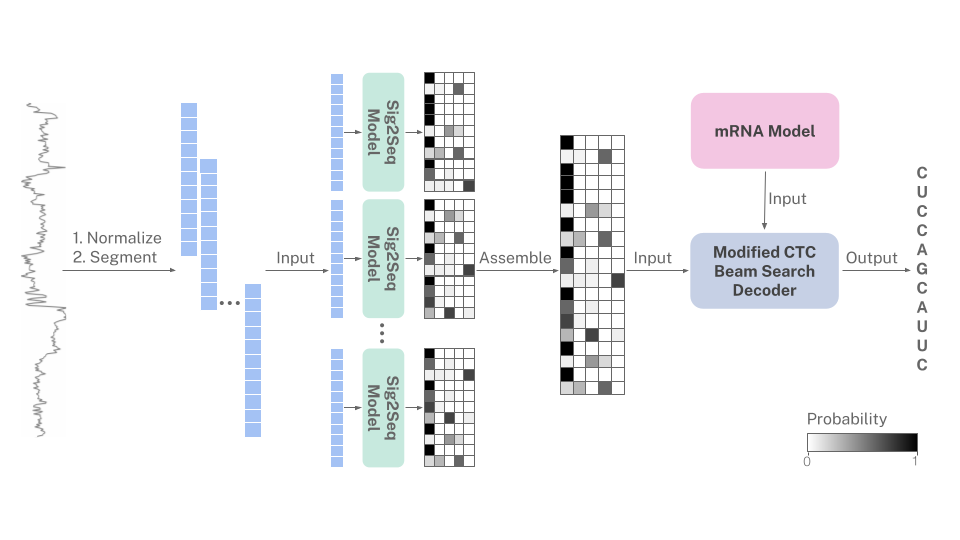
Поскольку РНК всегда секвенирована от 3 'до 5 -дюймового направления, сигналы нанопор неявно кодируют смещения нуклеотидов в мРНК. Этот базовый ролик использует вероятностную модель языка мРНК человека, чтобы направлять базовый счет, когда прогнозирование сигнала является неоднозначным. Модель мРНК включена в модифицированный алгоритм декодирования поиска луча CTC.
Прекрата : https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
Мы предоставляем файл FAST5, содержащий 5 чтения для тестирования в данных/reads.fast5.
Чтобы обосновать отдельные или многобыточные файлы (ы) в и вывод и вывод Fasta to <put_dir>:
cd radian
mkdir out_dir
python3 basecall.py data out_dir