radian
1.0.0
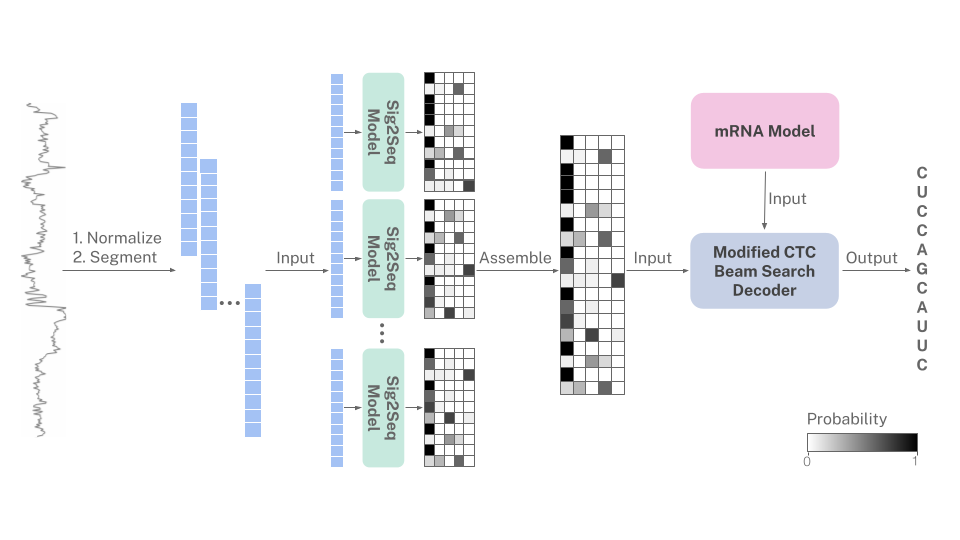
r na l a nguageは、n a nopore sig n alsのデコードを情報提供
mRNA言語のモデルを利用するナノポア直接RNAベースカラー。
RNAは常に3 'から5'の方向から配列決定されるため、ナノポアはmRNAのヌクレオチドバイアスを暗黙的にエンコードします。このbasecallerは、シグナル予測が曖昧なときにベースコールを導くために、人間のmRNA言語の確率論的モデルを使用します。 mRNAモデルは、変更されたCTCビーム検索デコードアルゴリズムに組み込まれています。
プリプリント:https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
データ/reads.fast5でテストするために5つの読み取りを含むFast5ファイルを提供します。
単一またはMulti-Fast5ファイルを<out_dir>に出力し、出力します。
cd radian
mkdir out_dir
python3 basecall.py data out_dir