radian
1.0.0
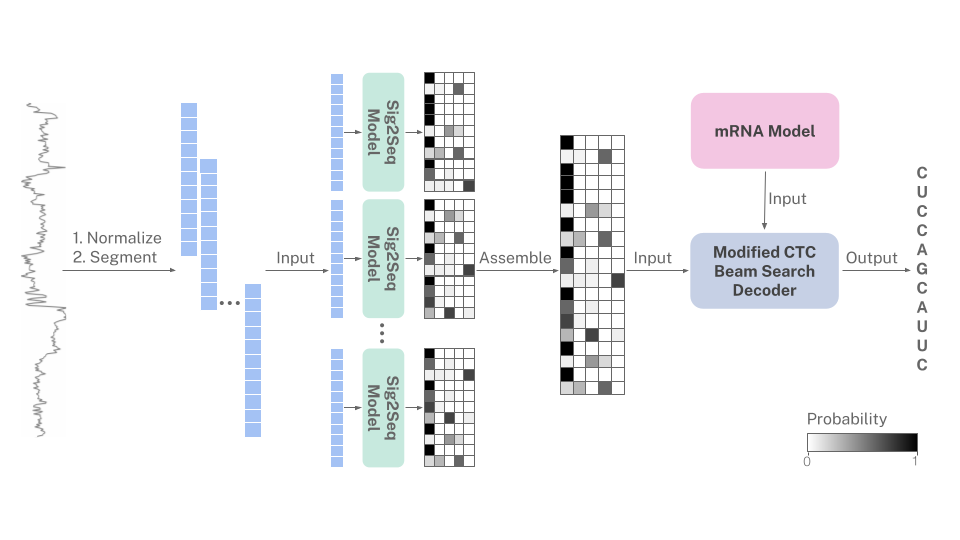
R na l a nguage informe d decod i ng of n a nopore sig n als
Nanopore Direct RNA Basecaller yang menggunakan model bahasa mRNA.
Karena RNA selalu diurutkan dari arah 3 'hingga 5', sinyal nanopore secara implisit mengkodekan bias nukleotida dalam mRNA. Basecaller ini menggunakan model probabilistik bahasa mRNA manusia untuk memandu basis ketika prediksi sinyal ambigu. Model mRNA dimasukkan dalam algoritma decoding pencarian balok CTC yang dimodifikasi.
Preprint : https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
Kami menyediakan file FAST5 yang berisi 5 bacaan untuk pengujian dalam data/bacaan.fast5.
Untuk membasmi file tunggal atau multi-fast5 di dan output fasta ke <out_dir>:
cd radian
mkdir out_dir
python3 basecall.py data out_dir