R na l a nguage informe d decod i ng von n a nopore sig n als
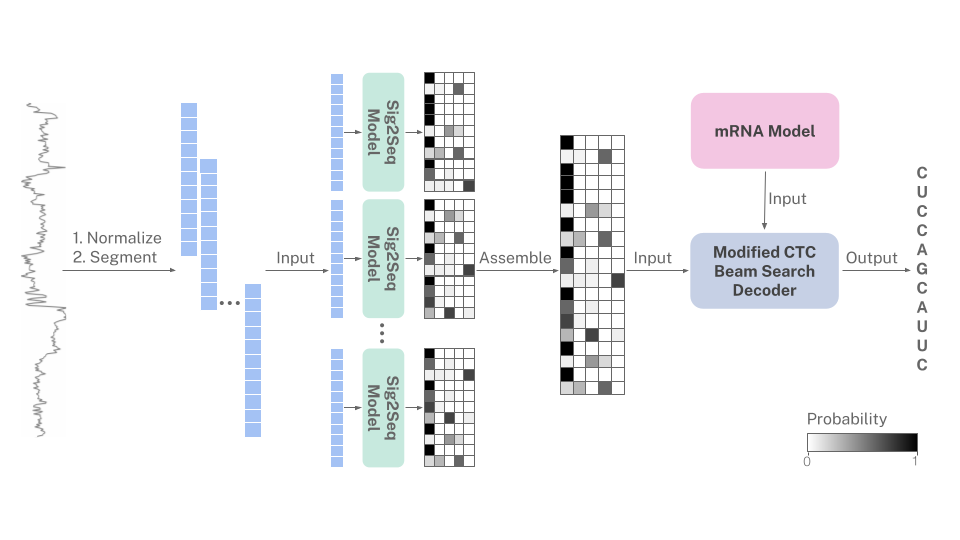
Nanopore Direct RNA Basecaller, die ein Modell der mRNA -Sprache verwendet.
Da RNA immer von der 3 'auf 5' Richtung sequenziert wird, codieren Nanoporensignale implizit die Nucleotid -Verzerrungen in mRNA. Dieser Basiszusatz verwendet ein probabilistisches Modell der menschlichen mRNA -Sprache, um das Basisuntersuchung zu steuern, wenn die Signalvorhersage nicht eindeutig ist. Das mRNA -Modell ist in einen modifizierten CTC -Strahl -Such -Dekodierungsalgorithmus aufgenommen.
Preprint : https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
Wir bieten eine Fast5 -Datei mit 5 Lesevorgängen zum Testen in Daten/Reads.fast5.
Um die einzelne oder mehrfaste 5-Datei (en) in FASTA zu basieren und zu <out_dir> auszugeben:
cd radian
mkdir out_dir
python3 basecall.py data out_dir