R Na L a Nguage Informated D Decody ING OF N A NOPORE SIG N ALS
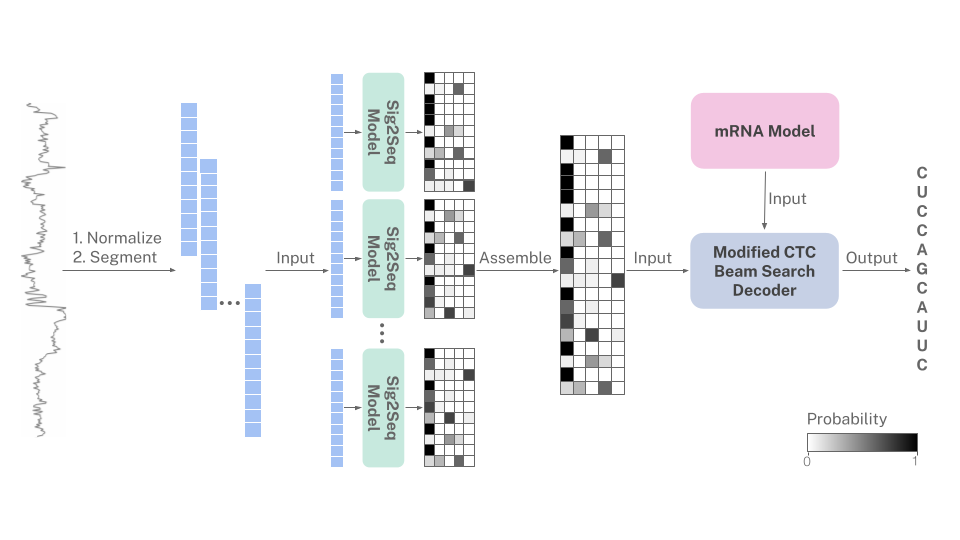
O Nanopore Direct RNA Basecaller que utiliza um modelo de linguagem de mRNA.
Como o RNA é sempre sequenciado da direção 3 'a 5', os sinais de nanoporos codificam implicitamente os vieses de nucleotídeos no mRNA. Este Basecaller usa um modelo probabilístico da linguagem do mRNA humano para orientar a base de base quando a previsão do sinal é ambígua. O modelo de mRNA é incorporado em um algoritmo de decodificação de pesquisa de feixe CTC modificado.
Preprint : https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
Fornecemos um arquivo Fast5 contendo 5 leituras para testes em dados/leituras.fast5.
Para secar o (s) arquivo (s) único ou multi-Fast5, em e sair da fase para <O out_Dir>:
cd radian
mkdir out_dir
python3 basecall.py data out_dir