R na l a nguage informe d décod i ng of n a nopore sig n als
Nanopore Direct ARN Basaller qui utilise un modèle de langage d'ARNm.
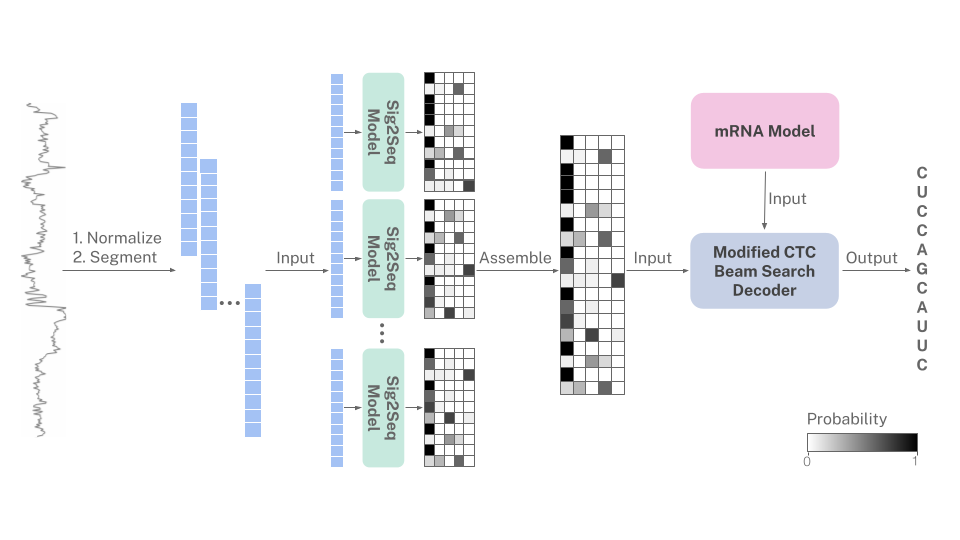
Étant donné que l'ARN est toujours séquencé de la direction de 3 'à 5', les signaux de nanopore codent implicitement les biais nucléotidiques dans l'ARNm. Ce tarif utilise un modèle probabiliste du langage de l'ARNm humain pour guider l'attracture de base lorsque la prédiction du signal est ambiguë. Le modèle d'ARNm est incorporé dans un algorithme de décodage de recherche de faisceau CTC modifié.
Préimpression : https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
Nous fournissons un fichier Fast5 contenant 5 lectures pour les tests dans Data / ReadS.fast5.
Pour BasCall, le (s) fichier (s) et de sortie Fasta à <out_dir>:
cd radian
mkdir out_dir
python3 basecall.py data out_dir