radian
1.0.0
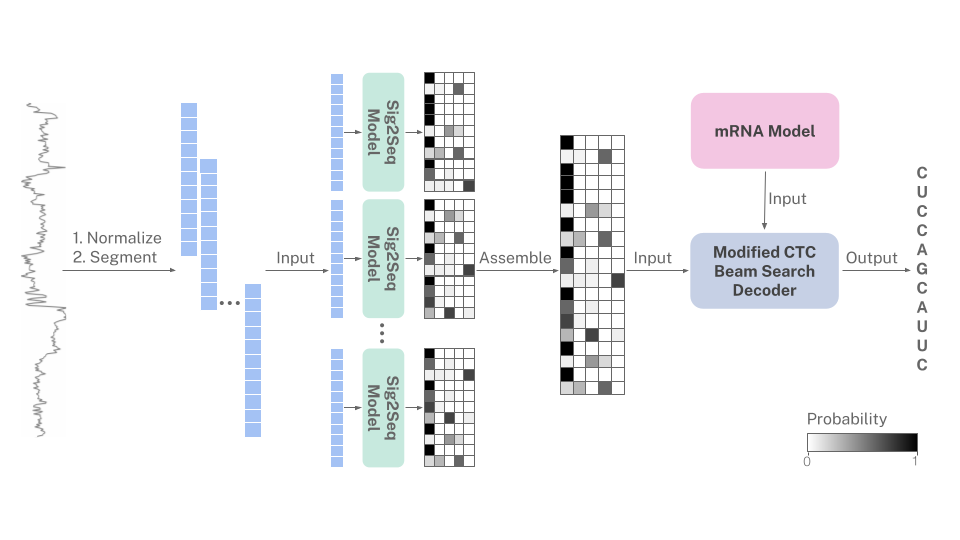
r na l a nguage informe d decod i ng of n a nopore sig n als
Nanopore Direct RNA Basecaller الذي يستخدم نموذج لغة مرنا.
نظرًا لأن الحمض النووي الريبي يتم تسلسله دائمًا من اتجاه 3 إلى 5 "، فإن إشارات النانوبور تشفر ضمنيًا تحيزات النوكليوتيدات في مرنا. يستخدم هذا الأساس نموذجًا احتماليًا للغة الرنا البشرية البشرية لتوجيه الأساس عندما يكون التنبؤ بالإشارة غامضًا. يتم دمج نموذج مرنا في خوارزمية فك تشفير حزمة CTC المعدلة.
preprint : https://www.biorxiv.org/content/10.1101/2022.10.19.512968v1
cd <path/to/radian>
pip install --upgrade pip
pip install -r requirements.txt
tar -xvzf radian/models/rnamodel_12mer_pc.tar.gz
usage: basecall.py [-h] fast5_dir fasta_dir [--local] [--chunk-len] [--step-size]
[--batch-size] [--outlier-clip] [--rna-model]
[--sig-model] [--sig-config] [--beam-width]
[--decode-type] [--sig-threshold]
[--rna-threshold] [--context-len]
positional arguments:
fast5_dir Directory of single/multi fast5 files.
fasta_dir Directory to output fasta files.
optional arguments:
-h, --help
--local
--chunk-len
--step-size
--batch-size
--outlier-clip
--rna-model
--sig-model
--sig-config
--beam-width
--decode-type {global,chunk}
--sig-threshold
--rna-threshold
--context-len
نحن نقدم ملف fast5 يحتوي على 5 قراءة للاختبار في البيانات/reads.fast5.
لتصاعد الملف (ملفات) (ملفات) واحدة أو متعددة الصب في وإخراج fasta إلى <out_dir>:
cd radian
mkdir out_dir
python3 basecall.py data out_dir