多任務變壓器將回歸重新定義為條件序列建模任務。這產生了一個二分法模型,該模型將回歸與財產驅動的條件產生無縫整合。
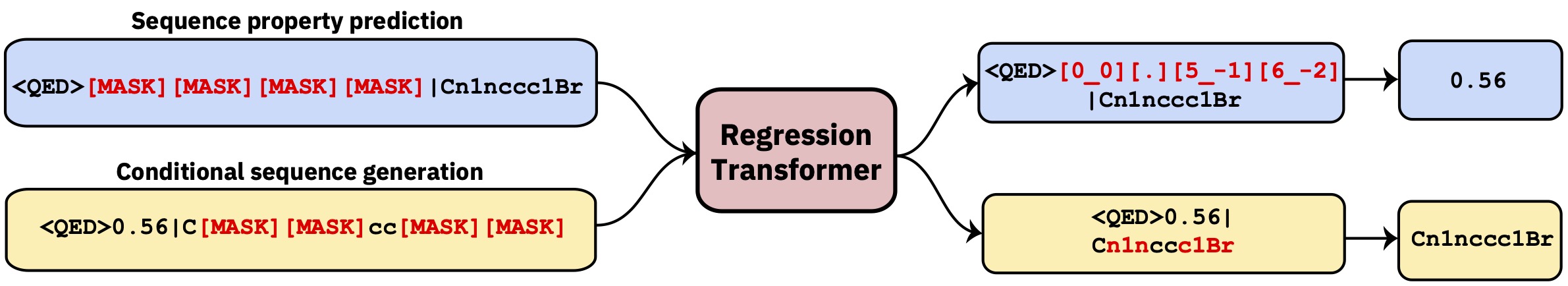
此存儲庫包含開發代碼。閱讀自然機器智能中的論文。
?在擁抱面空間上可以使用帶有簡單UI的Gradio演示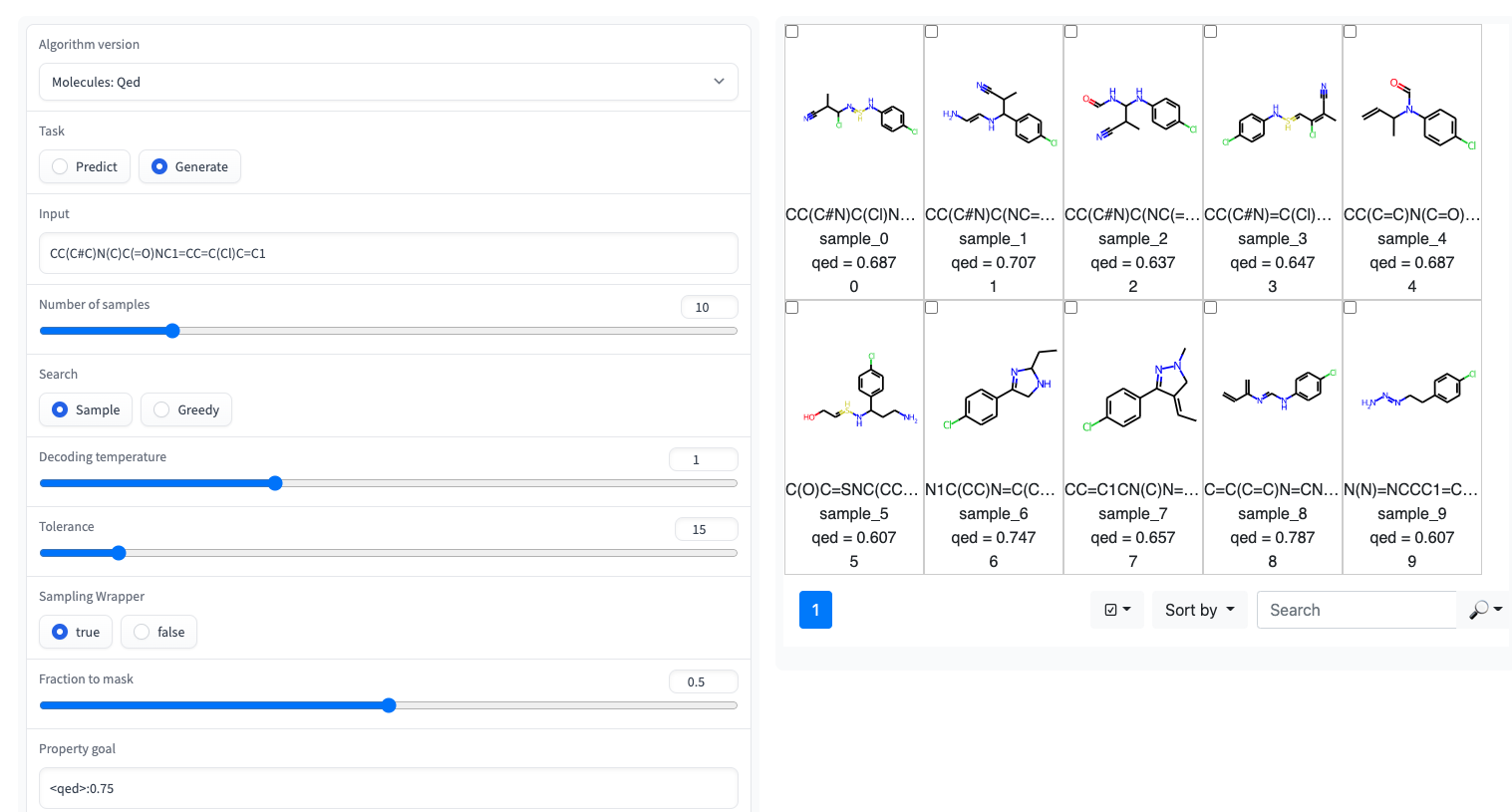
回歸變壓器在GT4SD庫中實現。通過GT4SD,使用幾個驗證的回歸變形物是幾行代碼的問題。可以在此處找到一個完整的跑步推理,對RT模型(或從頭開始訓練)以及將其共享並部署到GT4SD模型中心的教程。
例如,通過GT4SD,您可以在紙張中所示的某些特性上使用的RT使用,尤其是QED和ESOL(水溶性)。 RT也有幾種多層變體:例如,在LOGP和合成性(又稱SCSCORE)的聯合訓練的模型。對於蛋白質語言建模,您還將發現從磁帶基準測試的肽穩定性數據集上訓練的RT。總而言之,GT4SD提供了鑑定的RT模型:
qed , esol , crippen_logp )或多個( logp_and_synthesizability , cosmo_acdl , pfas )屬性。除了使用笑容crippen_logp外,所有這些模型都使用自拍照。stabilityuspto (使用反應微笑)rop_catalyst和block_copolymer均在Park等人(2023; Nature Communications )中描述。 rop_catalyst使用常規自拍照,但block_copolymer模型使用了一種新型聚合物語言,稱為CMDL,在Park等人(2023; Nature Communications )中也描述了。GT4SD也提供了一個帶有玩具用途酶的jupyter筆記本,該筆記本也提供了適應分子的溶解度。如果您使用GT4SD,則可以產生這樣的分子:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 ))探索Buturon周圍局部化學空間的溶解度。改變屬性底漆後,您可能會得到這樣的東西: 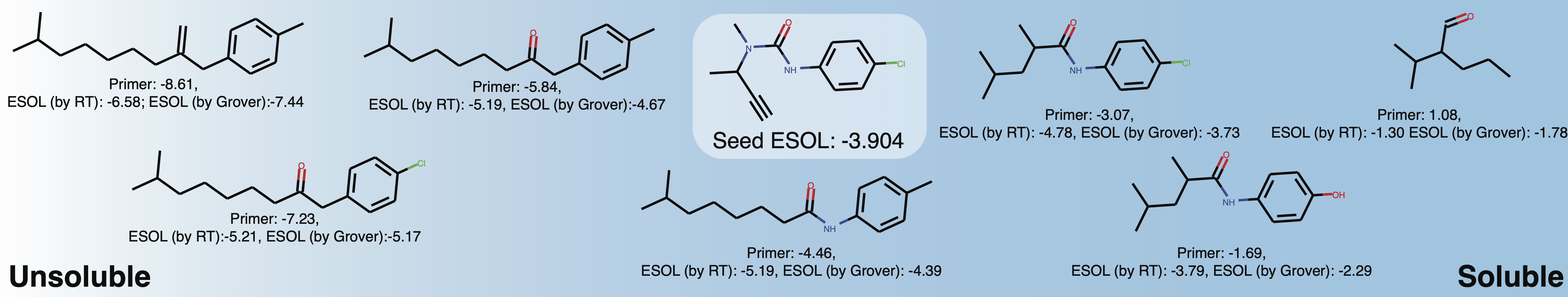
這主要旨在復製或擴展紙張的結果。
conda env create -f conda.yml
conda activate terminator
pip install -e .用於訓練模型的處理數據可通過框獲得。
您可以下載數據並通過指向培訓和測試數據來啟動培訓:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path參數指向指定培訓制度的文件。這是可選的,如果沒有給出該參數,我們默認為均等的香草PLM訓練,以同等的概率掩蓋各地(僅用於初始預處理)。有關精緻的示例,請參閱training_configs文件夾。
另請注意, vocabs夾包含用於小分子,蛋白質和化學反應訓練的詞彙文件。
可以在Configs文件夾中找到示例性的模型配置(頭部,圖層等)。
要評估經過訓練的模型,例如,在QED任務上,運行以下內容:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.json預審計的模型可通過GT4SD模型中心可用。總共有9個型號也可以通過擁抱面空間使用。作為出版物一部分的模型也可以通過上面提到的盒子文件夾獲得。
要以RT兼容格式為QED任務生成自定義數據,請運行腳本/generate_example_data.py,並指向第一列中帶有笑容的.smi文件。
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txt對於用戶定義的屬性,請調整文件或打開問題。
如果您需要為數據集創建一個新的詞彙,則可以使用腳本/create_vocabulary.py。它也將自動在詞彙文件的頂部添加一些特殊令牌。
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt此時,包含詞彙文件的文件夾可用於加載與任何ExpressionBertTokenizer兼容的令牌:
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]如果您使用回歸變壓器,請引用:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}