Un transformateur multitâche qui reformula la régression comme tâche de modélisation de séquence conditionnelle. Cela donne un modèle de langue dichotomique qui intègre de manière transparente la régression à la génération conditionnelle axée sur la propriété.
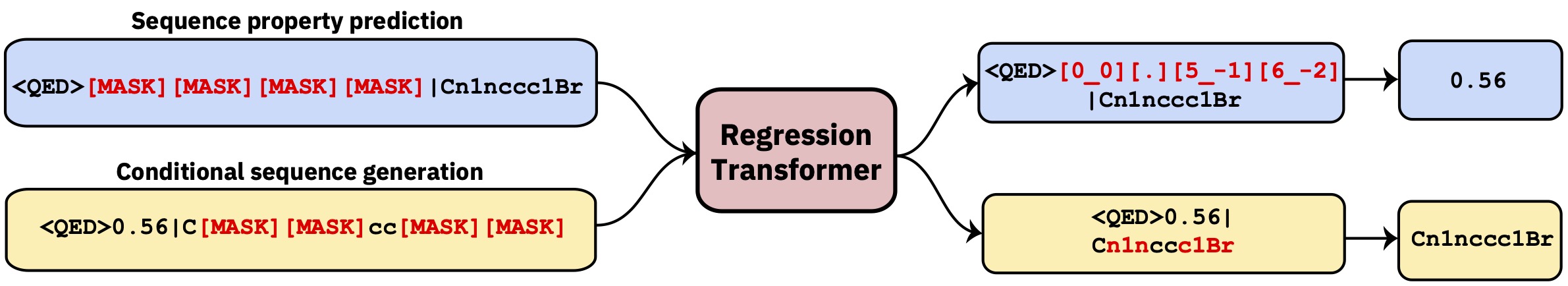
Ce repo contient le code de développement. Lisez le papier dans Nature Machine Intelligence .
? Une démo Gradio avec une interface utilisateur simple est disponible sur les espaces de câlins 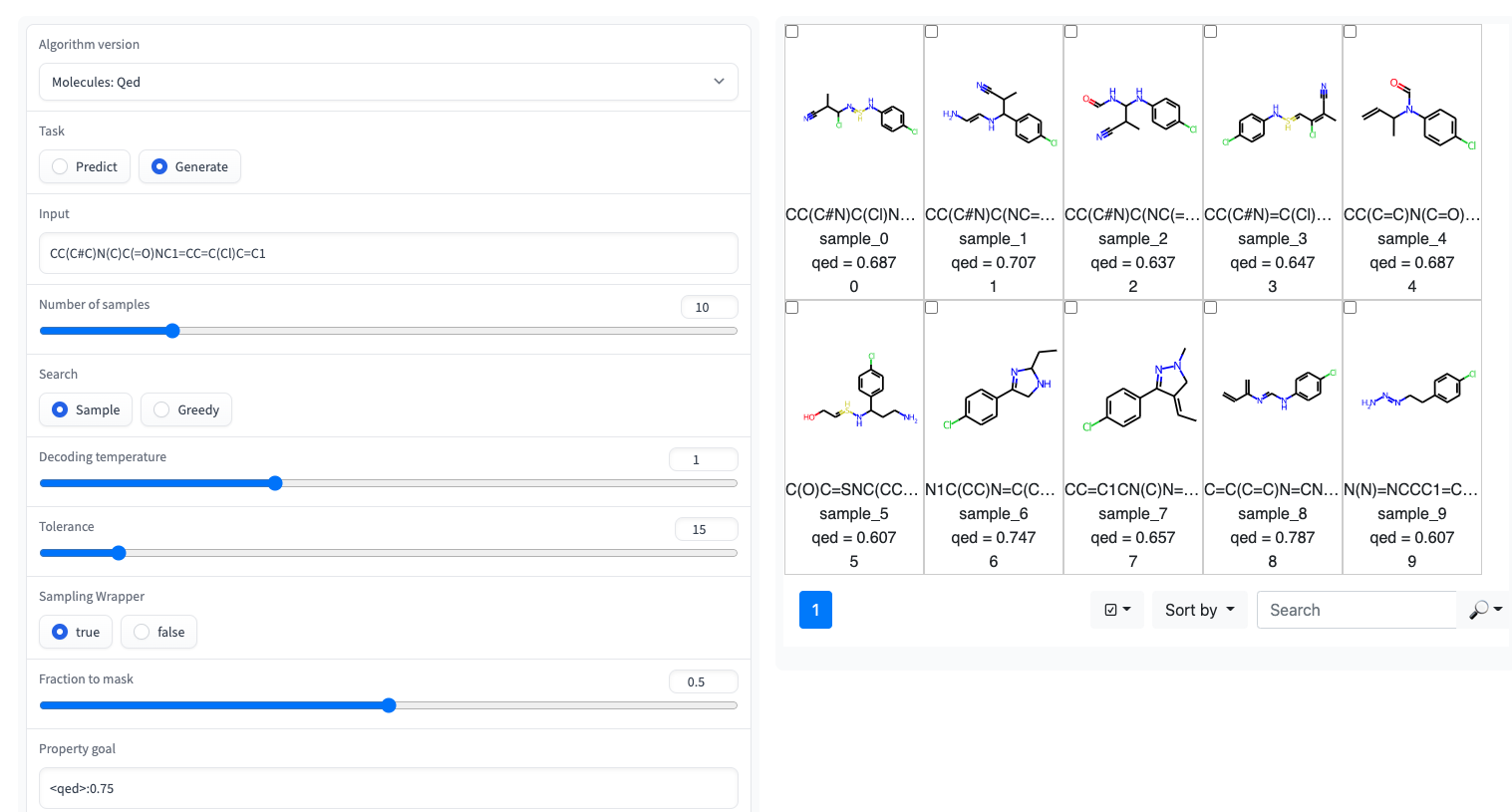
Le transformateur de régression est implémenté dans la bibliothèque GT4SD. Via GT4SD, l'utilisation de plusieurs transformateurs de régression pré-entraînés est une question de quelques lignes de code. Un tutoriel complet de l'exécution de l'inférence, de la fineturation d'un modèle RT (ou de la formation à partir de zéro) et du partage et du déploiement dans le Hub du modèle GT4SD, peut être trouvé ici.
Par exemple, via GT4SD, vous pouvez utiliser le RT pré-entraîné sur de petites molécules avec certaines propriétés comme indiqué dans le papier, en particulier QED et ESOL (solubilité dans l'eau). Il existe également plusieurs variantes multiproperty du RT: EG, un modèle formé conjointement sur LogP et la synthétisabilité (AKA SCSCORE). Pour la modélisation du langage des protéines, vous trouverez également un RT formé sur un ensemble de données de stabilité des peptides à partir de la référence à bande. En somme, GT4SD fournit des modèles RT pré-entraînés sur:
qed , esol , crippen_logp ) ou multiple ( logp_and_synthesizability , cosmo_acdl , pfas ). Tous ces modèles utilisent des selfies en dehors de crippen_logp qui utilise des sourires.stabilityuspto (en utilisant des sourires de réaction)rop_catalyst et block_copolymer sont tous deux décrits dans Park et al., (2023; Nature Communications ). Le rop_catalyst utilise des selfies conventionnels mais le modèle block_copolymer utilise un nouveau langage polymère appelé CMDL décrit également dans Park et al., (2023; Nature Communications ).Un cahier Jupyter avec un jouet ucase sur l'adaptation d'une molécule à la solubilité est également fourni dans GT4SD. Si vous utilisez GT4SD, vous pouvez générer des molécules comme ceci:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) Explorez la solubilité de l'espace chimique local autour de Buturon. En variant les amorces de propriété, vous pourriez obtenir quelque chose comme ceci: 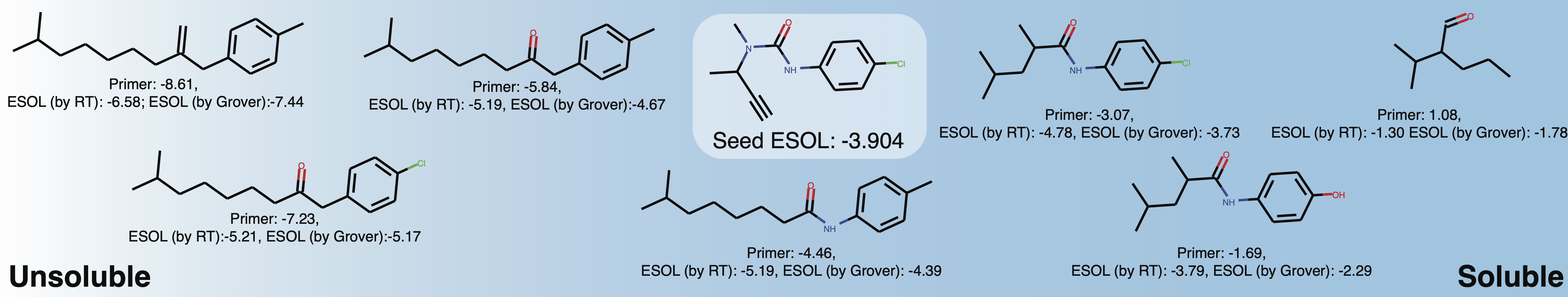
Ceci est principalement destiné à reproduire ou à étendre les résultats du papier.
conda env create -f conda.yml
conda activate terminator
pip install -e .Les données traitées utilisées pour former les modèles sont disponibles via la boîte.
Vous pouvez télécharger les données et lancer une formation en pointant de former et de tester les données:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path pointe vers un fichier qui spécifie le régime de formation. Ceci est facultatif, si l'argument n'est pas donné, nous avons par défaut la formation Vanilla PLM qui masque partout avec une probabilité égale (recommandée pour la prélèvement initial uniquement). Pour des exemples raffinés, veuillez consulter le dossier training_configs .
Notez également que le dossier vocabs contient les fichiers de vocabulaire pour une formation sur les petites molécules, les protéines et les réactions chimiques.
Des configurations de modèle exemplaires (nombre de têtes, couches, etc.) peuvent être trouvées dans le dossier Configs.
Pour évaluer un modèle formé par EG, sur la tâche QED, exécutez ce qui suit:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonLes modèles pré-entraînés sont disponibles via le Hub du modèle GT4SD. Il existe un total de 9 modèles qui peuvent également être utilisés via des espaces HuggingFace. Les modèles qui font partie de la publication sont également disponibles via le dossier de la boîte mentionné ci-dessus.
Pour générer des données personnalisées pour la tâche QED dans un format compatible RT, exécutez les scripts / generate_example_data.py et pointez un fichier .smi avec des sourires dans la première colonne.
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtPour les propriétés définies par l'utilisateur, veuillez adapter le fichier ou ouvrir un problème.
Si vous devez créer un nouveau vocabulaire pour un ensemble de données, vous pouvez utiliser Scripts / Create_vocabulary.py, il ajoutera également automatiquement des jetons spéciaux en haut de votre fichier de vocabulaire.
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt À ce stade, le dossier contenant le fichier de vocabulaire peut être utilisé pour charger un tokenizer compatible avec n'importe quel ExpressionBertTokenizer :
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]Si vous utilisez le transformateur de régression, veuillez citer:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}