Um transformador multitarefa que reformula a regressão como uma tarefa de modelagem de sequência condicional. Isso produz um modelo de linguagem dicotômica que integra perfeitamente a regressão à geração condicional orientada por propriedades.
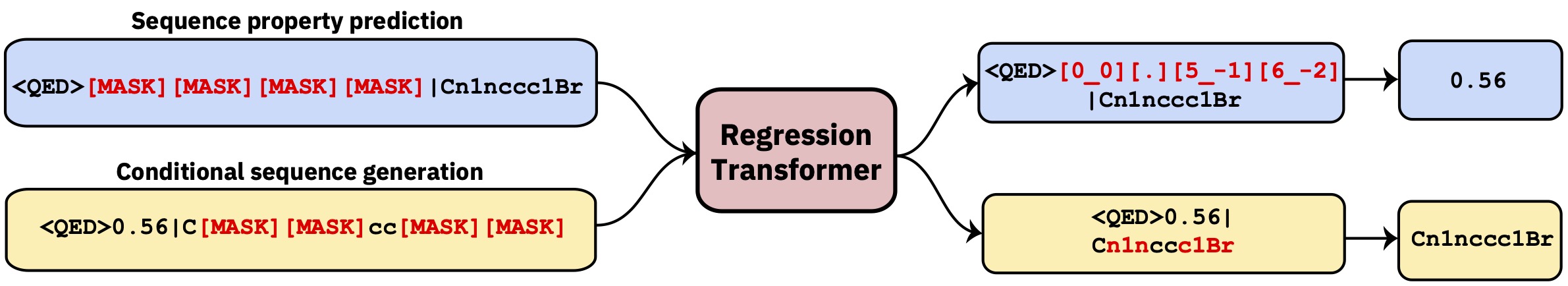
Este repo contém o código de desenvolvimento. Leia o artigo da Nature Machine Intelligence .
? Uma demonstração de graduação com uma interface do usuário simples está disponível em espaços Huggingface 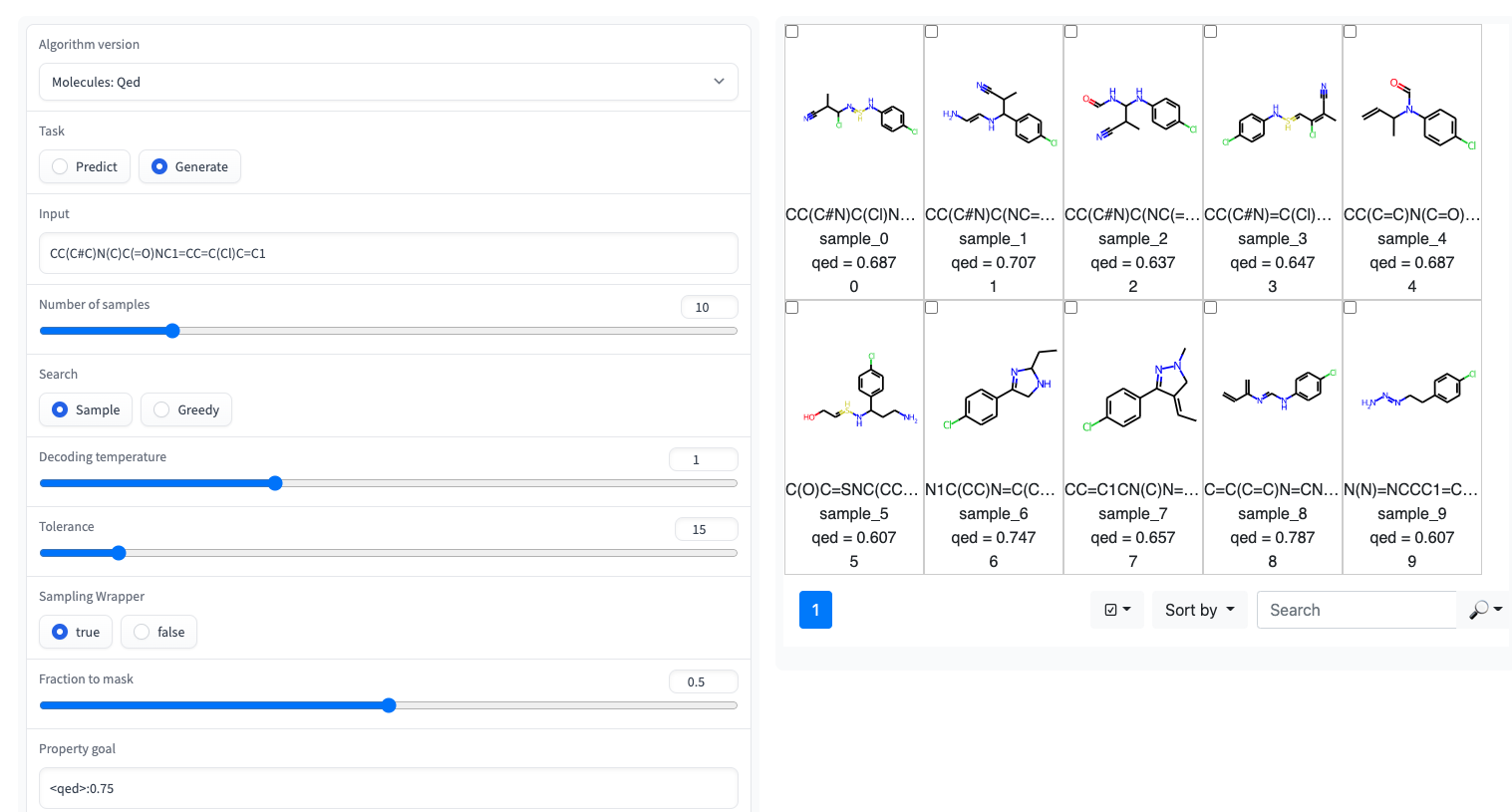
O transformador de regressão é implementado na biblioteca GT4SD. Via GT4SD, usando vários transformadores de regressão pré -traida, é uma questão de algumas linhas de código. Um tutorial completo de inferência de execução, Finetuning um modelo RT (ou treinando -o do zero) e compartilhando e implantando -o no Hub do modelo GT4SD, pode ser encontrado aqui.
Por exemplo, via GT4SD, você pode usar o RT pré -terenciado em pequenas moléculas com algumas propriedades, como mostrado no papel, em particular QED e ESOL (solubilidade em água). Há também várias variantes multiproperty da RT: por exemplo, um modelo treinado em conjunto em LOGP e sintetizabilidade (também conhecido como SCSCore). Para modelagem de linguagem de proteínas, você também encontrará um RT treinado em um conjunto de dados de estabilidade peptídica a partir da referência de fita. Em suma, o GT4SD fornece modelos de RT pré -terenciados em:
qed , esol , crippen_logp ) ou múltiplas ( logp_and_synthesizability , cosmo_acdl , pfas ). Todos esses modelos usam selfies além de crippen_logp , que usa sorrisos.stabilityuspto (usando sorrisos de reação)rop_catalyst e block_copolymer são descritos em Park et al., (2023; Nature Communications ). O rop_catalyst usa selfies convencionais, mas o modelo block_copolymer usa uma nova linguagem de polímero chamada CMDL descrita também em Park et al., (2023; Nature Communications ).Um caderno Jupyter com uma USECASE de brinquedo para adaptar uma molécula à solubilidade também é fornecida no GT4SD. Se você usar o GT4SD, pode gerar moléculas como esta:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) Explore a solubilidade do espaço químico local em torno de Buturon. Ao variar os iniciadores de propriedade, você pode obter algo assim: 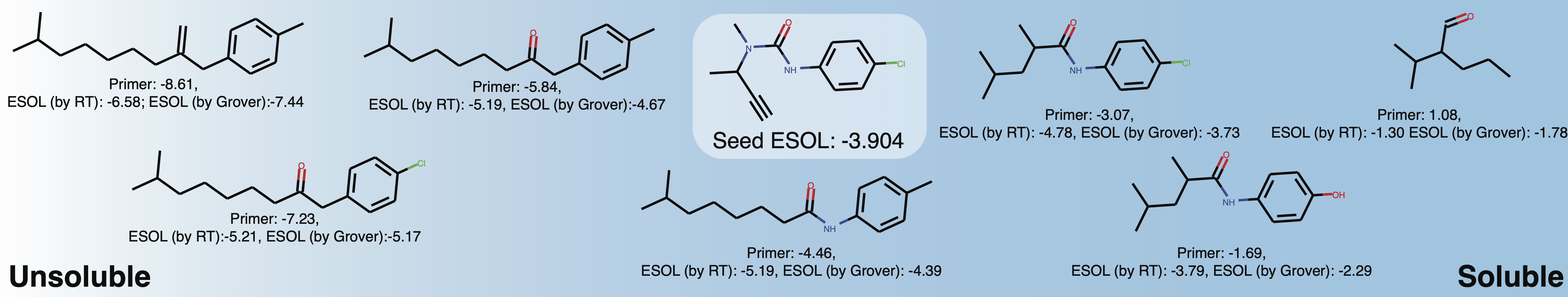
Isso se destina principalmente a reproduzir ou estender os resultados do artigo.
conda env create -f conda.yml
conda activate terminator
pip install -e .Os dados processados usados para treinar os modelos estão disponíveis via caixa.
Você pode baixar os dados e iniciar um treinamento apontando para treinar e testar dados:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path aponta para um arquivo que especifica o regime de treinamento. Isso é opcional, se o argumento não for dado, padrão para o treinamento de baunilha PLM que máscara em todos os lugares com igual probabilidade (recomendada apenas para pré -treinamento inicial). Para exemplos refinados, consulte training_configs Pasta.
Observe também que a pasta vocabs contém os arquivos de vocabulário para treinamento em pequenas moléculas, proteínas e reações químicas.
Configurações de modelo exemplares (número de cabeças, camadas etc.) podem ser encontradas na pasta Configs.
Para avaliar um modelo treinado por exemplo, na tarefa QED, execute o seguinte:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonModelos pré -tenhados estão disponíveis no Hub do modelo GT4SD. Há um total de 9 modelos que também podem ser usados por meio de espaços Huggingface. Os modelos que fazem parte da publicação também estão disponíveis na pasta da caixa mencionada acima.
Para gerar dados personalizados para a tarefa QED em um formato compatível com RT, execute scripts/generate_example_data.py e aponte para um arquivo .smi com sorrisos na primeira coluna.
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtPara propriedades definidas pelo usuário, adapte o arquivo ou abra um problema.
Se você precisar criar um novo vocabulário para um conjunto de dados, poderá usar scripts/create_vocabulary.py, ele também adicionará automaticamente alguns tokens especiais na parte superior do seu arquivo de vocabulário.
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt Neste ponto, a pasta que contém o arquivo de vocabulário pode ser usada para carregar um tokenizer compatível com qualquer ExpressionBertTokenizer :
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]Se você usar o transformador de regressão, cite:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}