Transformator multitask yang merumuskan kembali regresi sebagai tugas pemodelan urutan bersyarat. Ini menghasilkan model bahasa dikotomis yang secara mulus mengintegrasikan regresi dengan generasi bersyarat yang digerakkan oleh properti.
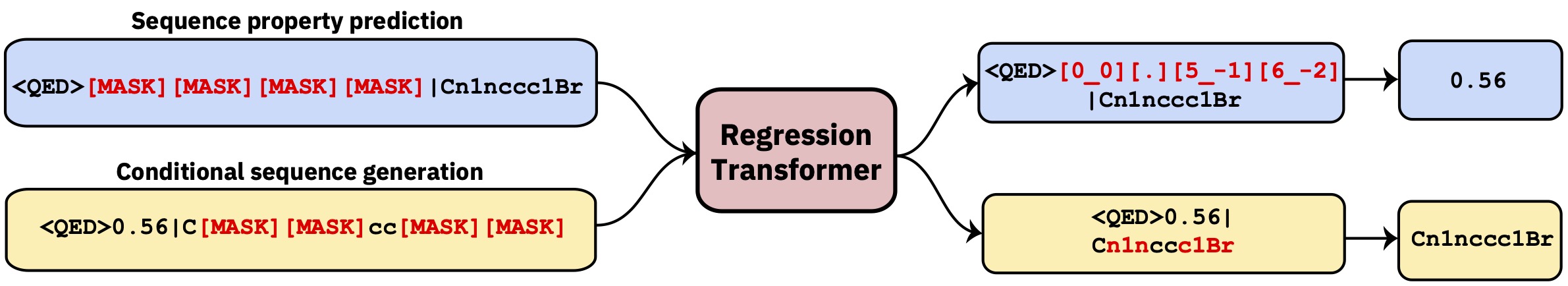
Repo ini berisi kode pengembangan. Bacalah makalah di Nature Machine Intelligence .
? Demo gradio dengan UI sederhana tersedia di ruang pelukan permukaan 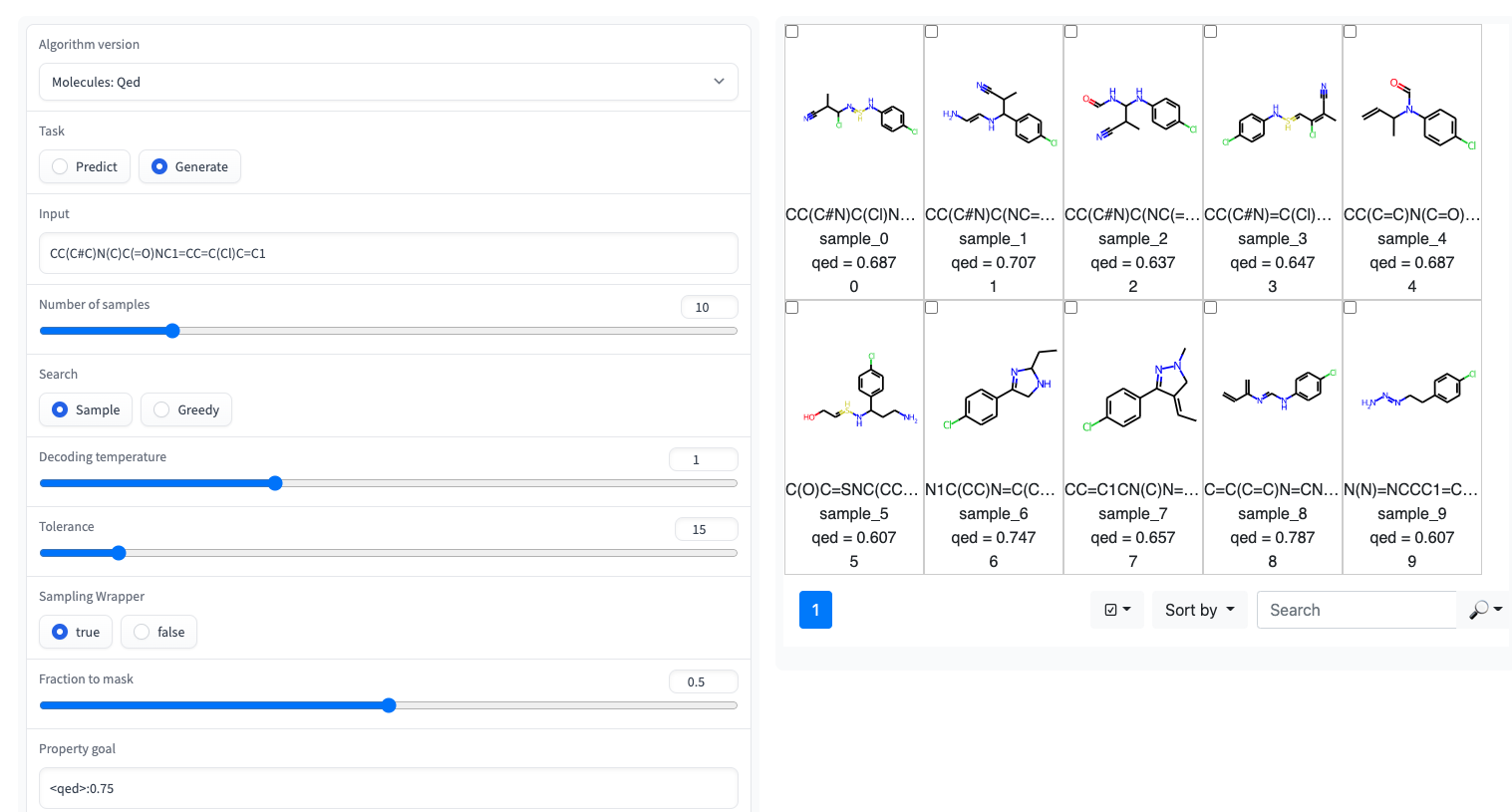
Transformator regresi diimplementasikan di perpustakaan GT4SD. Via GT4SD, menggunakan beberapa regresi pretrainransformers adalah masalah beberapa baris kode. Tutorial lengkap dalam menjalankan inferensi, finetuning model RT (atau melatihnya dari awal) dan berbagi dan menggunakannya ke hub model GT4SD, dapat ditemukan di sini.
Misalnya, melalui GT4SD Anda dapat menggunakan RT pretrain pada molekul kecil dengan beberapa sifat seperti yang ditunjukkan dalam kertas, khususnya QED dan ESOL (kelarutan air). Ada juga beberapa varian multiproperty dari RT: misalnya, model yang dilatih bersama pada logp dan sintesizabilitas (alias scscore). Untuk pemodelan bahasa protein, Anda juga akan menemukan RT yang dilatih pada dataset stabilitas peptida dari tolok ukur pita. Singkatnya, GT4SD memberikan model RT yang diatur sebelumnya:
qed , esol , crippen_logp ) atau multiple ( logp_and_synthesizability , cosmo_acdl , pfas ) properti. Semua model itu menggunakan selfie selain dari crippen_logp yang menggunakan senyum.stabilityuspto (Menggunakan Senyum Reaksi)rop_catalyst dan block_copolymer keduanya dijelaskan dalam Park et al., (2023; Nature Communications ). rop_catalyst menggunakan selfie konvensional tetapi model block_copolymer menggunakan bahasa polimer baru yang disebut CMDL yang dijelaskan juga dalam Park et al., (2023; Nature Communications ).Buku catatan Jupyter dengan mainan Usecase tentang mengadaptasi molekul menuju kelarutan disediakan di GT4SD juga. Jika Anda menggunakan GT4SD, Anda dapat menghasilkan molekul seperti ini:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) Jelajahi kelarutan ruang kimia lokal di sekitar Buturon. Setelah memvariasikan primer properti, Anda mungkin mendapatkan sesuatu seperti ini: 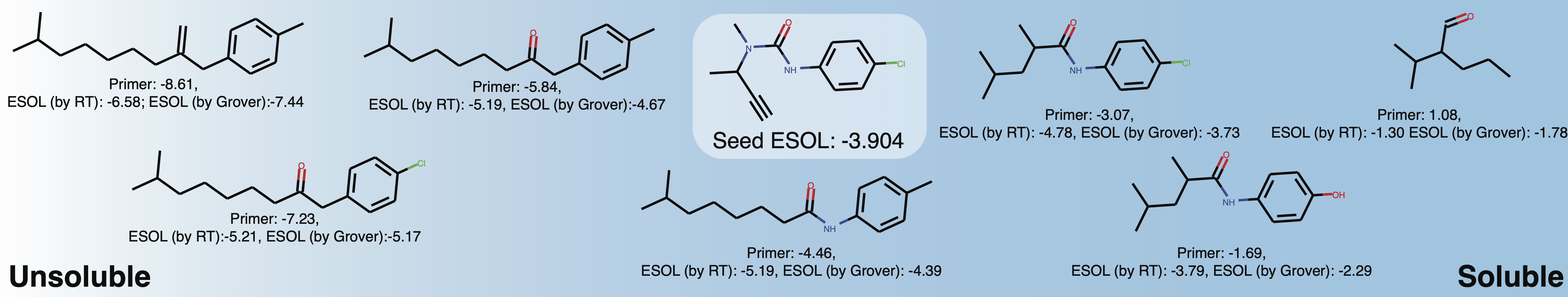
Ini terutama dimaksudkan untuk mereproduksi atau memperluas hasil kertas.
conda env create -f conda.yml
conda activate terminator
pip install -e .Data yang diproses yang digunakan untuk melatih model tersedia melalui kotak.
Anda dapat mengunduh data dan meluncurkan pelatihan dengan menunjuk untuk melatih dan menguji data:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path menunjuk ke file yang menentukan rezim pelatihan. Ini adalah opsional, jika argumen tidak diberikan, kami default untuk pelatihan PLM vanilla yang menutupi di mana -mana dengan probabilitas yang sama (direkomendasikan hanya untuk pretraining awal). Untuk contoh yang halus, silakan lihat folder training_configs .
Perhatikan juga bahwa folder vocabs berisi file kosa kata untuk pelatihan molekul kecil, protein dan reaksi kimia.
Konfigurasi model contoh (jumlah kepala, lapisan, dll.) Dapat ditemukan di folder Configs.
Untuk mengevaluasi model yang dilatih misalnya, pada tugas QED, jalankan yang berikut:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonModel pretrain tersedia melalui hub model GT4SD. Ada total 9 model yang juga dapat digunakan melalui ruang pelukan. Model yang merupakan bagian dari publikasi juga tersedia melalui folder kotak yang disebutkan di atas.
Untuk menghasilkan data khusus untuk tugas QED dalam format yang kompatibel dengan RT, jalankan skrip/generate_example_data.py dan arahkan ke file .smi dengan senyum di kolom pertama.
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtUntuk properti yang ditentukan pengguna, silakan beradaptasi file atau buka masalah.
Jika Anda perlu membuat kosakata baru untuk dataset, Anda dapat menggunakan skrip/create_vocabulary.py itu juga akan secara otomatis menambahkan beberapa token khusus di bagian atas file kosa kata Anda.
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt Pada titik ini folder yang berisi file kosa kata dapat digunakan untuk memuat tokenizer yang kompatibel dengan ExpressionBertTokenizer apa pun:
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]Jika Anda menggunakan transformator regresi, silakan kutip:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}