Ein Multitask -Transformator, der die Regression als bedingte Sequenzmodellierungsaufgabe neu formuliert. Dies liefert ein dichotomes Sprachmodell, das die Regression nahtlos mit der Eigenschaftsbedingung integriert.
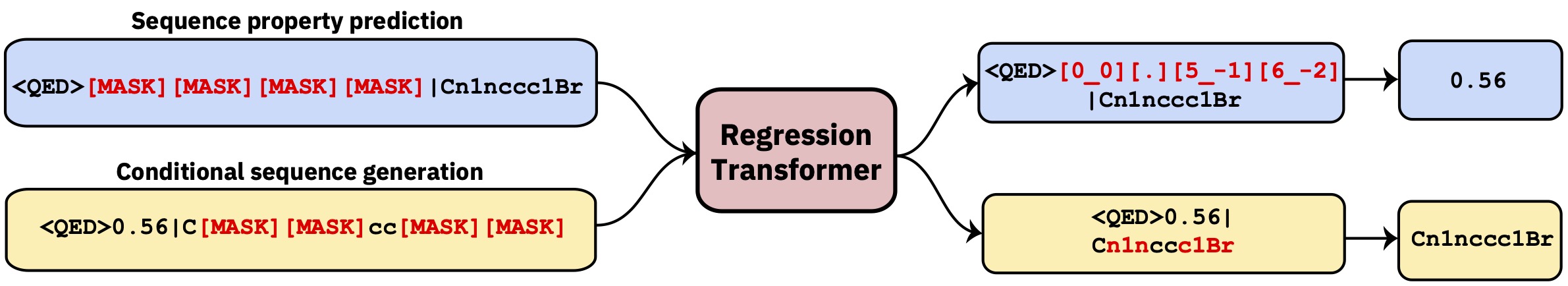
Dieses Repo enthält den Entwicklungscode. Lesen Sie das Papier in Nature Machine Intelligence .
? Eine Gradio -Demo mit einer einfachen Benutzeroberfläche ist auf Umarmungsflächen erhältlich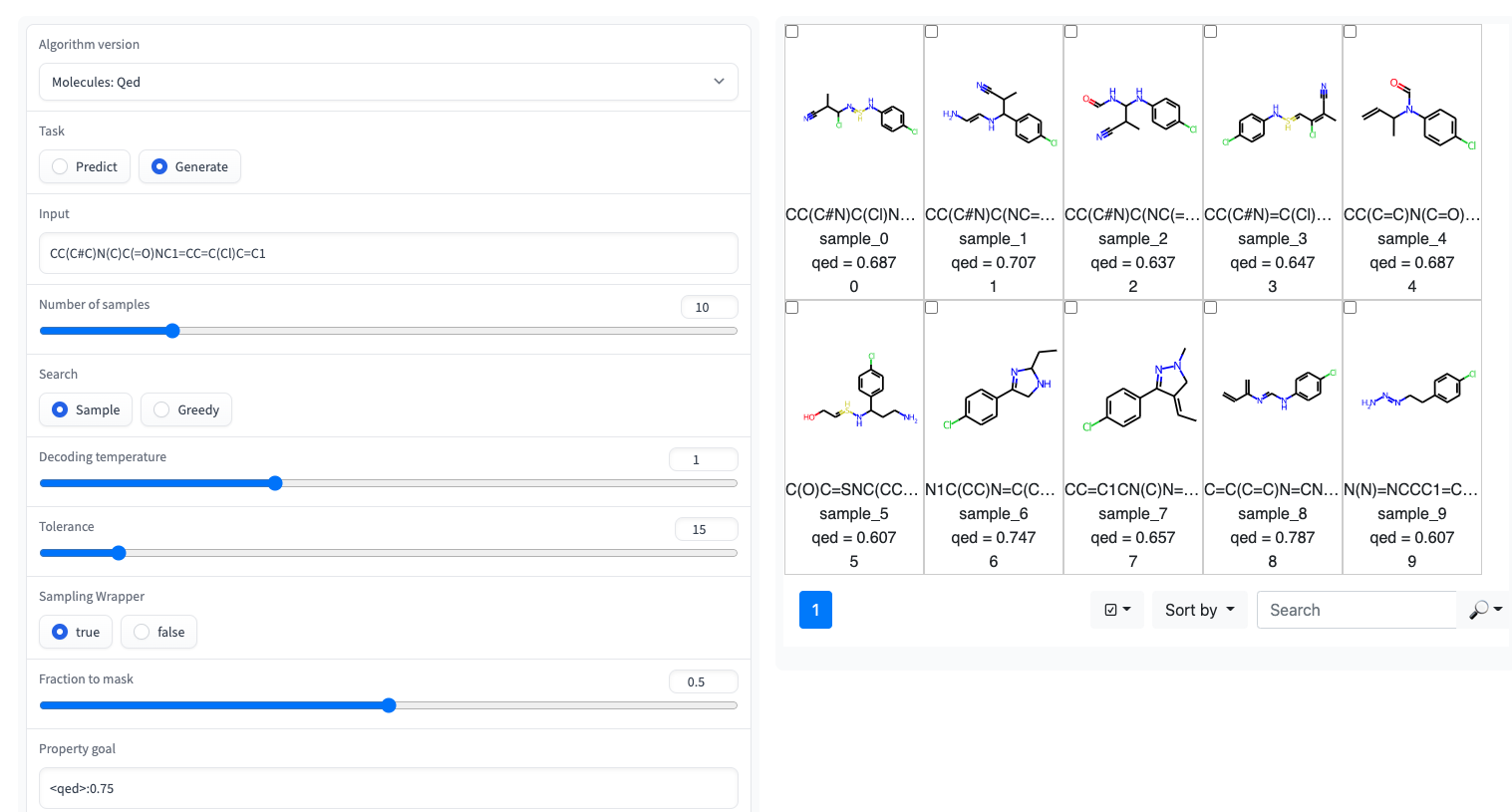
Der Regressionstransformator wird in der GT4SD -Bibliothek implementiert. Über GT4SD ist die Verwendung mehrerer vorbereiteter Regressionstransformatoren eine Frage einiger Codezeilen. Ein vollständiges Tutorial zum Ausführen von Inferenz, dem Finetuning eines RT -Modells (oder dem Training von Grund auf) und dem Teilen und Bereitstellen an den GT4SD -Modellzentrum finden Sie hier.
Zum Beispiel können Sie über GT4SD die RT -RT verwenden, die auf kleinen Molekülen mit einigen Eigenschaften vorgelegt sind, wie in dem Papier gezeigt, insbesondere QED und ESOL (Wasserlöslichkeit). Es gibt auch mehrere Multiproperty -Varianten des RT: z. Für die Proteinsprachmodellierung finden Sie auch einen RT, der auf einem Peptid -Stabilitätsdatensatz aus dem Band -Benchmark trainiert wird. Insgesamt bietet GT4SD RT -Modelle, die vorab vorgelegt sind:
qed , esol , crippen_logp ) oder Multiple ( logp_and_synthesizability , cosmo_acdl , pfas ) Eigenschaften. Alle diese Modelle verwenden Selfies, abgesehen von crippen_logp , das ein Lächeln verwendet.stabilityuspto (unter Verwendung von Reaktionslächeln)rop_catalyst und block_copolymer werden beide in Park et al. (2023; Nature Communications ) beschrieben. Der rop_catalyst verwendet herkömmliche Selfies, aber das block_copolymer -Modell verwendet eine neuartige Polymersprache namens CMDL, die auch in Park et al. (2023; Nature Communications ) beschrieben wird.Ein Jupyter -Notizbuch mit einem Spielzeug, das ein Molekül an die Löslichkeit anpasst, wird auch in GT4SD bereitgestellt. Wenn Sie GT4SD verwenden, können Sie solche Moleküle erzeugen:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) Erforschen Sie die Löslichkeit des lokalen chemischen Raums um Buturon. Wenn Sie die Immobilienprimer variieren, können Sie so etwas erhalten: 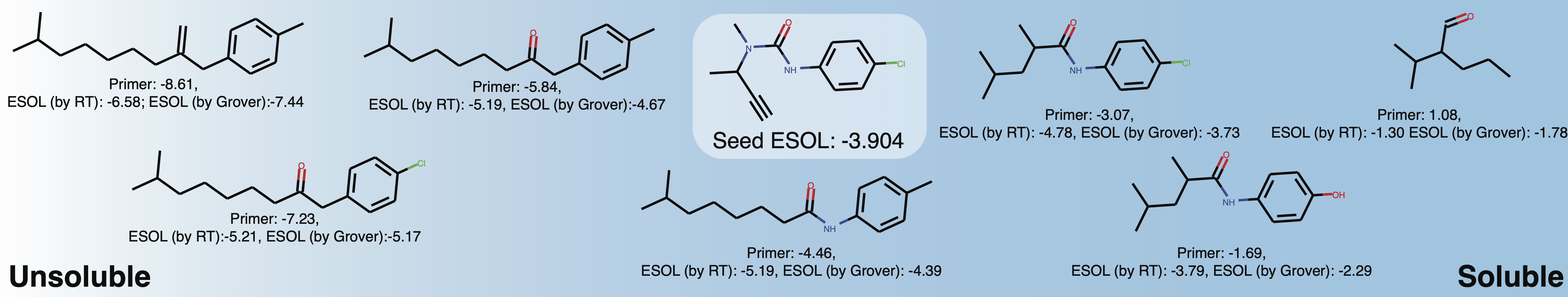
Dies soll hauptsächlich die Ergebnisse des Papiers reproduzieren oder erweitern.
conda env create -f conda.yml
conda activate terminator
pip install -e .Die zum Training der Modelle verwendeten verarbeiteten Daten sind über Box verfügbar.
Sie können die Daten herunterladen und ein Training starten, indem Sie darauf hinweisen, um Daten zu trainieren und zu testen:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path verweist auf eine Datei, die das Trainingsregime angibt. Dies ist optional. Wenn das Argument nicht angegeben ist, sind wir standardmäßig ein Vanilla -PLM -Training, das überall mit gleicher Wahrscheinlichkeit maskiert (nur für die anfängliche Vorabbildung empfohlen). Für raffinierte Beispiele finden Sie unter dem Ordner training_configs .
Beachten Sie außerdem, dass der Ordner vocabs die Vokabulardateien zum Training auf kleinen Molekülen, Proteinen und chemischen Reaktionen enthält.
Vorbildliche Modellkonfigurationen (Anzahl der Köpfe, Ebenen usw.) finden Sie im Ordner configs.
Um ein Modell ausgebildet zu bewerten, z. B. bei der QED -Aufgabe, führen Sie Folgendes aus:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonVorbereitete Modelle sind über den GT4SD -Modell Hub erhältlich. Es gibt insgesamt 9 Modelle, die auch über Harming -Face -Räume verwendet werden können. Modelle, die Teil der Veröffentlichung sind, sind auch über den oben genannten Boxordner erhältlich.
Um benutzerdefinierte Daten für die QED-Aufgabe in einem RT-kompatiblen Format zu generieren, führen Sie Skripte aus/generate_example_data.py und verweisen Sie auf eine .smi Datei mit Lächeln in der ersten Spalte.
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtFür benutzerdefinierte Eigenschaften passen Sie bitte die Datei an oder öffnen Sie ein Problem.
Wenn Sie ein neues Vokabular für einen Datensatz erstellen müssen, können Sie Skripts/create_vocabulary.py verwenden. Außerdem fügen Sie auch einige spezielle Token oben in Ihrer Vokabulardatei hinzu.
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt Zu diesem Zeitpunkt kann der Ordner, der die Vokabulardatei enthält, verwendet werden, um einen mit jedem ExpressionBertTokenizer kompatibelen Tokenizer zu laden:
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]Wenn Sie den Regressionstransformator verwenden, zitieren Sie bitte:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}