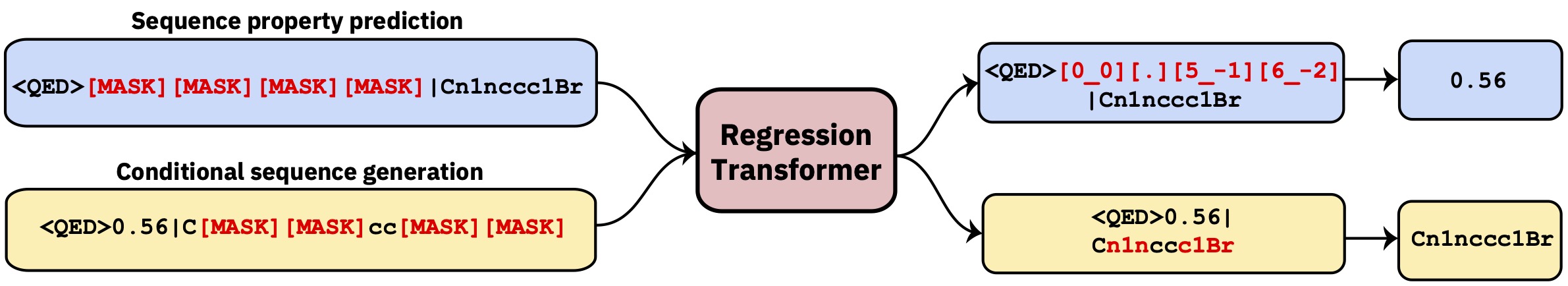
محول متعدد المهام يعيد إعادة الانحدار كمهمة نمذجة تسلسل مشروطة. هذا يعطي نموذج لغة ثنائية التفرع يدمج بسلاسة الانحدار مع توليد مشروط القائم على الممتلكات.
هذا الريبو يحتوي على رمز التطوير. اقرأ الورقة في ذكاء آلة الطبيعة .
؟ يتوفر عرض Gradio مع واجهة مستخدم بسيطة على مساحات Huggingface 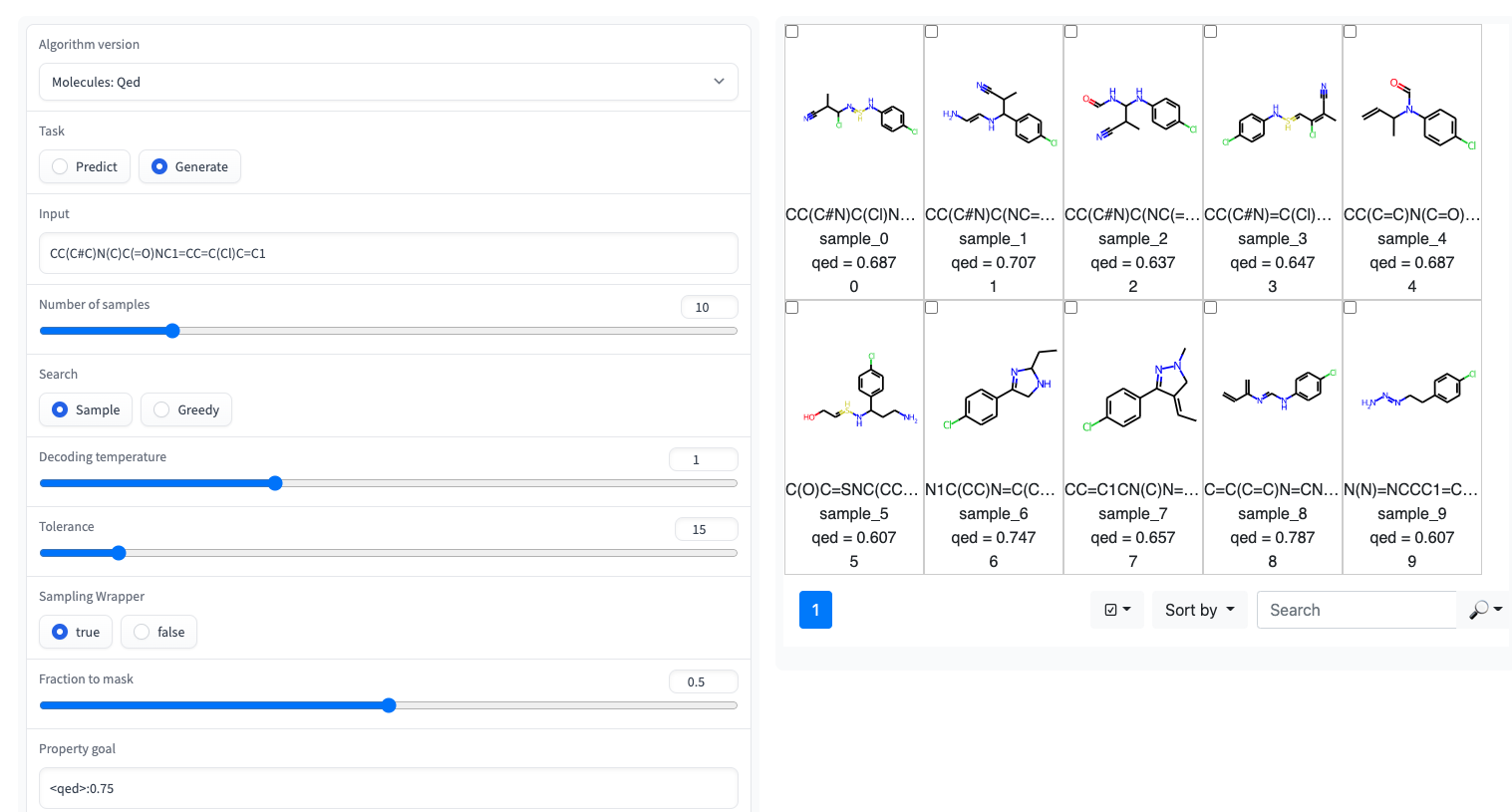
يتم تنفيذ محول الانحدار في مكتبة GT4SD. عبر GT4SD ، فإن استخدام العديد من الانحدارات المسبق هو مسألة بضعة أسطر من التعليمات البرمجية. يمكن العثور هنا على البرنامج التعليمي الكامل للاستدلال على التشغيل ، أو استغلال نموذج RT (أو تدريبه من نقطة الصفر) ومشاركته ونشره على محور طراز GT4SD.
على سبيل المثال ، عبر GT4SD ، يمكنك استخدام RT PretRained على جزيئات صغيرة مع بعض الخصائص كما هو موضح في الورقة ، وخاصة QED و ESOL (قابلية ذوبان الماء). هناك أيضًا العديد من المتغيرات المتعددة في RT: على سبيل المثال ، نموذج مدرب بشكل مشترك على logp وتوليف (ويعرف أيضًا باسم SCSCORE). لنمذجة لغة البروتين ، ستجد أيضًا RT مدربة على مجموعة بيانات استقرار الببتيد من معيار الشريط. باختصار ، يوفر GT4SD نماذج RT التي تم تجهيزها على:
qed ، esol ، crippen_logp ) أو خصائص متعددة ( logp_and_synthesizability ، cosmo_acdl ، pfas ). تستخدم جميع هذه النماذج صور شخصية بصرف النظر عن crippen_logp التي تستخدم الابتسامات.stabilityuspto (باستخدام ابتسامات التفاعل)rop_catalyst و block_copolymer في Park et al. ، (2023 ؛ Nature Communications ). يستخدم rop_catalyst صور شخصية تقليدية ولكن نموذج block_copolymer يستخدم لغة بوليمر جديدة تسمى CMDL الموصوفة أيضًا في Park et al. ، (2023 ؛ Nature Communications ).يتم توفير دفتر Jupyter مع لعبة usecase حول تكييف جزيء نحو الذوبان في GT4SD أيضا. إذا كنت تستخدم GT4SD ، فيمكنك إنشاء جزيئات مثل هذا:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) استكشاف قابلية ذوبان المساحة الكيميائية المحلية حول بوتورون. عند تغيير الاشعال في الممتلكات ، قد تحصل على شيء مثل هذا: 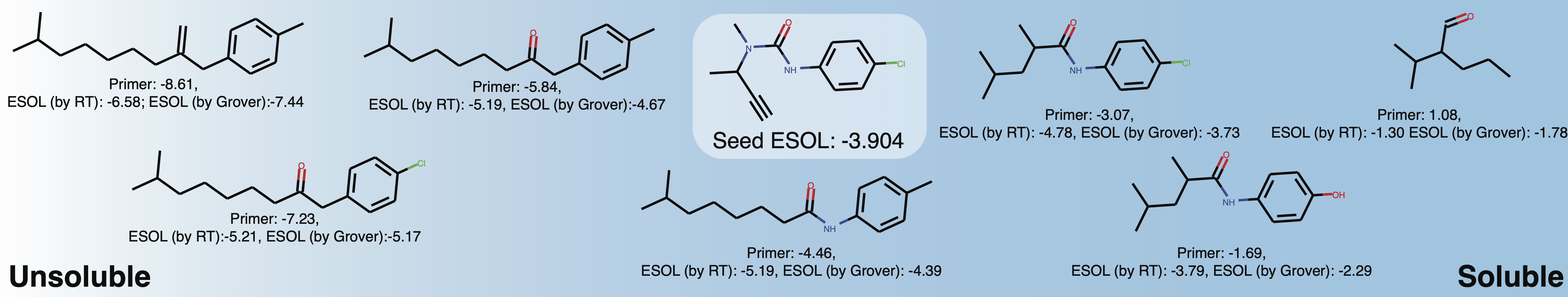
هذا يهدف بشكل أساسي إلى إعادة إنتاج أو تمديد نتائج الورقة.
conda env create -f conda.yml
conda activate terminator
pip install -e .تتوفر البيانات المعالجة المستخدمة لتدريب النماذج عبر المربع.
يمكنك تنزيل البيانات وإطلاق تدريب عن طريق الإشارة إلى تدريب البيانات واختبارها:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path إلى ملف يحدد نظام التدريب. هذا اختياري ، إذا لم يتم إعطاء الوسيطة ، فإننا نتخلف عن تدريب الفانيليا PLM الذي يخفف في كل مكان باحتمال متساو (موصى به لتدريب الأولي فقط). للحصول على الأمثلة المكررة ، يرجى الاطلاع على مجلد training_configs .
لاحظ أيضًا أن مجلد vocabs يحتوي على ملفات المفردات للتدريب على الجزيئات الصغيرة والبروتينات والتفاعلات الكيميائية.
يمكن العثور على تكوينات النموذج النموذجية (عدد الرؤوس ، الطبقات ، إلخ) في مجلد التكوينات.
لتقييم نموذج مدرب على سبيل المثال ، في مهمة QED ، قم بتشغيل ما يلي:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonتتوفر النماذج المسبقة عبر مركز GT4SD Model. هناك ما مجموعه 9 نماذج يمكن استخدامها أيضًا عبر مساحات Huggingface. تتوفر النماذج التي تشكل جزءًا من المنشور أيضًا عبر مجلد Box المذكور أعلاه.
لإنشاء بيانات مخصصة لمهمة QED بتنسيق متوافق مع RT ، قم بتشغيل البرامج النصية/cenderate_example_data.py وأشار إلى ملف .smi مع الابتسامات في العمود الأول.
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtبالنسبة للخصائص المعرفة من قبل المستخدم ، يرجى تكييف الملف أو فتح مشكلة.
إذا كنت بحاجة إلى إنشاء مفردات جديدة لمجموعة بيانات ، فيمكنك استخدام البرامج النصية/create_vocabulary.py ، فسيتم إضافة بعض الرموز الخاصة تلقائيًا في الجزء العلوي من ملف المفردات.
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt في هذه المرحلة ، يمكن استخدام المجلد الذي يحتوي على ملف المفردات لتحميل Tokenizer متوافق مع أي ExpressionBertTokenizer :
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]إذا كنت تستخدم محول الانحدار ، يرجى الاستشهاد:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}