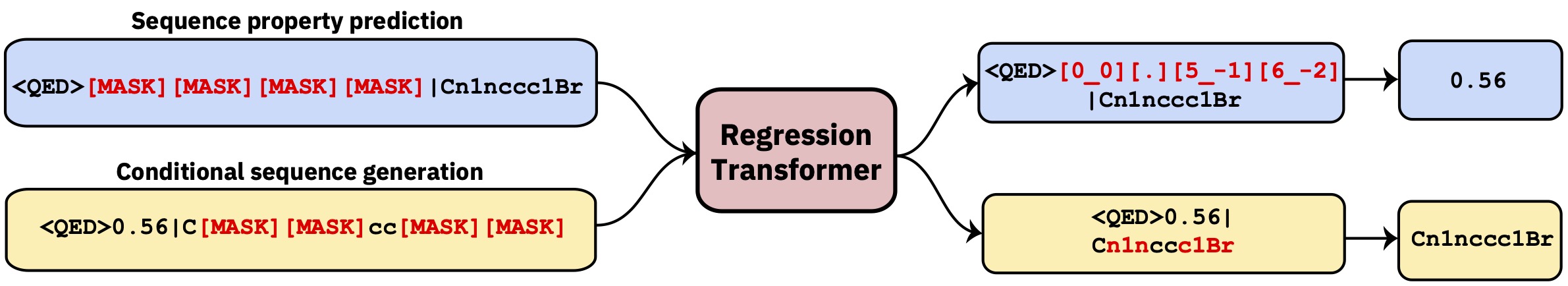
หม้อแปลงมัลติทาสก์ที่ปรับปรุงการถดถอยเป็นงานสร้างแบบจำลองลำดับตามเงื่อนไข สิ่งนี้ให้รูปแบบภาษาแบบแบ่งขั้วซึ่งรวมการถดถอยเข้ากับการสร้างเงื่อนไขที่ขับเคลื่อนด้วยทรัพย์สินได้อย่างราบรื่น
repo นี้มีรหัสการพัฒนา อ่านกระดาษใน Intelligence Machine Machine
- การสาธิต gradio พร้อม UI แบบง่ายมีอยู่ในพื้นที่ HuggingFace 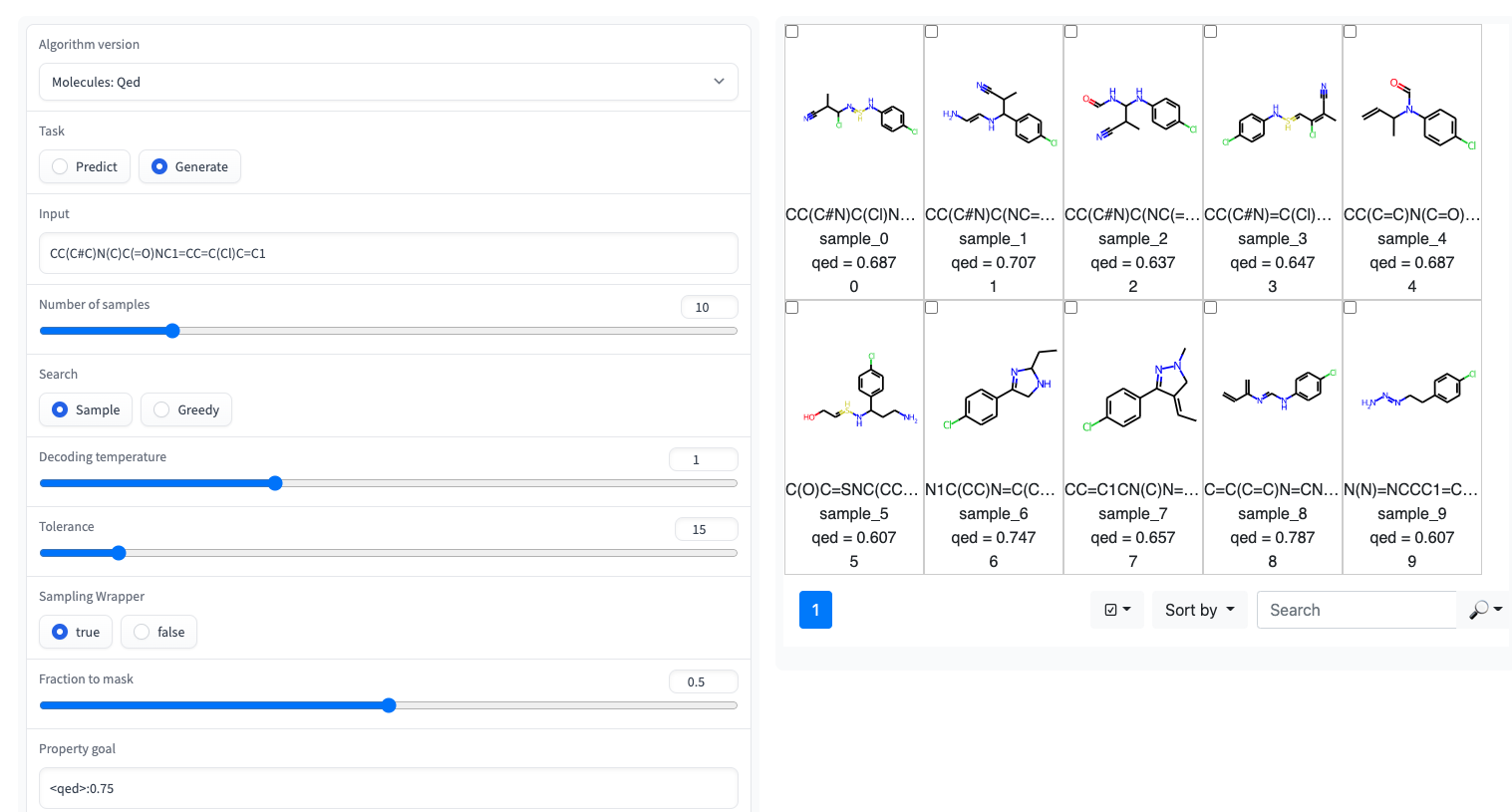
หม้อแปลงการถดถอยถูกนำไปใช้ในไลบรารี GT4SD ผ่าน GT4SD โดยใช้การถดถอยแบบ pretrened หลายครั้งเป็นเรื่องของรหัสสองสามบรรทัด การสอนที่สมบูรณ์ของการอนุมานการใช้งาน, การปรับรูปแบบ RT (หรือฝึกอบรมตั้งแต่เริ่มต้น) และแบ่งปันและปรับใช้กับศูนย์กลางรุ่น GT4SD สามารถพบได้ที่นี่
ตัวอย่างเช่นผ่าน GT4SD คุณสามารถใช้ RT pretrained บนโมเลกุลขนาดเล็กที่มีคุณสมบัติบางอย่างดังแสดงในกระดาษโดยเฉพาะอย่างยิ่ง QED และ ESOL (ความสามารถในการละลายน้ำ) นอกจากนี้ยังมีหลายตัวแปรหลายตัวแปรของ RT: เช่นแบบจำลองที่ผ่านการฝึกอบรมร่วมกันใน LOGP และการสังเคราะห์ (aka SCSCORE) สำหรับการสร้างแบบจำลองภาษาโปรตีนคุณจะพบกับ RT ที่ได้รับการฝึกฝนในชุดข้อมูลความเสถียรของเปปไทด์จากเกณฑ์มาตรฐานเทป โดยรวมแล้ว GT4SD ให้แบบจำลอง RT ที่ได้รับการปรับแต่งบน:
qed , esol , crippen_logp ) หรือหลาย ( logp_and_synthesizability , cosmo_acdl , pfas ) คุณสมบัติ ทุกรุ่นเหล่านั้นใช้เซลฟี่นอกเหนือจาก crippen_logp ซึ่งใช้รอยยิ้มstabilityuspto (ใช้รอยยิ้มปฏิกิริยา)rop_catalyst และ block_copolymer อธิบายไว้ใน Park et al., (2023; การสื่อสารธรรมชาติ ) rop_catalyst ใช้เซลฟี่ทั่วไป แต่โมเดล block_copolymer ใช้ภาษาพอลิเมอร์ใหม่ที่เรียกว่า CMDL ที่อธิบายไว้ใน Park et al., (2023; การสื่อสารธรรมชาติ )สมุดบันทึก Jupyter ที่มีของเล่น usecase ในการปรับโมเลกุลที่มีต่อความสามารถในการละลายใน GT4SD ด้วย หากคุณใช้ GT4SD คุณสามารถสร้างโมเลกุลเช่นนี้:
from gt4sd . algorithms . conditional_generation . regression_transformer import (
RegressionTransformer , RegressionTransformerMolecules
)
buturon = "CC(C#C)N(C)C(=O)NC1=CC=C(Cl)C=C1"
target_esol = - 3.53
config = RegressionTransformerMolecules (
algorithm_version = "solubility" ,
search = "sample" ,
temperature = 2 ,
tolerance = 5 ,
sampling_wrapper = {
'property_goal' : { '<esol>' : target_esol },
'fraction_to_mask' : 0.2
}
)
esol_generator = RegressionTransformer ( configuration = config , target = buturon )
generations = list ( esol_generator . sample ( 8 )) สำรวจความสามารถในการละลายของพื้นที่เคมีในท้องถิ่นรอบ Buturon เมื่อเปลี่ยนไพรเมอร์คุณสมบัติคุณอาจได้รับสิ่งนี้: 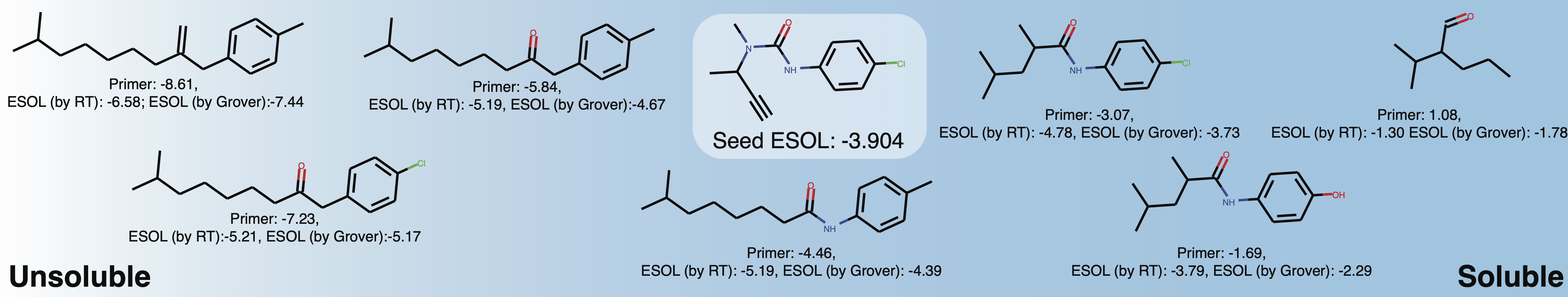
นี่คือจุดประสงค์หลักที่จะทำซ้ำหรือขยายผลลัพธ์ของกระดาษ
conda env create -f conda.yml
conda activate terminator
pip install -e .ข้อมูลที่ประมวลผลที่ใช้ในการฝึกอบรมโมเดลนั้นมีให้ผ่านกล่อง
คุณสามารถดาวน์โหลดข้อมูลและเปิดการฝึกอบรมได้โดยชี้ไปที่การฝึกอบรมและทดสอบข้อมูล:
python scripts/run_language_modeling.py --output_dir rt_example
--config_name configs/rt_small.json --tokenizer_name ./vocabs/smallmolecules.txt
--do_train --do_eval --learning_rate 1e-4 --num_train_epochs 5 --save_total_limit 2
--save_steps 500 --per_gpu_train_batch_size 16 --evaluate_during_training --eval_steps 5
--eval_data_file ./examples/qed_property_example.txt --train_data_file ./examples/qed_property_example.txt
--line_by_line --block_size 510 --seed 42 --logging_steps 100 --eval_accumulation_steps 2
--training_config_path training_configs/qed_alternated_cc.jsontraining_config_path ชี้ไปที่ไฟล์ที่ระบุระบอบการฝึกอบรม นี่เป็นทางเลือกหากไม่ได้รับการโต้แย้งเราจะเริ่มต้นการฝึกอบรม Vanilla PLM ที่มาสก์ทุกที่ด้วยความน่าจะเป็นที่เท่าเทียมกัน (แนะนำสำหรับการเตรียมการครั้งแรกเท่านั้น) สำหรับตัวอย่างที่ได้รับการกลั่นโปรดดูโฟลเดอร์ training_configs
โปรดทราบว่าโฟลเดอร์ vocabs มีไฟล์คำศัพท์สำหรับการฝึกอบรมเกี่ยวกับโมเลกุลขนาดเล็กโปรตีนและปฏิกิริยาทางเคมี
การกำหนดค่าโมเดลที่เป็นแบบอย่าง (จำนวนหัว, เลเยอร์ ฯลฯ ) สามารถพบได้ในโฟลเดอร์ configs
ในการประเมินแบบจำลองที่ผ่านการฝึกอบรมเช่นในงาน QED ให้เรียกใช้สิ่งต่อไปนี้:
python scripts/eval_language_modeling.py --output_dir path_to_model
--eval_file ./examples/qed_property_example.txt --eval_accumulation_steps 2 --param_path configs/qed_eval.jsonรุ่นที่ผ่านการฝึกอบรมมีให้บริการผ่านศูนย์กลางรุ่น GT4SD มีทั้งหมด 9 รุ่นที่สามารถใช้งานผ่านช่องว่าง HuggingFace รุ่นที่เป็นส่วนหนึ่งของสิ่งพิมพ์ยังมีให้ผ่านโฟลเดอร์กล่องที่กล่าวถึงข้างต้น
ในการสร้างข้อมูลที่กำหนดเองสำหรับงาน QED ในรูปแบบที่เข้ากันได้ RT ให้เรียกใช้สคริปต์/generate_example_data.py และชี้ไปที่ไฟล์ .smi พร้อมรอยยิ้มในคอลัมน์แรก
python scripts/generate_example_data.py examples/example.smi examples/qed_property_example.txtสำหรับคุณสมบัติที่ผู้ใช้กำหนดโปรดปรับไฟล์หรือเปิดปัญหา
หากคุณต้องการสร้างคำศัพท์ใหม่สำหรับชุดข้อมูลคุณสามารถใช้สคริปต์/create_vocabulary.py มันจะเพิ่มโทเค็นพิเศษบางอย่างโดยอัตโนมัติที่ด้านบนของไฟล์คำศัพท์ของคุณโดยอัตโนมัติ
python scripts/create_vocabulary.py examples/qed_property_example.txt examples/vocab.txt ณ จุดนี้โฟลเดอร์ที่มีไฟล์คำศัพท์สามารถใช้ในการโหลด tokenizer ที่เข้ากันได้กับ ExpressionBertTokenizer :
> >> from terminator . tokenization import ExpressionBertTokenizer
> >> tokenizer = ExpressionBertTokenizer . from_pretrained ( 'examples' )
> >> text = '<qed>0.3936|CBr'
> >> tokens = tokenizer . tokenize ( text )
> >> print ( tokens )
[ '<qed>' , '_0_0_' , '_._' , '_3_-1_' , '_9_-2_' , '_3_-3_' , '_6_-4_' , '|' , 'C' , 'Br' ]
> >> token_indexes = tokenizer . convert_tokens_to_ids ( tokenizer . tokenize ( text ))
> >> print ( token_indexes )
[ 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 ]
> >> tokenizer . build_inputs_with_special_tokens ( token_indexes )
[ 12 , 16 , 17 , 18 , 28 , 45 , 34 , 35 , 19 , 15 , 63 , 13 ]หากคุณใช้หม้อแปลงการถดถอยโปรดอ้างอิง:
@article { born2023regression ,
title = { Regression Transformer enables concurrent sequence regression and generation for molecular language modelling } ,
author = { Born, Jannis and Manica, Matteo } ,
journal = { Nature Machine Intelligence } ,
volume = { 5 } ,
number = { 4 } ,
pages = { 432--444 } ,
year = { 2023 } ,
publisher = { Nature Publishing Group UK London }
}